科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-04-30
来源:BioArt
在基于靶点的药物研发过程中,针对疾病相关的靶点筛选苗头化合物或者先导化合物是必不可少的一环。由于化合物的高通量实验通常需要大量资源和时间投入,相对低成本而快速的虚拟筛选也已成为药物研发过程中的重要辅助工具。除了分子对接、分子模拟等计算机辅助药物设计方法以外,预测蛋白-小分子相互作用的机器学习和深度学习方法也成为了近年来的研究热点。
近日,清华大学交叉信息研究院曾坚阳团队发表在Cell Systems的一篇题为MONN: a multi-objective neural network for predicting compound-protein interactions and affinities的报道,介绍了一个新的深度学习模型,从亲和力和局部非共价键的角度对靶点蛋白和小分子化合物之间的相互作用进行预测。

亲和力是对化合物进行筛选的直接指标,而蛋白质和小分子化合物之间的局部相互作用则可以为化合物设计提供参考。亲和力数据相对容易获得,数据量相对较多,现有的深度学习模型主要将亲和力预测作为目标。由于深度学习的黑箱特性,这些方法对分子间相互作用机制的解析比较困难,仅有少数方法通过神经网络的注意力机制对分子间局部相互作用位点的捕获进行了尝试。
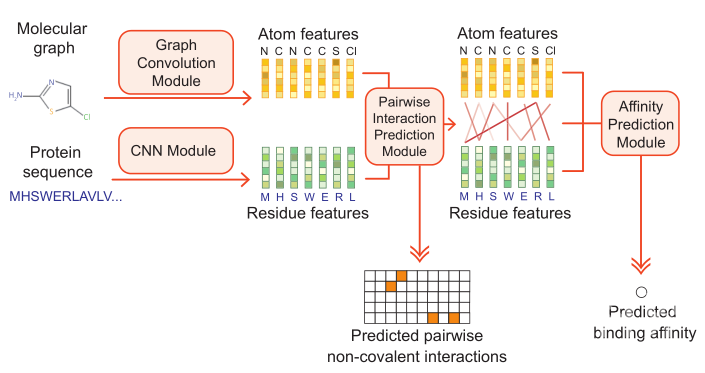
图2:MONN模型结构
曾坚阳研究组对预测蛋白-小分子相互作用的机器学习问题进行了重新定义,将蛋白质序列和小分子化学结构作为模型输入,亲和力和分子间局部非共价键同时作为预测目标。在提出的MONN模型结构中,图神经网络和卷积神经网络分别被用来提取小分子和蛋白质的特征,随后一个局部相互作用预测模块对小分子的每个原子和蛋白质的每个残基之间的所有可能组合进行非共价键形成概率的预测。最后,这一预测信息和小分子、蛋白质的特征则再次被下游模块利用来预测亲和力的数值(图2)。
为了大规模评估分子间非共价键的预测效果,本研究从PDBbind数据库中一万多条蛋白-小分子复合物的结构中提取了分子间的非共价键形成位点。对基于注意力的模型进行的评估表示,目前神经网络的注意力机制较难自动捕获非共价键的形成位点,因此,在MONN模型中,这一信息被用作监督信息从而对神经网络进行更有效的训练。交叉验证表明,MONN不仅能成功预测蛋白-小分子间的非共价键,还可以在亲和力预测任务上获得比现有方法更好的效果。
本研究提出的MONN模型有效整合了局部和整体的相互作用信息,在虚拟化合物筛选中具有潜在的应用价值。除此之外,曾坚阳研究组还开发了一系列基于机器学习和深度学习的药物预测方法,包括药物重定位算法DTINet【1】和NeoDTI【2】,基于表示学习的虚拟筛选算法DeepCPI【3】,药物-靶点关系自动抽取的文本挖掘模型BERE【4】等。最近,为应对新冠疫情,研究组还利用其药物重定位平台筛选出了具有体外抑制SARS-CoV-2活性的药物 【5】。
原文链接:
https://www.sciencedirect.com/science/article/pii/S2405471220300818
参考文献
2. Wan, F., Hong, L., Xiao, A., Jiang, T., & Zeng, J. (2019). NeoDTI: neural integration of neighbor information from a heterogeneous network for discovering new drug–target interactions. Bioinformatics, 35(1), 104-111.
3. Wan, F., Zhu, Y., Hu, H., Dai, A., Cai, X., Chen, L., ... & Zeng, J. (2019). DeepCPI: A Deep Learning-based Framework for Large-scale in silico Drug Screening. Genomics, Proteomics & Bioinformatics, 17(5), 478-495.
4. Hong, L., Lin, J., Tao, J., & Zeng, J. (2019). BERE: An accurate distantly supervised biomedical entity relation extraction network. arXiv preprint arXiv:1906.06916.
5. Ge, Y., Tian, T., Huang, S., Wan, F., Li, J., Li, S., ... & Cheng, L. (2020). A data-driven drug repositioning framework discovered a potential therapeutic agent targeting COVID-19. bioRxiv.
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652483875&idx=7&sn=07a893f67f46082366cb071f43742a93&chksm=84e22297b395ab8157aa8c149e1b63a6d64c68f5e17fad855958bef58eeeb3af5109c2458df7#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

深度学习神经网络方法获改进
谷歌开发新式AI算法,可绘制大脑神经系统图像
长短期记忆人工神经网络
细胞式类神经网络
递归神经网络

认知神经学家借助人工智能研究人脑

如何入手卷积神经网络
神经网络探索物理问题
神经网络正则变换
Hopfield神经网络