科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-04-30
来源:iNature
原标题:开年第一篇,CRISPR领域先驱David Liu开发新方法,评估CRISPR技术脱靶
位点特异性重组酶(SSR)具有作为理想基因组编辑因子的潜力,因为它们直接催化切割,链交换和DNA片段在确定的重组靶上的重新连接,而不依赖于双链断裂的内源性修复,这可以诱导插入缺失,易位,其他DNA重排或p53激活。 SSR催化的反应可导致靶DNA片段的直接替换,插入或缺失,其效率超过同源定向修复的效率。 SSR在多种细胞状态中具有活性,包括非分裂细胞,并且许多细胞在哺乳动物基因组上有效运作。但是,改变其DNA特异性以操纵任意目的序列仍然是它们广泛用于基因组编辑的障碍。
2019年4月26日,麻省理工大学David Liu团队在Nature Communications上发表题为“High-resolution specificity profiling and off-target prediction forsite-specific DNA recombinases”的文章,开发了一种快速绘制SSR特异性决定因素的方法。这种方法也可用于预测SSR的细胞脱靶活性,这是评估SSR作为潜在工具或治疗剂时的重要考虑因素。研究结果表明,Rec-seq可以为SSR的应用及其进一步发展提供信息。

为了表征相互作用,研究人员构建了14个Cre突变体,其中已知残基的Ala取代与loxP接触,纯化每个变体,并进行Rec-seq 绘制特定残基与Cre的DNA序列偏好之间的功能关系。Cre突变体和野生型Cre的Rec-seq谱的比较产生了对每个残基对整个loxP位点的DNA特异性的贡献的新见解。
结构和诱变研究表明Arg259的突变会影响半位点10位的特异性。实际上,Arg259→Ala变异显示10位富集下降(从野生型Cre的5.0倍增加到1.1倍)对于突变体而言,对左半位点的C或T以及右半位点的G或A的适度偏好。 Arg259→Ala突变体在loxP位点的几乎所有其他位置也显示出增加的偏好,在5-7和16位具有特别高的偏好。这一观察与推测的ZFNs,TALE和Cas9一致,其中Arg259中Ala取代的结合能的丧失需要更高的其他蛋白质保真度:DNA接触以保持足够的结合以支持重组。
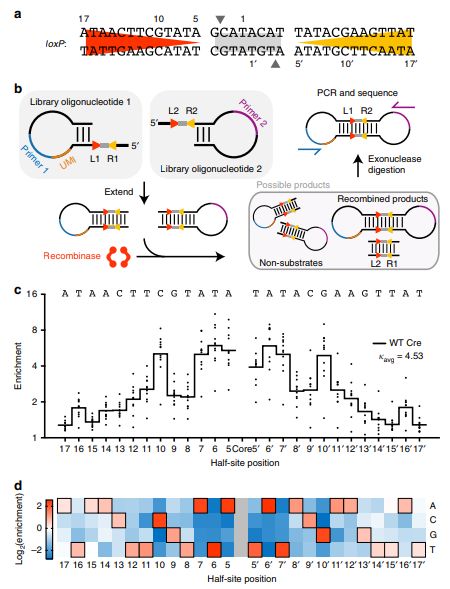
野生型Cre重组酶特异性谱分析
Rec-seq还有助于阐明loxP位置5-7的特异性决定因素候选相互作用残基分布在Cre的三个区域:螺旋B,螺旋D和环之间。螺旋J和K。在潜在的相互作用残基处的Ala突变体的Rec-seq谱显示了相邻残基的不同影响。结果表明Cre在5-7位的明显偏好是由多个弱或间接相互作用引起的,而不是由靠近这些位置的残基强烈决定。
此外,Rec-seq确定了先前未知参与指定位置5-7的二级残留物的贡献。 Gln94的Ala取代导致6和7位的特异性降低,但其他地方的补偿性增加。 Gln90和Gln94两者的双Ala取代与Gln90→Ala单突变体类似地进行,表明DNA接触残基Gln90在确定两个残基中的DNA特异性中起主导作用。Rec-seq概要分析一起阐明了在5-7位共同定义Cre识别的许多交互,并突出了次要和间接交互的重要作用。
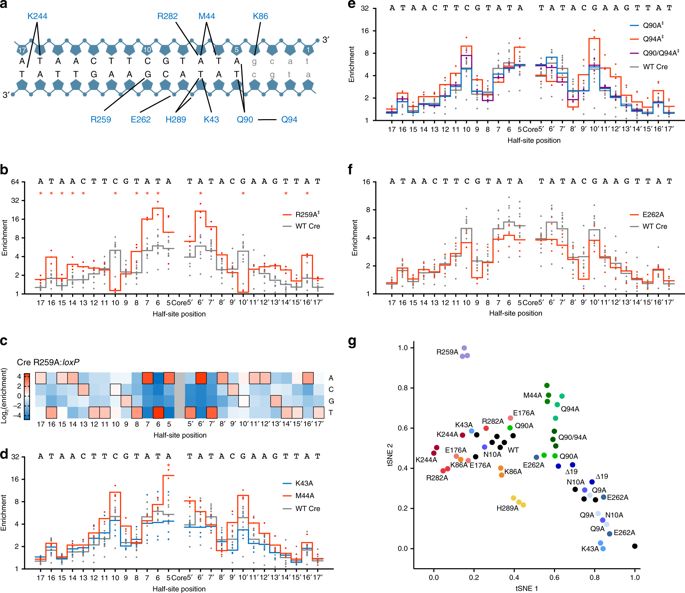
Cre:loxP特异性的决定因素由Rec-seq鉴定为野生型Cre和Ala取代的Cre变体
DNA相互作用主要来自静态晶体结构。虽然结构提供了基于邻近度的可能相互作用的列表,但Rec-seq生成了有助于特异性的残基功能图。研究人员使用t-SNE算法来使用多维相似性分析来关联各个Rec-seq实验的结果。 t-SNE可视化中的实验接近度与它们在整个Rec-seq谱中的相似性有关。例如,含有Met44和Gln94的簇代表在5-7位有助于特异性的功能相似的残基,而接近相同碱基的其他残基(Lys43,Lys86,Arg282)分别出现,与它们的不同作用一致。将野生型Cre簇的Rec-seq实验复制到图的中间;增加序列偏好的Ala取代的突变体出现在野生型分组的左侧,而偏好减少的变体出现在右侧。通过揭示和关联残留物在单核苷酸分辨率下确定整个底物位点的DNA识别中的个体作用,Rec-seq极大地增强了对SSR:DNA相互作用的理解。
作为基因组编辑剂的位点特异性重组酶(SSR)的开发受到改变其天然DNA特异性的困难的限制。该文章描述了Rec-seq,一种以全面和无偏见的方式揭示SSR的DNA特异性决定簇和潜在的脱靶底物的方法。研究团队应用Rec-seq来表征几种天然和进化的SSR的DNA特异性决定因子,包括Cre,Cre的进化变体和其他SSR家族成员。这些酶及其突变体的Rec-seq谱分析揭示了先前未表征的SSR相互作用,包括SSR:DNA结构不明显的特异性决定簇。并且使用Rec-seq特异性谱来预测Tre和Brec1重组酶的脱靶底物,包括内源性人基因组序列,并证实它们在人细胞中重组这些脱靶序列的能力。这些发现将Rec-seq确立为一种高分辨率方法,用于快速鉴定具有单核苷酸分辨率的重组酶的DNA特异性,并为其进一步发展提供信息。
来源:Plant_ihuman iNature
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3MTE3MjUyOA==&mid=2247500695&idx=4&sn=7c9a467165d39315271dfead9efcb634&chksm=fce6b048cb91395e64f4484bff2c525b5c8025ee63d5fc92e41c65995562cf4dd73eab67b119&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
科学家为基因魔剪“CRISPR”赋予了新的开关

植物特异性非编码RNA功能分析

增强CRISPR特异性以避免发生断裂 | Nature论文

新CRISPR技术可修复精子缺陷,完美婴儿越来越近

过去CRISPR系统无法靶向的特异性突变将迎来转机
胰腺特异性抗原(PaA)
颅内非特异性感染
工程CRISPR核酸酶DNA结合特性、切割的特异性及脱靶效应

番茄叶绿体发生的组织特异性

Nat Biotech丨提高CRISPR特异性的新方法