科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-10-24
来源:JIPB

由CRISPR/Cas9技术衍生的基因激活系统是植物基因超表达的新工具。该系统包括由核酸酶失活的Cas9(即dCas9)和转录激活结构域融合而成的dCas9人工转录激活因子以及靶向目标基因启动子的sgRNA,通过sgRNA引导dCas9人工转录因子与目标基因启动子结合而实现该基因的转录激活。中山大学李剑峰课题组在前期研究中研发了一种第二代dCas9人工转录因子——dCas9-TV系统。该系统使用了6个拷贝的黄单胞菌TALE和2个拷贝的疱疹病毒VP64转录激活结构域串联组成一个复合型转录激活结构域(简称TV),从而能够在拟南芥植株中实现单个sgRNA介导的高效基因激活(Li et al. 2017)。dCas9-TV随后可以通过红光诱导表达的启动子驱动而实现红光诱导的植物基因超表达(Ochoa-Fernandez et al. 2020)。通过搭载不同的dCas变体(如dCas9-NG和dCas12b),dCas-TV可结合的靶基因启动子序列得以进一步拓展(Ren et al. 2019; Ming et al. 2020)。此外,Cas9-TV还能在水稻细胞中实现更为高效的基因编辑,这可能得益于TV结构域可以帮助打开封闭的染色质结构(Liu et al. 2019)。然而,dCas9-TV在水稻中的基因(特别是多基因)激活效率以及遗传稳定性仍有待评估。此外,在动物和植物细胞中,由于dCas9人工转录因子与内源转录因子的竞争作用,前者(包括dCas9-TV)通常更易于激活本底表达较低的目标基因。这说明dCas9-TV介导的基因激活策略仍有优化空间。
JIPB近日在线发表了李剑峰课题组题为“Multiplex and optimization of dCas9-TV-mediated gene activation in plants”的研究论文。该研究表明dCas9-TV能够在水稻植株中同时高效激活OsER1和OsGW7两个基因,实现基因超表达表型的堆叠,并成功利用“正反馈”策略提高了该系统对拟南芥中高本底表达基因的转录上调水平。
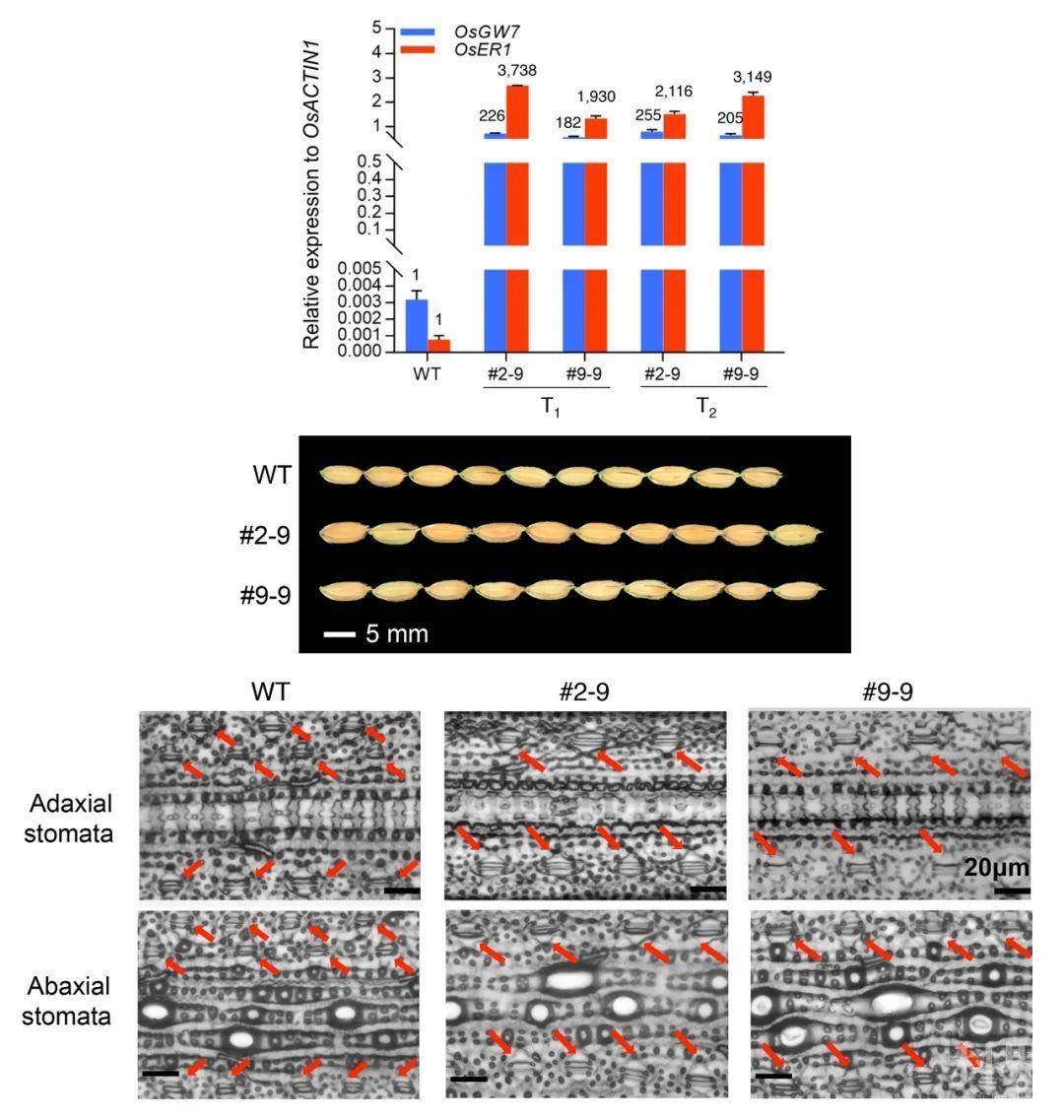
该研究首先利用dCas9-TV搭配Pol III启动子驱动表达的sgRNA(OsER1)和sgRNA(OsGW7)在T0代转基因水稻植株中实现了对内源OsER1和OsGW7基因的同时激活(OsER1激活近4,000倍,激活后的表达水平接近OsActin1的3倍;OsGW7激活200多倍,激活后的表达水平接近OsActin1)。这两个基因的共激活可使植株出现OsER1和OsGW7超表达表型的堆叠,并可维持高效激活至第3代,但在第4代出现不明原因引起的基因激活效率的下降。意外的是,利用dCas9-TV搭配ZmUbi1启动子(Pol II启动子)驱动表达的tRNA-sgRNA(OsER1)-tRNA-sgRNA(OsGW7)-tRNA可在T0代水稻植株中实现更强的基因共激活(OsER1激活近40,000倍,激活后的表达水平接近OsActin1的20倍;OsGW7激活900多倍)。这类植株均表现出生长迟缓和矮小表型,提示过度的基因激活会干扰植株正常生长。该研究还发现,利用靶基因启动子驱动dCas9-TV的表达可形成一个放大dCas9-TV表达的“正反馈”循环,使dCas9-TV同时激活自身及靶基因的表达,从而可在拟南芥中成功提高对高本底表达基因AtFLS2的基因上调水平。此外,基于原生质体的基因激活数据表明,插入酵母GCN4蛋白coiled-coil(CC)序列的dCas9-TV(即dCas9-CCTV)可以诱导该新型人工转录因子像内源转录因子一样发生二聚化,从而提升基因激活效率。
博士生熊翔宇和硕士生梁洁坪为该论文的共同第一作者,李剑峰教授为通讯作者。该论文得到国家转基因重大专项的资助。李剑峰课题组近年来在开展植物免疫信号转导研究的同时,构建和优化了一套全方位的植物基因功能调控工具,包括CRISPR及单碱基编辑器介导的基因敲除(Li et al. 2013; Li et al. 2019),基因沉默(Li et al. 2013; Zhang et al. 2018)以及基因激活工具(Li et al. 2017; Xiong et al. 2020)。
参考文献:
1. Li JF, Chung HS, Niu Y, et al (2013) Comprehensive protein-based artificial microRNA screens for effective gene silencing in plants. Plant Cell 25: 1507-1522.
2. Li JF, Norville JE, Aach J, et al (2013) Multiplex and homologous recombination-mediated genome editing in Arabidopsis and Nicotiana benthamiana using guide RNA and Cas9. Nat Biotechnol 31: 688-691.
3. Li Z, Xiong X, Wang F, et al (2019) Gene disruption through base editing-induced messenger RNA missplicing in plants. New Phytol 222: 1139-1148.
4. Li Z, Zhang D, Xiong X, et al (2017) A potent Cas9-derived gene activator for plant and mammalian cells. Nat Plants 3: 930-936.
5. Liu G, Yin K, Zhang Q, et al (2019) Modulating chromatin accessibility by transactivation and targeting proximal dsgRNAs enhances Cas9 editing efficiency in vivo. Genome Biol 20: 145.
6. Ming M, Ren Q, Pan C, et al (2020) CRISPR-Cas12b enables efficient plant genome engineering. Nat Plants 6: 202–208.
7. Ochoa-Fernandez R, Abel NB, Wieland FG, et al (2020) Optogenetic control of gene expression in plants in the presence of ambient white light. Nat Methods 17: 717–725.
8. Ren B, Liu L, Li S, et al (2019) Cas9-NG greatly expands the targeting scope of the genome-editing toolkit by recognizing NG and other atypical PAMs in rice. Mol Plant 12: 1015–1026.
9. Xiong X, Liang J, Li Z, et al (2020) Multiplex and optimization of dCas9-TV-mediated gene activation in plants. J Integr Plant Biol.
10. Zhang N, Zhang D, Chen SL, et al (2018) Engineering Artificial MicroRNAs for Multiplex Gene Silencing and Simplified Transgenic Screen. Plant Physiol 178: 989-1001.
来源:jipb1952 JIPB
原文链接:http://mp.weixin.qq.com/s?__biz=MjM5ODc5NzY4OQ==&mid=2650208920&idx=1&sn=3087410d2343bb285e179dfcce2d389d&chksm=bec7128e89b09b989099a13861bc94f301d9ad1ad58e79d89fb14178847deab3c304c19a4b70&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

中科院植物所曲乐庆组开发出一种能降血压的转基因水稻
第二届“药食同源与植物代谢”国际学术研讨会在沪举行

植物适应土壤氮素环境的基因选择: 以水稻为例
2017国际植物日之--南开大学站成功举办
《生命科学》出版植物营养与人类健康专刊
水稻小穗发育新基因克隆成功
2015上海辰山“药食同源与植物代谢”国际学术研讨会顺利召开
李新荣:沙漠书写草根论文

水稻自私基因首次被发现

基因融合被证明是 水稻新基因产生的重要机制