科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-12-15
来源:BioArt
肺腺癌(LUAD)是最常见的肺癌组织学亚型,平均5年生存率为15%【1, 2】。相比之下,LUAD的浸润前阶段,例如原位腺癌(AIS)和微浸润腺癌(MIA),在手术切除后5年内不复发的比例接近100%【3-5】。AIS的定义为≤3cm的无浸润性腺癌,而MIA是≤3cm的≤5mm浸润性腺癌【6】。尽管一些重点研究已经确定了AIS和MIA中肺癌驱动程序的突变,但对于驱动这些病变向侵袭性LUAD发展的分子事件仍然缺乏深入的了解【7-10】。原位腺癌和微浸润腺癌是肺腺癌的浸润前病变,并且我们对这些病变的基因组和免疫学特征了解得很少。
近日,来自复旦大学附属肿瘤医院的陈海泉教授团队和来自哈佛大学医学院的Matthew Meyerson团队在Nature Communications上发表文章Genomic and immune profiling of pre-invasive lung adenocarcinoma。研究对24个原位腺癌、74个微浸润性腺癌和99个浸润性腺癌进行全外显子和转录组测序。发现EGFR、RBM10、BRAF、ERBB2、TP53、KRAS、MAP2K1和MET在原位腺癌/微浸润性癌中为显著突变基因,并且揭示了在肿瘤进展过程中频率显著增加的基因组改变的类别, 包括TP53突变、臂级拷贝数改变和HLA杂合缺失。免疫浸润与6p号染色体的拷贝数变化有关,表明6p染色体事件与肿瘤免疫环境之间也存在一定联系。

在该研究中,研究人员在AIS和MIA标本中鉴定了8个显著突变的基因,包括EGFR、RBM10、BRAF、ERBB2、TP53、KRAS、MAP2K1和MET,这些基因先前均被TCGA的LUAD队列中报道过【11, 12】。在该研究的浸润性腺癌病例中,EGFR、TP53、RB1和KRAS为显著突变(图1)。同时,研究者在AIS或MIA样品中发现了MDM2、MYC、TERT、KRAS、NKX2-1和CDK6的扩增(图1),并且在复旦大学附属肿瘤医院肺腺癌患者队列中发现了RIT1的新型扩增。RNA-seq分析揭示了MIA样品中的RET融合以及浸润性腺癌中的ALK和ROS1融合(图1)。浸润性腺癌与AIS/MIA突变频率差异最大的是TP53突变(38%),其次是EGFR和RB1突变(图1)。与AIS/MIA相比,浸润性腺癌的肿瘤突变负荷(TMB)显著更高,APOBEC突变特征活性更高,染色体臂级别事件发生频率也更高。
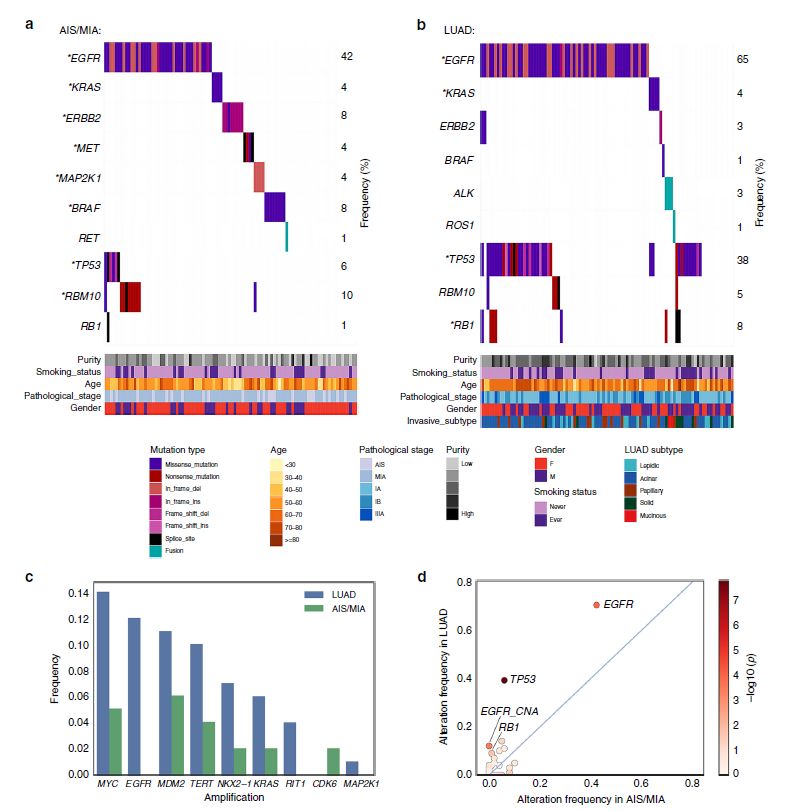
图1
人白细胞抗原(HLA)等位基因的丢失已被确定为肺癌的潜在免疫逃逸机制【13, 14】。在本研究中,研究人员发现3.1%的AIS/MIA和16.7%的浸润性腺癌标本中出现HLA杂合丢失(LOH)(图2)。与浸润前组相比,浸润组中HLA LOH的频率显着增加(Fisher精确检验,p <0.01)表明,HLA等位基因缺失在肿瘤发展过程中起到一定的作用。进一步研究发现,浸润性腺癌中约60%的HLA LOH事件与6p染色体丢失有关。有趣的是,作者发现6p的扩增与T细胞丰度显著相关(Mann-Whitney U检验,p = 0.038,图3),并且在分析B细胞浸润与6p CNA相关时也观察到这种趋势。研究人员随后使用TCGA LUAD研究组的样本测试了免疫浸润与大染色体拷贝数改变的相关性,观察到在控制TMB和整体非整倍性程度后,白细胞分数与6p染色体CNA的相关性最显著(p = 0.0030,coef = -0.74,95%CI:-1.23至-0.25),其次是1q(p = 0.0033,coef = -0.60,95%CI:-1至-0.2)和19p CNA(p = 0.0047,系数= 0.53,95%CI:0.16至0.9)(图2)。6p和1q CNA在复旦大学附属肿瘤医院研究队列中显示从AIS/MIA到LUAD的频率显着增加(Fisher精确检验,p <0.001)。
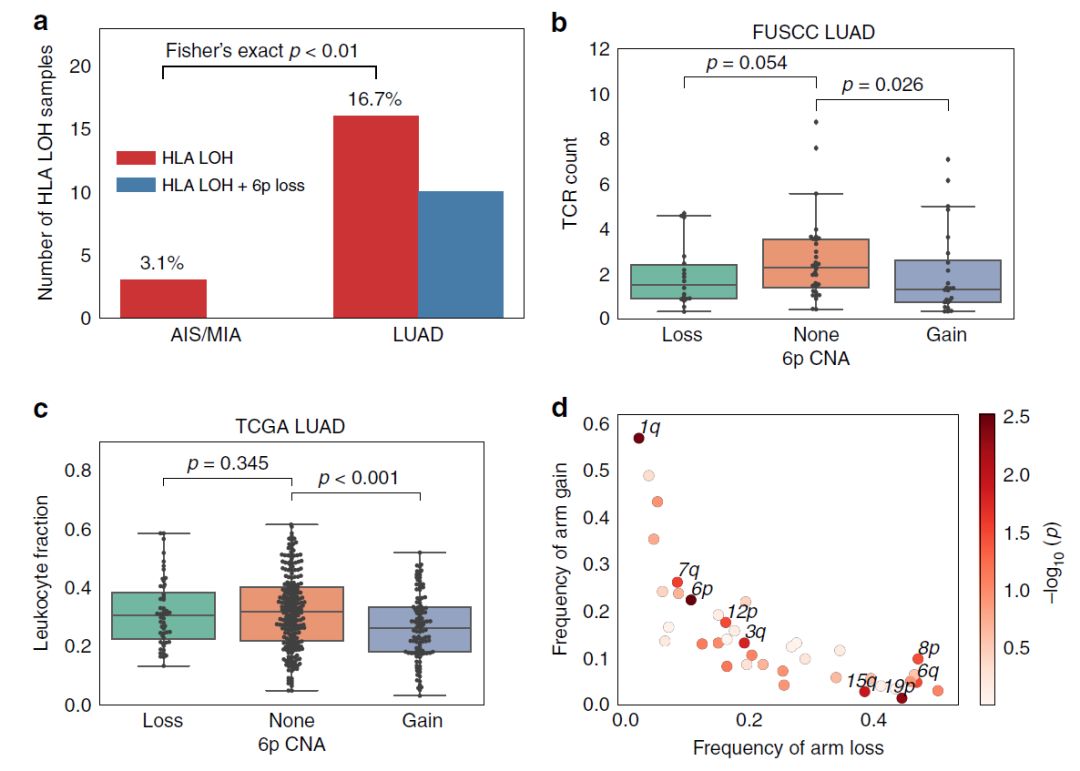
图2
本文揭示了浸润前/浸润性肺癌的基因组和免疫学特征。71%的AIS和MIA患者携带至少一个在RTK/RAS/RAF通路中先前鉴定的癌症基因突变,这与浸润性肺腺癌中的致癌驱动事件高度相似。该研究也揭示了TP53是肺癌浸润的关键介质。先前在Barrett食管中进行的研究表明,TP53在食道腺癌前体中较早发生,然后是致癌性扩增【15】。TP53在原位肺鳞癌中也经常发生突变,这是鳞状细胞癌的癌前病变【16】。本文显示了致癌的驱动突变的高频率,但在AIS/MIA中TP53突变的频率较低。先前的研究表明TP53突变与癌症的潜在侵袭功能相关【17】,本文还表明TP53突变与染色体的非整倍性密切相关,与TGCA的最新研究一致【18】。鉴于先前有关非整倍性与免疫浸润减少相关的报道【18, 19】,本文的研究者提出了特定染色体的拷贝数变化可能影响肿瘤微环境的可能性,提供了对肺恶性肿瘤生物学的新见解,对疾病监测和预后以及未来的治疗干预具有重要意义。
原文链接:
https://www.nature.com/articles/s41467-019-13460-3.pdf
参考文献
1. Siegel, R. L. et al. Cancer statistics, 2018. CA Cancer J. Clin. 68, 7-30 (2018).
2. Chen, W. et al. Cancer statistics in China, 2015. CA Cancer J. Clin. 66, 115-132 (2016).
3. Yim, J. et al. Histologic features are important prognostic indicators in early stage lung adenocarcinomas. Mod. Pathol. 20, 233-241 (2007).
4. Borczuk, A. C. et al. Invasive size is an independent predictor of survival in pulmonary adenocarcinoma. Am. J. Surg. Pathol. 33, 462-469 (2009).
5. Maeshima, A. M. et al. Histological scoring for small lung adenocarcinomas 2 cm or less in diameter: a reliable prognostic indicator. J. Thorac. Oncol. 5, 333-339 (2010).
6. Travis, W. D. et al. International association for the study of lung cancer/American thoracic society/European respiratory society international multidisciplinary classification of lung adenocarcinoma. J. Thorac. Oncol. 6, 244-285 (2011).
7. Murphy, S. J. et al. Genomic rearrangements define lineage relationships between adjacent lepidic and invasive components in lung adenocarcinoma. Cancer Res. 74, 3157-3167 (2014).
8. Izumchenko, E. et al. Targeted sequencing reveals clonal genetic changes in the progression of early lung neoplasms and paired circulating DNA. Nat. Commun. 6, 8258 (2015).
9. Kobayashi, Y. et al. Genetic features of pulmonary adenocarcinoma presenting with ground-glass nodules: the differences between nodules with and without growth. Ann. Oncol. 26, 156-161 (2015).
10. Vinayanuwattikun, C. et al. Elucidating genomic characteristics of lung cancer progression from in situ to invasive adenocarcinoma. Sci. Rep. 6, 31628 (2016).
11. The Cancer Genome Atlas Research Network. Comprehensive molecular profiling of lung adenocarcinoma. Nature 511, 543-550 (2014).
12. Campbell, J. D. et al. Distinct patterns of somatic genome alterations in lung adenocarcinomas and squamous cell carcinomas. Nat. Genet. 48, 607-616 (2016).
13. The Cancer Genome Atlas Research Network. Comprehensive genomic characterization of squamous cell lung cancers. Nature 489, 519-525 (2012).
14. McGranahan, N. et al. Allele-specific HLA loss and immune escape in lung cancer evolution. Cell 171, 1259-1271 (2017).
15. Stachler, M. D. et al. Paired exome analysis of Barrett’s esophagus and adenocarcinoma. Nat. Genet. 47, 1047–1055 (2015).
16. Teixeira, V. H. et al. Deciphering the genomic, epigenomic, and transcriptomic landscapes of pre-invasive lung cancer lesions. Nat. Med. 25, 517–525 (2019).
17. Goh, A. M. et al. The role of mutant p53 in human cancer. J. Pathol. 223, 116–126 (2011).
18. Taylor, A. M. et al. Genomic and functional approaches to understanding cancer aneuploidy. Cancer Cell 33, 676–689 (2018).
19. Davoli, T. et al. Tumor aneuploidy correlates with markers of immune evasion and with reduced response to immunotherapy. Science 355, eaaf8399 (2017).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652478910&idx=5&sn=d6010588f4c45c497fa9536b32dc9955&chksm=84e20e0ab395871cd078a591af5ac2094563d9842ad1b4ee9726f0193e69bcdc81b5e176d007&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

临床癌症基因组学 |《自然-癌症》专题

微生物基因组学和功能基因组学相关软件和数据库的研究进展

王宪辉:“寂寞”科学家钟情“爱热闹”蝗虫

华大基因CEO回应热点质疑
中国科学院北京基因组所开发比较群体基因组学新算法
影像学特征可用于预测肝癌患者生存期

泛基因组如何改变作物基因组学研究和遗传改良
园艺作物驯化研究的基因组学方法

南北方6大遗传差异首次被揭示

如何减少基因组学研究的批次效应?