科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-06-22
6月13日,Molecular & Cellular Proteomics在线发表了中国科学院生物物理研究所研究员杨福全团队和中科院院士陈润生团队,关于长链非编码RNA(lncRNA)编码多肽的系统挖掘和鉴定的研究论文Deeply Mining a Universe of Peptides Encoded by Long Noncoding RNAs。
lncRNAs被认为是一类长度大于200个碱基且不编码蛋白的非编码RNAs转录本。然而,研究表明,较多lncRNAs转录本中的小开放阅读框(small open reading frame,smORFs)可以编码多肽(smORF encoded polypeptides,SEPs),后者可广泛参与肌肉形成、粘膜免疫、RNA脱帽及肿瘤增殖等生物学过程。考虑到lncRNAs转录本及其smORFs的庞大数量,SEPs或代表着一个被忽视且待开发的富含蛋白质活性调节因子的宝库。因此,大规模地发现和鉴定SEPs并系统探索它的功能及其在生物进化中的作用,可为揭示由非编码RNA介导的遗传信息传递方式和表达调控网络的研究,以及从一个不同于蛋白质编码基因的角度为基因组的结构与功能注释提供新的突破口。
SEPs的规模性发现和鉴定仍面临挑战:lncRNA在物种间的保守性相对较差,且具有组织特异性与时空特异性,使得lncRNA编码多肽的组织和时空表达具有很强的动态性;目前发现和鉴定的SEPs数目相对有限,较难对lncRNA编码多肽的生物特征,例如序列信息、保守性、化学性质(如RNA和多肽的稳定性)、结构特征、基因组位置信息和转录本自身结构等,开展系统分析和挖掘,导致基于生物信息学的SEPs发现和鉴定及功能研究仍具挑战;SEPs的翻译与调控机制尚不清楚,有研究报道SEPs的翻译并不完全遵循AUG起始的规则,而是存在以非AUG为起始的翻译,表明SEPs可能存在一些特有的翻译及其调控机制;lncRNA编码多肽的高灵敏度、高通量发现与鉴定能力有待提高。
针对以上局限和挑战,杨福全课题组和陈润生课题组合作,通过对NONCODE数据库中人和小鼠的lncRNA转录本中的smORF进行系统挖掘,分别构建出含有397万和871万条目的人和小鼠潜在SEP理论数据库,并系统整合了基于分子量截留膜过滤和固相萃取的多肽富集策略,建立了基于生物质谱的高灵敏、高通量lncRNA编码多肽的发现与鉴定技术平台。
研究人员利用该技术平台,取得如下成果:(1)在8种人源肿瘤细胞系、3种鼠源细胞系和8种健康小鼠组织中,发现和鉴定了762个具有高可信度的SEP,是目前已知的鉴定数目最多的基于生物质谱技术的SEP数据集;(2)研究显示,部分SEP在多种细胞系或组织中均有表达,而多数SEP只在某类或某种细胞系和组织中鉴定到,具有细胞或组织特异性分布;(3)编码SEP的人源lncRNA转录本主要来源于基因间lncRNA(48.6%),18.6%和17.7%分别来源于外显子lncRNA和反义lncRNA,少部分来源于正义非外显子lncRNA(15.1%);(4)起始密码子统计结果显示,仅有28%的人源lncRNA编码多肽的起始密码子为AUG,而 67%的人源SPEs的起始密码子为non-AUG。以上研究成果将为SEPs翻译与调控机制的研究提供了一定的数据基础,并为基因组中非编码RNA及其基因的系统发现和功能鉴定提供数据和理论支持。
生物物理所博士研究生张青、吴尔重,硕士研究生唐以恒为论文共同第一作者,杨福全、陈润生,副研究员蔡潭溪、研究员骆健俊为论文共同通讯作者。研究工作得到国家自然科学基金重大研究计划项目“基因信息传递过程中非编码RNA的调控作用机制”培育项目、国家重点研发计划等的资助。
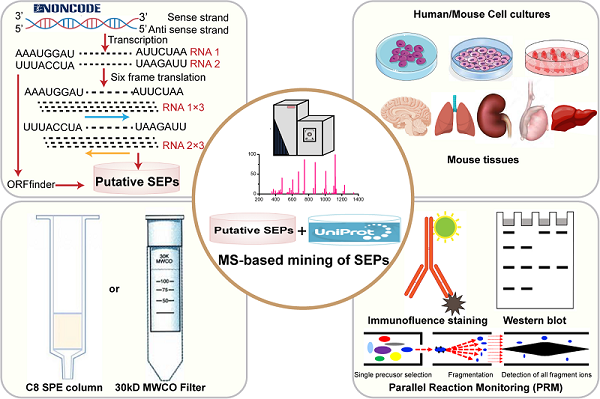
基于生物质谱的lncRNA编码多肽(SEPs)的系统发现和鉴定
来源:中国科学院
原文链接:http://www.cas.cn/syky/202106/t20210622_4794233.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

来看看「LncRNA」如何超越RNA家族其他成员,占据国自然申报热点
RNA编辑

miRNA,lncRNA,circRNA都是RNA,那它们的研究方法一样吗?
核糖体RNA
科学家最新发现,RNA在DNA“失能”时主持生命密码传递工作
Cell 亮点| 反义lncRNA如何调控基因的表达?

我国科学家发现裸鼹鼠的LncRNA抗癌特性
科学家构建RNA结合蛋白的剪接调控作用预测模型----中国科学院

文献详解:m6A相关lncRNA生信挖掘套路解析
【科技前沿】陈玲玲等发表lncRNA综述论文,详述lncRNA的转录、剪接、定位及功能