科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-08-02
大豆(Glycine max)是原产于中国,在世界各地广泛种植的重要经济作物,是人类饮食蛋白、植物油脂、畜牧饲料和化学工业等行业的重要原料[1]。由于大豆基因组复杂,基因重复以及功能冗余现象严重,使得大豆育种需要耗费较长的时间。基因编辑是近年来迅速发展起来的能够快速高效地改变物种性状的基因工程方法。它结合农杆菌介导的遗传转化、基因枪等方法对植物基因组上特定位点进行编辑,进而快速获得具有目标性状的种质资源,彻底改变了作物育种进程。尤其是多基因编辑技术更是作为一种强有力的工具在作物育种中发挥着巨大的应用潜力。CRISPR/Cpf1是近年来新发现的一种基因编辑系统,相较于Cas9,Cpf1具有双重切割酶活性,能够独自地对crRNA前体进行加工。这使得Cpf1在实现多重基因编辑以及染色体片段删除方面具有明显优势。此外,Cpf1具有更为扩展的富含T的PAM识别序列(TTTN),大大增加了它的应用范围。目前CRISPR/LbCpf1实现了在哺乳动物和水稻中的多基因编辑[2, 3],但在大豆中利用CRISPR/LbCpf1进行多基因编辑和染色体片段删除尚未报道。
JIPB近日在线发表了南京农业大学王源超教授团队题为“Large chromosomal segment deletions by CRISPR/LbCpf1-mediated multiplex gene editing in soybean”(https://doi.org/10.1111/jipb.13158)的研究论文。该研究在大豆中建立了一种高效、便捷的CRISPR/LbCpf1多基因编辑系统,实现了8个靶标的同时编辑,并在大豆中成功实现了染色体小片段(<1kb)、大片段(~10Kb)和超大片段(>10Kb)的删除。
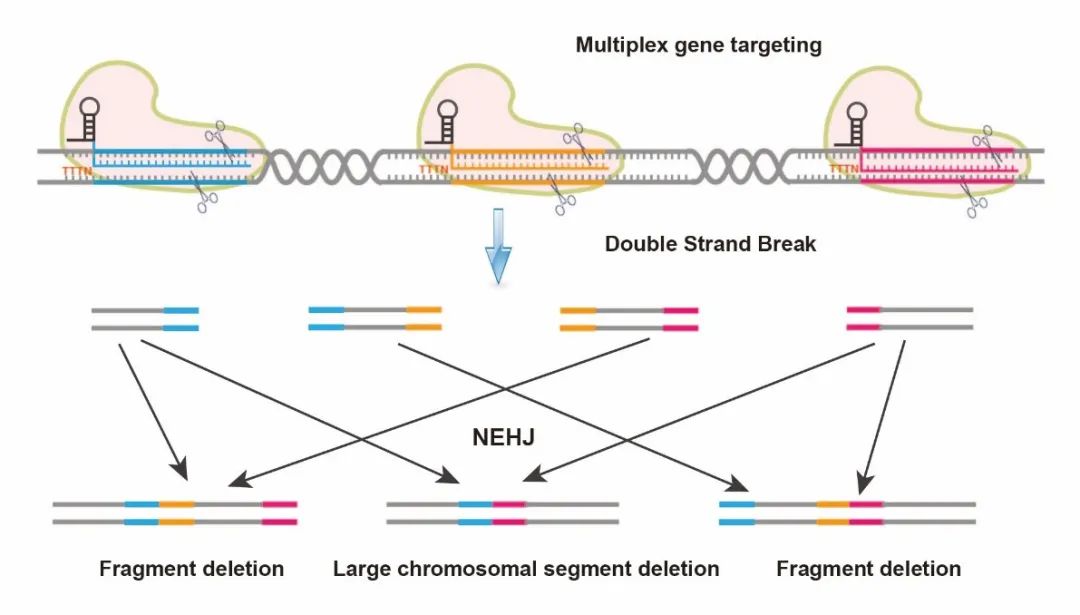
CRISPR/LbCpf1实现多基因编辑和染色体片段删除的示意图
该研究通过比较不同启动子以及crRNA array结构,建立了大豆CRISPR/LbCpf1基因编辑工具,编辑效率可达91.7%,测定到LbCpf1编辑窗口为-3rd-41rdbp。在此基础上,串联多个DR(Direct Repeat)和crRNA,构建了CRISPR/LbCpf1多基因编辑体系,实现了8个靶标的同时编辑,编辑效率可达17.1%。此外利用优化的CRISPR/LbCpf1多基因编辑体系,对大豆FAD2-A基因簇进行片段删除,成功实现了染色体小片段(<1Kb)、大片段(~10Kb)和超大片段(100Kb-1Mb)的删除,片段删除效率分别为36.5%、21.8%、9.5%。该研究在大豆中实现了高效的多基因编辑及染色体片段删除,同时也扩充了在大豆研究中可运用到的基因编辑工具,可望在将来加速大豆分子育种进程。
南京农业大学植物保护学院段凯旋副教授和硕士研究生成媛媛为该论文的第一作者,王源超教授为通讯作者。该研究得到了国家自然科学基金以及中央高校基本科研业务费的资助。
来源:植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247520308&idx=7&sn=24d988dbde6c1eee931eb0dd0e4de939
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
2016年全国植物生物学大会在湖北武汉召开
《生命科学》出版植物营养与人类健康专刊
最新、最全CRISPR/Cas 植物基因组编辑技术综述



基因编辑介导的基因驱动技术助力植物快速改良

植物基因编辑跨入病毒递送时代:浙大李正和组开发植物RNA病毒侵染基因编辑新系统

各国如何监管基因组编辑植物?

我科学家建立高效植物引导基因编辑系统

植物基因编辑技术与应用专题研讨会
第二届“分子植物”国际学术研讨会在清华大学成功召开

sgRNA长度调控编辑类型的植物双功能基因组编辑系统的建立