科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-02-05
来源:BioArt植物
维持植物CHH(H:A, T 或 C)甲基化水平的RdDM(RNA-dependent DNA methylation)通路需要两种植物所特有的聚合酶——DNA依赖的RNA聚合酶IV和V (Pol IV 和 Pol V)【1-3】。在经典的RdDM通路中,Pol IV从转座子序列所转录的转录本会被加工成24-nt小RNA以进一步介导CHH的建立。最近的一些研究揭示了花粉中21/22-nt表观激活小RNA(epigenetically activated siRNAs, easiRNAs) 的合成也需要Pol IV。更有意思的是,Pol IV还参与建立了拟南芥三倍体杂交障碍的建立 【4-6】。
拟南芥花粉的形成伴随着CHH甲基化重编程。CHH甲基化水平在减数分裂过程中大量减少,而后在不对称的有丝分裂过程中,在营养细胞和精细胞的特异位点上得到了恢复 【7-9】。尽管之前的研究暗示了RdDM对于拟南芥减数分裂的可能重要作用,但是在缺失RdDM通路各种重要元件的拟南芥突变体内(Col accession),仅能观察到非常少量的减数分裂异常【9,10】。缺失RdDM通路的拟南芥突变体也基本没有表现出强烈的发育缺陷表型。造成拟南芥对于RdDM缺失耐受的一种可能解释是,它含有较其他植物而言比较低的重复序列(约占135M基因组的24%)【11】。和这个观点相符的是,番茄 (Solanum lycopersicum) 和油菜(Brassica rapa)基因组中含有比拟南芥更高的转座子序列,缺失Pol IV在他们中分别导致了生殖缺陷和种子形成缺陷 【12,13】,但是相关的机制仍不清楚。
近日,瑞典农业科学大学Claudia Köhler院士团队在The Plant Cell上发表了题为Polymerase IV Plays a Crucial Role in Pollen Development in Capsella的研究论文(该团队王振兴博士为论文的第一作者)。该工作以含有更多转座子序列的拟南芥十字花科近亲——荠菜(Capsella rubella,219M基因组中近半为转座子)【14】为研究材料,探讨了Pol IV对于具有不同转座子含量的十字花科植物发育,特别是花粉发育的影响。The Plant Cell期刊配发了题为Pol IV Function is Differentially Essential within the Brassicaceae的评论文章,对该项工作进行了解读。该研究揭示了Pol-IV在植物生殖发育中的作用,为Pol-IV的功能提供了新的见解。该研究极大地扩展了我们对Pol-IV或RdDM相关基因功能的理解。
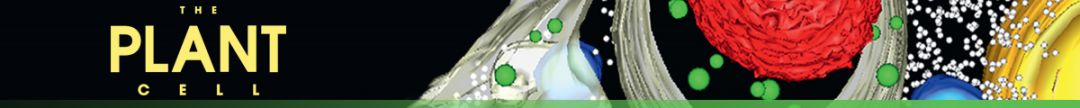


研究人员首先利用Crispr/Cas9删除了荠菜Pol IV关键亚基基因NRPD1的一段编码序列,得到了突变体。该突变体表现为雄性不育,且突变体的花粉大部分停留在了小孢子期。小RNA测序发现,野生型小孢子中积累了大量依赖于Pol IV 的21-24-nt 小RNA,表明easiRNAs 生成于减数分裂过程中或在减数分裂之后立即开始。进一步研究发现,荠菜和拟南芥小孢子中21/22-nt和24-nt小RNA是从同样的转座子区域产生,表明Pol IV生成的前体是这两个小RNA群体合成所共同需要的。和拟南芥相比,荠菜nrpd1突变体中有更多异常表达基因,其中包括许多已知的具有调控花粉发育功能的基因。
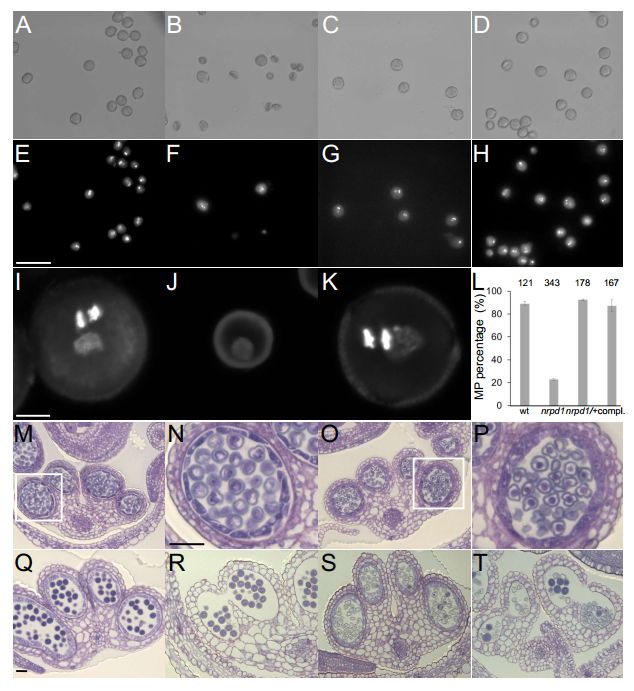
Cr nrpd1 pollen arrest at the microspore stage
综上所述,Pol IV调控荠菜中21-24-nt小RNA的生成,对花粉发育起着重要的作用。同时,该研究结果也说明了研究不同植物模型的必要性。
参考文献:
[1] A. J. Herr, M. B. Jensen, T. Dalmay, and D. C. Baulcombe, “RNA polymerase IV directs silencing of endogenous DNA,” Science (80-. )., vol. 308, no. 5718, pp. 118–120, 2005.
[2] Y. Onodera, J. R. Haag, T. Ream, P. Costa Nunes, O. Pontes, and C. S. Pikaard, “Plant nuclear RNA polymerase IV mediates siRNA and DNA methylation-dependent heterochromatin formation,” Cell, vol. 120, no. 5, pp. 613–622, 2005.
[3] Z. Xie et al., “Genetic and functional diversification of small RNA pathways in plants,” PLoS Biol, vol. 2, no. 5, p. E104, 2004.
[4] G. Martinez et al., “Paternal easiRNAs regulate parental genome dosage in Arabidopsis,” Nat Genet, vol. 50, no. 2, pp. 193–198, 2018.
[5] F. Borges et al., “Transposon-derived small RNAs triggered by miR845 mediate genome dosage response in Arabidopsis,” Nat Genet, vol. 50, no. 2, pp. 186–192, 2018.
[6] P. R. V. Satyaki and M. Gehring, “Paternally Acting Canonical RNA-Directed DNA Methylation Pathway Genes Sensitize Arabidopsis Endosperm to Paternal Genome Dosage,” Plant Cell, 2019.
[7] J. P. Calarco et al., “Reprogramming of DNA methylation in pollen guides epigenetic inheritance via small RNA,” Cell, vol. 151, no. 1, pp. 194–205, 2012.
[8] C. A. Ibarra et al., “Active DNA demethylation in plant companion cells reinforces transposon methylation in gametes,” Science (80-. )., vol. 337, no. 6100, pp. 1360–1364, 2012.
[9] J. Walker et al., “Sexual-lineage-specific DNA methylation regulates meiosis in Arabidopsis,” Nat Genet, vol. 50, no. 1, pp. 130–137, 2018.
[10] C. Oliver, J. L. Santos, and M. Pradillo, “Accurate Chromosome Segregation at First Meiotic Division Requires AGO4, a Protein Involved in RNA-Dependent DNA Methylation in Arabidopsis thaliana,” Genetics, vol. 204, no. 2, pp. 543–553, 2016.
[11] F. Maumus and H. Quesneville, “Ancestral repeats have shaped epigenome and genome composition for millions of years in Arabidopsis thaliana,” Nat Commun, vol. 5, p. 4104, 2014.
[12] Q. Gouil and D. C. Baulcombe, “DNA Methylation Signatures of the Plant Chromomethyltransferases,” PLOS Genet., vol. 12, no. 12, p. e1006526, Dec. 2016.
[13] J. W. Grover et al., “Maternal components of RNA-directed DNA methylation are required for seed development in Brassica rapa,” Plant J, 2018.
[14] T. Slotte et al., “The Capsella rubella genome and the genomic consequences of rapid mating system evolution,” Nat Genet, vol. 45, no. 7, pp. 831–835, 2013.
论文链接:
http://www.plantcell.org/content/early/2020/01/27/tpc.19.00938
来源:bioartplants BioArt植物
原文链接:https://mp.weixin.qq.com/s?__biz=MzU3ODY3MDM0NA==&mid=2247493900&idx=1&sn=6f37875d37b310d5e97ffd100fd4da7b&chksm=fd73736bca04fa7d735bae1052f6f4a42ee0a2ea28c815329ad5f88828a3fe3ef8b85ab362e3#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

维持异源多倍体物种减数分裂稳定性的分子机制被揭示

植物前减数分裂过程中phasiRNA形成的新机制!
新蛋白调控减数分裂细胞核产生用于清除转座子的非编码RNA

EMBO Journal :拟南芥减数分裂同源染色体联会的磷酸化调控机制

Cell Res:蓝斐等发现表观遗传调控减数分裂过程

中科院遗传所程祝宽课题组创制水稻无融合生殖遗传材料
减数分裂起始研究取得进展

Nature Plants新研究发现控制拟南芥减数分裂中交换频率的抑制因子
Annu Rev 植物如何从有丝分裂转换到减数分裂

生殖细胞减数分裂百年之谜获解