科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-05-08
来源:BioArt
撰文 | 王小果
责编 | 兮
小鼠脑是研究中枢神经系统的理想动物模型。目前国际上正在开展许多重大合作, 对小鼠大脑中的神经元类型、神经连接和基因表达从空间尺度和时间尺度上收集海量数据建立图谱。这些数据的成功整合、显示和分析需要三维标准脑图谱。
目前被广泛应用于脑科学中的小鼠二维脑图谱主要是Allen Reference Atlas(西雅图艾伦脑科学研究所发布的)和Mouse Brain in Stereotaxic Coordinates (MBSC),但数字化的三维图谱更符合当前的研究需求。小鼠脑三维图谱第二版存在两个致命的缺点:(1) 在非冠状平面中将冠状视野下注释的2D结构转换为3D体积引起图像失真;(2) 缺乏目前关于大脑神经元的转录组学数据需要的细胞水平分辨率。
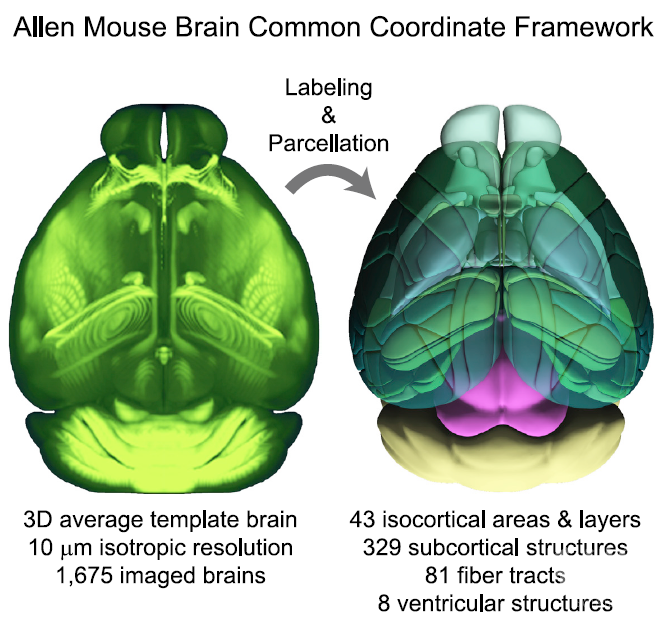
2020年5月7日,艾伦脑科学研究所Lydia Ng和Julie A. Harris团队在Cell杂志上发表文章The Allen Mouse Brain Common Coordinate Framework: A 3D Reference Atlas,通过双光子断层扫描技术获得1675只成年小鼠脑的图像, 构建大脑模型并详细注解, 最终建立小鼠脑三维图谱第三版 (Allen Mouse Brain Common Coordinate Framework,CCFv3)。

由于每只小鼠的大脑在体积和结构上存在细微差异,为了能够设计符合小鼠脑的模型,研究人员计算所有小鼠脑的平均值,并进一步融合为清晰的平均图像,显示明显的解剖学细节。具体来说,首先以50 微米的分辨率进行平均估算,再次以25 微米的分辨率进行融合处理,最终以10微米的分辨率呈现独特、详细、并具有解剖特征的3D图谱模型。在10微米体素分辨率下,这个脑模型包含5.06亿个体素。
基于这个脑模型的内部解剖特征以及转基因小鼠的数据、轴突投射、免疫组化、原位杂交、尼氏染色等具体信息进行注释,Lydia Ng和Julie A. Harris团队组建了三维数据平台,也就是CCFv3。
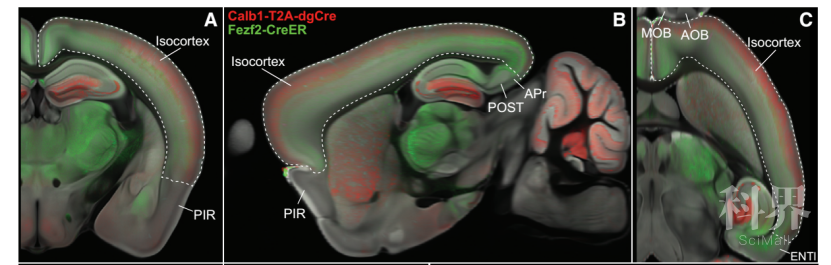
那么在CCFv3中是如何对脑区进行划分的?以皮层为例,皮层是大脑中比较复杂的结构,也是目前研究较多的脑区之一。研究人员利用Calb1-Cre(Calb基因绝大部分在浅表皮层表达)和 Fezf2-CreER(Fezf在深部皮层表达)小鼠区分浅皮层和深皮层;在冠状切面上, 皮层与梨状区域的背侧分界;在矢状面上,与后下托和area prostriata的后方分界;在水平面上,与嗅球的后方分界。通过进一步的分区, 皮层被分为43个亚区。除了皮层的划分外,CCFv3还将皮层下的结构细分为329个区,81条纤维束和8个脑室结构。
CCFv3具有哪些优点呢?第一,与艾伦小鼠脑图谱第二版相比,CCFv3(第三版图谱)的空间分辨率是第二版图谱的1000多倍。第二,CCFv3采集了高信噪比全脑高分辨率图像,更容易分辨解剖学细节。第三,可以通过网站采用二维或三维方式浏览CCFv3,进行很好的人机交互,用户可以选定目标脑区根据目的(脑区基因表达或投射等)查看数据集。第四,除了提供基本功能以外,CCFv3还新增了两项功能:reverse mapping(可在原始图谱中查看注释)和体积估计。
总的来说,本文通过对近2000只小鼠的脑解剖结构进行高分辨重构对脑图谱进行了升级,为小鼠脑研究提供了更多的便利。
原文链接:
https://doi.org/10.1016/j.cell.2020.04.007
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652484172&idx=1&sn=724bf9d3a24d81461c869962b91e809e&chksm=84e221f8b395a8ee8ff7460b981a230b6408bbf708fe18e11b62f61502e99a892e5e1d4313cb#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

高分辨率“资源世界地图”绘成

山东实现优于0.2米分辨率影像陆域全覆盖、2米分辨率周发布
标称分辨率
以创纪录的分辨率看到原子
镜面提升生物显微镜分辨率

《Cell》发布高分辨率完整小鼠大脑三维地图集
阿贝分辨率
光学分辨率
细胞级分辨率人类卵巢图谱发布
分辨率是什么?它又有什么作用?