科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-05-31
来源:BioArt
技术的快速变革,可以使我们以前所未有的空间和分子分辨率观察组织的三维结构。在接下来的七年里,NIH共同基金资助的人类生物分子图谱计划(Human Biomolecular Atlas Program, HuBMAP)计划通过支持技术发展、数据收集和空间图谱开发建立一个可广泛访问的框架,以达到在单细胞水平综合地全面地解析人体的目的。HuBMAP将尽最大可能地整合自己和其它基金机构、项目、联合体和生物医药研究团体的实力,以完成这一共同愿景:即建成一个综合且易访问的包含在健康和各种疾病状态下的人体分子和细胞三维图谱。近日,HuBMAP Consortium在Nature杂志上发表观点文章The human body at cellular resolution: the NIH Human Biomolecular Atlas Program,详细介绍了HuBMAP计划。

HuBMAP(https://commonfund.nih.gov/hubmap)是NIH资助的一个公益项目,旨在开发一种在细胞分辨率水平绘制人体图谱的开放的框架和技术,并且从不同年龄正常人的一些组织中绘制出基础图谱。虽然NIH之前发起的一个项目GTEx,检验了大约一千个人中DNA变异和大块组织中的表达模式,但HuBMAP是一个截然不同的计划,它旨在产生单细胞水平上空间分辨率的分子图谱,但是使用的样本来自更少的个体。为了达到这些目标,HuBMAP联合体被设计成一个紧密相连的合作组织,并且基于“团队科学”的方式,具有开放-分享的文化。HuBMAP联合体(https://hubmapconsortium.org/)将与其它正在进行中的项目积极开展工作,这些项目包括人类细胞图谱计划 (Human Cell Atlas) 、人类蛋白图谱 (Human Protein Atlas) 、生命时间项目(LIfeTime)以及NIH资助的靶向特殊器官和组织的联合体(包括脑、肺、肾、泌尿生殖器官)以及其他新出现的程序。
一、HuBMAP的组织形式和工作方式HuBMAP TMCs将广泛收集和分析大量正常组织,包含两不同性别、不同种族和不同年龄的成年人。这些组织包括(1)分离的或复杂的器官(肾、输尿管、膀胱、肺、胸、小肠和结肠);(2)分散的器官脉管系统;(3)不同微环境中包含动态或运动细胞的系统(淋巴器官:脾、胸腺、淋巴结)。组织将在解剖定位准确的部位根据已经建立的操作步骤收集(可能的话,还会拍照记录),这将保证组织的质量并使降解程度降到最低。除了有特殊需要,在尽可能大的程度上,只要获得批准,开放读取的数据就可以共享(那也就是说,公众可以不经数据委员会批准获得数据),以使生物医学同行最大化地利用它。
TMCs将通过一个互补迭代的两步路径,获得空间上解析的单细胞图谱。HuBMAP所获得数据具有多样性,跨越了宏观-微观尺度(例如,解剖、组织、细胞、分子和基因组)和不同个体,这对于其核心目标是至关重要的。通过对这些数据进行探索和研究,将对人体有更整体的看法。因此,HuBMAP将开发分析和可视化工具,连接空间和分子关系,以帮助产生和理解高分辨率的人体三维分子图谱。
为了处理数据,HuBMAP将为产生和收集的数据搭建利用、拓展、开发的工具和流程。虽然我们期待原始数据加工工具能依赖团队成员的开发,但HuBMAP也将使用和吸纳其它程序和不同研究团体的算法来补充、增强或更新它的流程。最终,依据一系列的用户需求,HuBMAP将开发一个或或多个可供使用的网站。这些开放的资源门户将使用统一认证的标准、并与其它平台(诸如HCA数据合作平台)交互操作,使之可以适于添加、更新和使用新的软件(例如,和Dockstore以及Toil)。HuBMAP数据的一部分将作为开放资源,可以搭载到多种平台上被登录,以增加其使用率。这一基础设施性的工作将使外部开发者可以应用他们自己的密码、应用程序、开放应用程序界面和数据纲要来进行定制化的加工,分析HuBMAP数据以及其它来源的数据。更多地,通过和其他全球和NIH的项目组一起积极工作,可以在跨门户和平台上克服浏览、搜索、汇集和分析数据的障碍。在同一具有高空间分辨率、敏感性、特异性和通量的组织中,定量成像不同类的生物分子是开发详尽的组织图谱的中心工作。虽然目前没有单一的技术可以完全达成这些挑战,这种开发和随后互补能力的倍增提供了一种可加快组织图谱化效果的途径。HuBMAP创新技术团队旨在开发一些创新方法,达成突破现存的最先进技术的局限性的目的。例如,为了对多种组织中的RNA和蛋白进行成像,变革性技术诸如SABER、seqFISH和Lumiphore probes实现的信号扩增将被改进以提升放大能力、敏感性和通量。进一步地,新质谱成像技术将使高空间分辨率和敏感性的同一组织切片中的数百种脂肪、代谢物和蛋白定量成像。在这个项目中也有机会开发和检测新的技术。这些尝试都将得益于开发新的计算工具和机器学习算法。而这些工具或算法是在前导期为了整合不同形式的数据,首先从一个共同的组织中优化得到的。
五、挑战六、资源和团队参与
HuBMAP是建立高分辨率人体细胞和解剖图谱国际使命的重要组成部分,始终致力于和之前提到的项目的密切合作与协作,以建立一个容易使用的平台和交互数据集,这将促进高分辨率人类图谱的完成。围绕开放数据、工具和路径的共享的指引性原则将有助于不同项目间的合作以及对其所产生数据的分析。为了达成这一协作,HuBMAP和其它项目将并肩工作,来处理共同的计算性难题(比如细胞注释)。他们工作的方式包括:聚焦于解决遇到问题的正式或非正式的聚会,精心安排的联合基准化分析法以及亲历亲为的大型聚会和研讨会。另一个潜在的密切合作案例是结肠的研究;多种HuBMAP、人类肿瘤图谱网络以及维康信托资助的项目还将由Leona M.和Harry B. Helmsley Charitable Trust资助的项目补充资助。对于关注部分不同区域和疾病(例如,正常组织、结肠癌和克罗恩病)的课题,确保所有项目收集的数据能整理成可获取的统一格式是重要的,HuBMAP将积极参与这方面的努力。作为坚实的下一步,HuBMAP将与其它NIH项目密切合作,并计划在2020年春天和人类细胞图谱计划一起组织一个联席会议,确认并开展协调与合作工作。同时,HuBMAP参与了其它工程项目(如人类细胞图谱或人类肿瘤图谱网络)的会议和活动,由此建立密切联系。我们已经开创了一系列开放的会议来开发CCF,比如最近举行的和肾脏精准医学项目的合作就算一个。实际上,参与、拓展到更宽的科学团体和其它图谱中心是确保HuBMAP产生的资源能够被广泛驱动以产生持续的影响。为了确保 HuBMAP的浏览器和可视化工具是有价值的,该项目将与解剖学家、病理学家以及可视化及用户体验专家(包括那些在虚拟现实和增强现实方面具有专门知识的专家)密切工作。正如上文描述的,我们期待这个计划中正规样品的多样性将促进有价值的比较分析。通过这种分析,查明整个生命过程中,包括功能紊乱和疾病新发阶段不同个体中细胞和组织结构的定位。为了与领域专家密切合作,这个项目将根据使用情况搭建自身的资源,并通过示范项目为未来提供机会。我们也期待这些数据在以下方面发挥作用:产生新的生物医学假说,组织工程,开发能模仿时空互作的仿真、对组织特征的机器学习和教育用途。
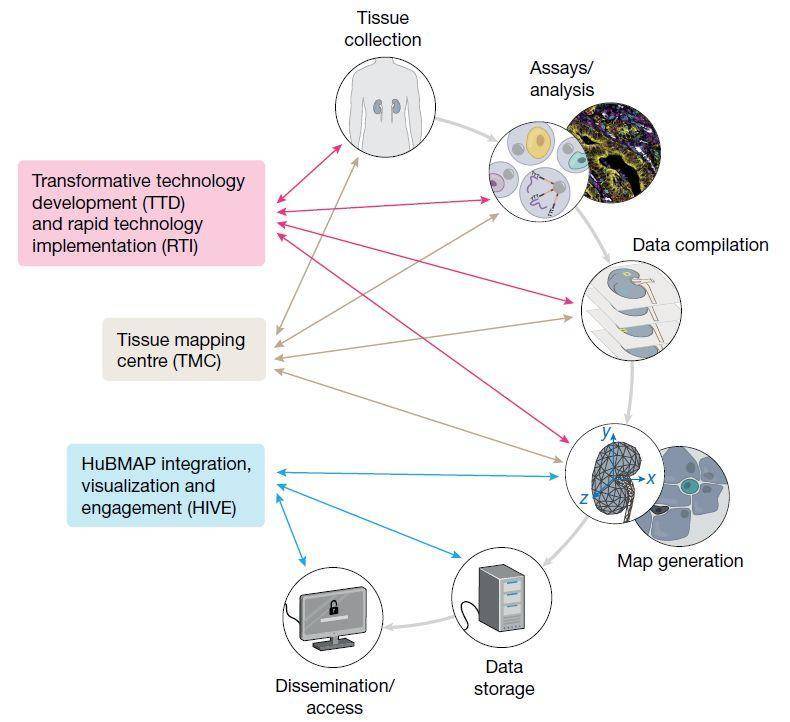 HuBMAP项目示意图
HuBMAP项目示意图七、结论
类似人类基因组的发布,我们期待这第一个参考性的三维组织图谱将代表其最终范围和最终影响的冰山一角。HuBMAP正与其他起始者密切合作,期望通过在正常人体中对关键器官建立一种高分辨率的图谱并捕获个体间的差异来建立一个基础,而这也是对组织生物学和细胞生态系统的新贡献。鉴于HuBMAP关注的焦点是空间分子制图,这个联合体将为此而努力,包括领导数据类型分析方法的开发,开发一个通用的坐标框架来整合数据。最终,我们希望在机体组织的组织方面能催化产生新的观点,这依据的不仅是哪一种细胞临近另一种细胞,而是也有能够定义这些细胞和他们的表型以及功能互作的基因和蛋白表达模式。除了鼓励这些建立的项目内、项目间的合作,对照HuBMAP整体的使命,我们预想了一种容易进入的、公开的用户界面。通过这个界面,数据可以用于在单细胞水平观察分子形态、感兴趣分子的通路和网络,以及给定的感兴趣的细胞类型的时空交换。研究者也将能够用丰富的宏数据的标准格式浏览、搜索、下载和分析数据,久而久之,研究者就能够质问和分析类似程序中的数据集。
重要地,我们相信这个项目对单细胞水平空间方式中不同类型多组学信息的编绘,将成为推进我们理解人类生物学和精准医学的重要步骤。这些数据有潜力重新定义细胞的类型和亚型以及它们在组织内和组织间的关系,这将超越通过标准方法(比如显微镜和流式细胞术)获得的传统理解。我们希望这个工作将成为基础的一部分,使诊断询问、模拟、导航和靶向治疗的介入达到史无前例的分辨率,来革新生物医学领域。
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652486416&idx=1&sn=8ac7a85e34b3b26f2ebb6f526ddc0983&chksm=84e228a4b395a1b270c0feeff82e5d1a60b3f299ef00b4e41ac685439ac33ffd8abba059fe58#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

最高分辨率图像显示DNA分子活性惊人
Cell:在单细胞分辨率下揭示小鼠大脑衰老的分子和空间特征
解析甲状旁腺激素受体三维结构登上《科学》杂志
基于Landsat 8的成都市城市热岛时空格局变化研究
解析人类胆汁盐外排蛋白三维结构
【大师讲堂】“单分子下拉”——以单分子分辨率解析蛋白“身份”
上科大揭示首个卷曲受体三维结构

科研人员研制出分子尺度分辨率干涉定位显微镜
重置打印技术构建液态金属三维结构


学术 | 高分遥感在自然资源调查中的应用综述