科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-08-25
来源:宏基因组
某些淡水湖泊中的大噬菌体或能加速好氧甲烷氧化Large Freshwater Phages with the Potential to Augment Aerobic Methane Oxidation
杂志:Nature Microbiology [IF: 15.540]
DOI: https://doi.org/10.1038/s41564-020-0779-9
发表日期:2020-08-24
第一作者:陈林兴(Lin-Xing Chen)
通讯作者:Jillian F. Banfield (jbanfield@berkeley.edu)
主要单位:美国加州大学伯克利分校地球与行星科学学院(Department of Earth and Planetary Sciences, University of California, Berkeley, CA, USA)
写在前面噬菌体是指能够感染微生物的病毒,其基因组大小通常为55 kbp左右。自然界和动物(包括人)肠道内也分布着基因组在200 kbp以上的噬菌体(Jumbo phage 或 huge phage;大噬菌体)。到目前为止,人体和其他动物肠道内发现的基因组最大的噬菌体是 megaphage,约为550 kbp (Devoto et al. 2019),而自然界中的则约为735 kbp (Al-Shayeb et al. 2020),它们的基因组甚至超过了某些细菌和古菌的基因组大小。虽然大噬菌体在自然界和动物肠道中广泛存在,但我们对它们的代谢潜能以及它们在地球元素循环中所扮演的角色却知之甚少。
研究背景油砂(oil sands)是指富含天然沥青的沉积砂(也称为“沥青砂”),是一种沥青(含量10-12%)、沙和粘土(80-85%)以及水的混合物。地球上已知的油砂中,85%集中在加拿大阿尔伯塔省(Alberta, Canada)北部地区。加拿大油砂中沥青的总含量达4000亿立方米,是世界上最大的沥青资源。油砂的开采会带来诸多环境问题,其中油砂分离所产生的尾矿(油砂尾矿)是主要的环境污染物。油砂尾矿的堆积和扩散会破坏野生动物的栖息地,同时可以持续产生硫化氢,氨和甲烷等,当它们进入大气和水体后,便会引起其他一系列的环境问题。解决油砂尾矿环境问题的方法之一是在开采地附近建立人工湖来存储这些废弃物,并在其上面覆盖一定深度的水体来进行隔绝。与此同时,采取相应的手段,使得人工湖水体可以维持相当的含氧量水平,以便硫化氢,氨和甲烷等污染物能够在水体中被微生物所利用(主要通过氧化),生成其他无害或低害的产物。本研究对加拿大地区的第一个商业化油砂尾矿处理人工湖(Base mine lake; 以下称BML)进行了为期四年的水样收集(2015-2018;图1a),并结合理化测定和宏基因组学分析,以揭示其中好氧甲烷氧化菌的多样性等特征 (Chen et al. 2020)。
摘要越来越多的证据表明,大噬菌体在各种微生物群落(包括动物肠道和自然界)中都非常常见,但我们对它们的代谢潜能,或者说是它们在生态系统中的潜在作用却知之甚少。在本研究中,我们从含有好氧甲烷氧化菌的淡水湖泊系统中,重建了22个大噬菌体的基因组(其中18个恢复了完整的基因组)。这些大噬菌体基因组的长度为159-527 kbp,并且都编码了pmoC基因,因此被称为“pmoC-噬菌体”。pmoC基因是颗粒性甲烷单加氧酶(particulate methane monooxygenase;pMMO)的关键亚基,而pMMO是自然界中最主要的甲烷氧化酶。这些大噬菌体的PmoC序列与样品中共存的好氧甲烷氧化菌(包括Methyloparacoccus, Methylocystis和Methylobacter)的相似性高达 90%以上,并在系统发育上与来自这些细菌的PmoC序列具有一致性。另外,pmoC-噬菌体的丰度与样品中共存的好氧甲烷氧化菌的丰度,具有很强的相关性,这些证据支持了这些甲烷氧化菌是相应的pmoC大噬菌体的宿主。来自瑞士罗特湖(Lake Rotsee, Switzerland)的转录组学数据显示,某些大噬菌体的pmoC基因具有高度表达活性。有趣的是,群落中生长速度最快的好氧甲烷氧化菌被3个pmoC-噬菌体感染。综上,我们认为,在pmoC-噬菌体感染其宿主的过程中,它们会增强细菌的甲烷氧化能力,这暗示了pmoC-噬菌体具有调节甲烷这种强力温室气体从淡水湖泊中释放的潜能。本研究将会推动对噬菌体相关的pmoC基因的功能研究,其中包括验证噬菌体PmoC是否与好氧甲烷氧化菌编码的额外PmoC拷贝具有相似的功能。
方法1.Genome-resolved metagenomics: 宏基因组拼接,覆盖度计算,基因组装箱(MetaBAT),基因组curation(ggKbase)
2.好氧甲烷氧化基因预测:Prodigal基因预测,usearch功能注释(kegg, uniref, uniprot)
3.噬菌体序列预测(ggKbase):噬菌体特异性基因(large terminase, major capsid protein等等)
4.噬菌体基因组矫正: 手动(参考 Chen et al. 2020)
5.噬菌体与宿主关系:(1)CRISPR-Cas靶向分析;(2)pmoC相似度与co-occurrence
6.其他:详见文章
结果1.Methyloparacoccus_57是BML中主要的好氧甲烷氧化菌宏基因组分析恢复了共9个好氧甲烷氧化菌的基因组(图1b),除一个属于alphaproteobacteria纲之外(只在BML的水源样品BML_S中被检测到),其他8个都属于gammaproteobacteria纲,而Methyloparacoccus_57是其中丰度最高的。同时,reads mapping分析发现Methyloparacoccus_57在所有的28个BML样品中都存在,但在有些样品中丰度极低。通过对已发表的测序数据进行重新分析,我们发现Methyloparacoccus_57也是其他油砂尾矿相关水体中主要的好氧甲烷氧化菌。
图1 BML中的甲烷氧化的地球化学和生物学证据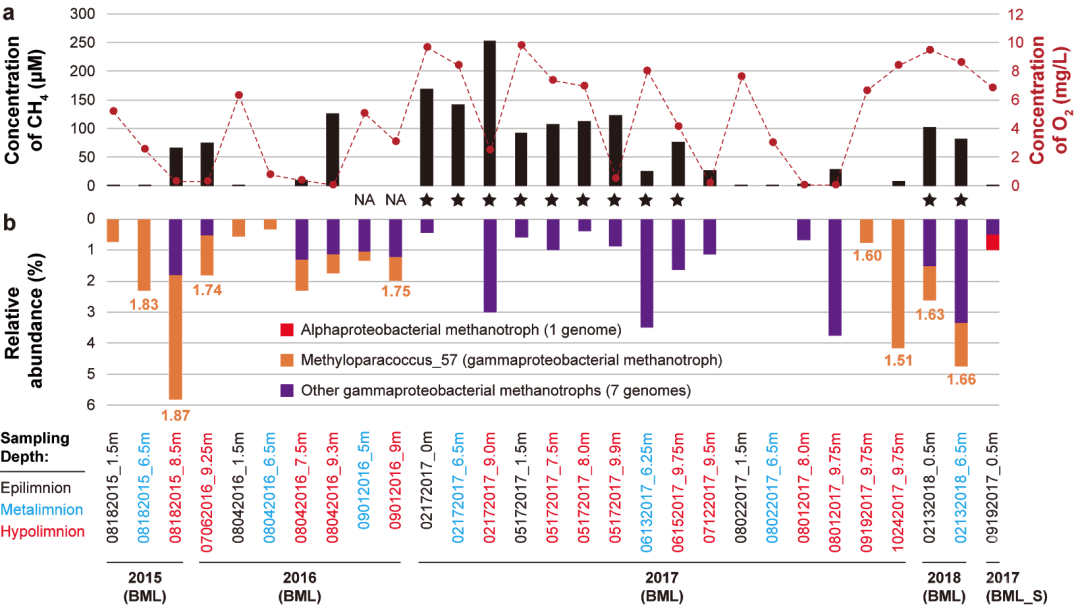
a. 不同采样时间点不同深度的样品中甲烷和氧气的浓度。甲烷氧化活性较低或无活性的样品用星号表示。NA,不可用。b. 甲烷氧化菌的相对丰度。橙色字值表示Methyloparacoccus_57的生长速率(iRep值,当该基因组在相应样品中的测序深度至少为5×时方可进行计算)。
2.编码pmoC基因的噬菌体序列在进行甲烷氧化潜能的分析时,我们发现一些含有噬菌体特异性基因(如 large terminase,major capsid protein等)的序列同时编码了pmoC基因。我们知道,pmoC基因是颗粒性甲烷单加氧酶(particulate methane monooxygenase;pMMO)的一个亚基,而pMMO是自然界中最主要的甲烷氧化酶,同时它的任何亚基从未在病毒基因组中被报道过。与此相关的是,氨单加氧酶亚基C(即amoC)曾在Thaumarchaeota的噬菌体基因组中被发现 (Roux et al. 2016; Ahlgren et al. 2019)。通过手动的基因组矫正,我们从BML和已发表的油砂相关的宏基因组数据中一共恢复了4个具有pmoC的噬菌体基因组;同时重新分析了来自其他已报道的具有甲烷释放的淡水湖泊的宏基因组数据(约200个样品),并获得了18个编码pmoC的噬菌体基因组。这22基因组中,有18个是完整的,因此我们证实这些噬菌体并未编码其他的pMMO亚基(即pmoA和pmoB)。
3.pmoC-噬菌体的基因组特征和系统分类除了上述的22个pmoC-噬菌体之外,我们还恢复其他5个与之相近的噬菌体基因组。这27个噬菌体基因组的大小为159-527 kbp(GC含量:32-44%),编码了224-559个基因,并且有些基因组编码了高达29个transfer RNA(tRNA)。基于13个噬菌体蛋白的串联序列的系统发育分析(图2),上述噬菌体都属于肌尾噬菌体科或肌病毒科(Myoviridae)。
图2 pmoC-噬菌体的系统发育和宿主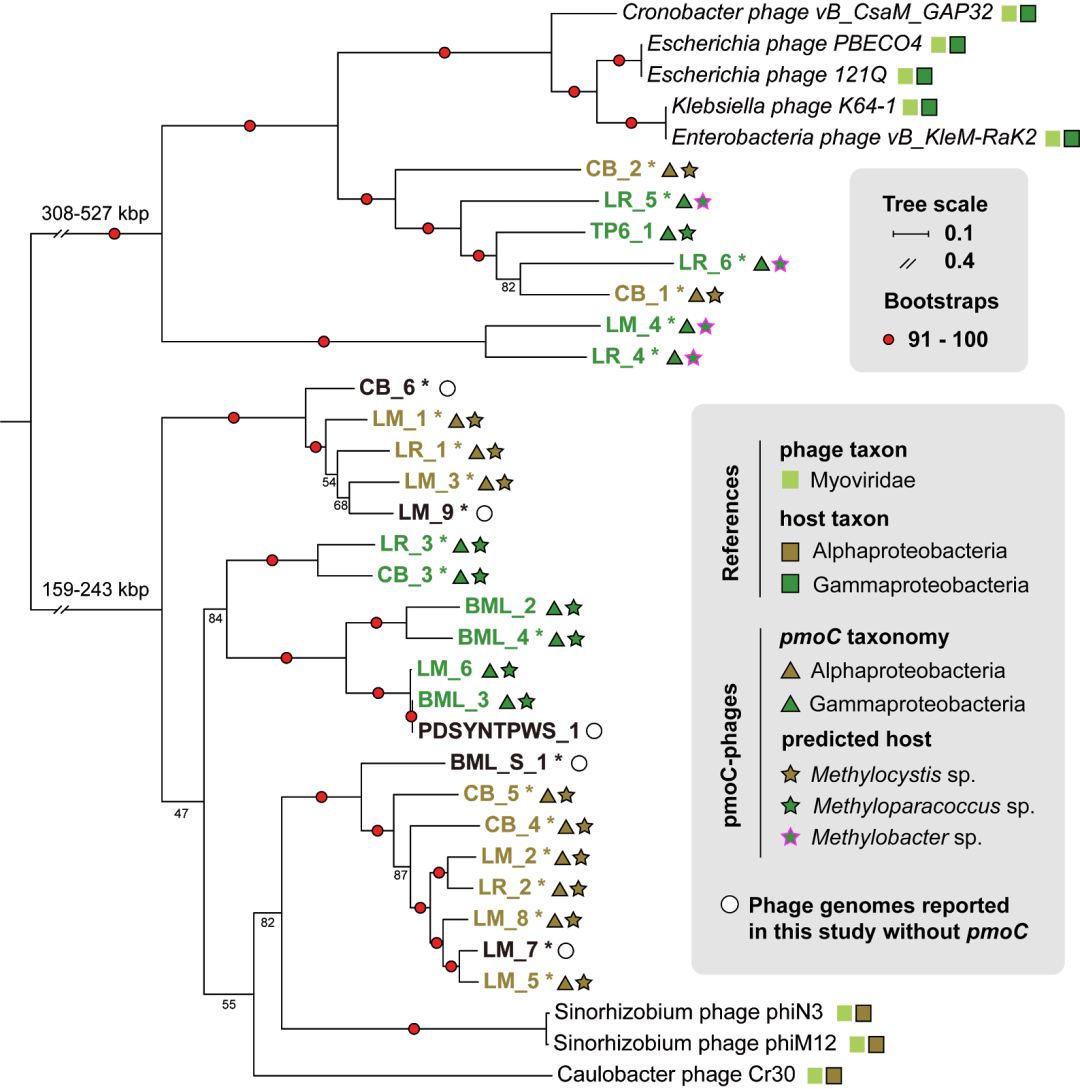
星号表示此论文中报道的完整噬菌体基因组。pmoC-噬菌体及其宿主的系统发育分类用彩色正方形,三角形或星形表示。红色圆圈表示bootstraps ≥ 91,否则显示为数字。
4.pmoC-噬菌体的宿主CRISPR-Cas targeting 分析发现Methyloparacoccus_57是pmoC-噬菌体BML_4的宿主,但未发现CRISPR-Cas系统抵抗其他pmoC-噬菌体的证据。基于噬菌体的pmoC是通过基因横向转移从它们的宿主获得的,我们便通过pmoC的序列相似性来确定各pmoC-噬菌体的宿主。分析结果表明,pmoC-噬菌体的宿主包括了Methyloparacoccus, Methylocystis 和Methylobacter 的相关物种,同时,pmoC-噬菌体及其宿主的co-occurrence分析结果支持了这一结论。
5.pmoC-噬菌体的代谢潜能为了揭示在pmoC-噬菌体中,是否有其他基因与pmoC 基因具有相关的功能,我们对上述27个噬菌体和相关的参考基因组进行蛋白家族分析。结果表明,pmoC 是所有pmoC-噬菌体唯一共同编码的基因,但heat shock protein HSP20基因在22个pmoC-噬菌体的19个中被发现,而且在其中的6个pmoC-噬菌体中,HSP20与pmoC基因相邻。因HSP20在蓝藻细菌的噬菌体(即cyanophages;与pmoC-噬菌体具有系统发育相关性)中广泛分布,且HSP20也被另外5个非pmoC-噬菌体编码,因此HSP20与pmoC的功能相关性难以确定。
6.pmoC-噬菌体的转录活性我们从Lake Rotsee的宏基因组数据中恢复了6个pmoC-噬菌体基因组。宏转录组分析表明其中基因组大小在300 kbp以上的三个pmoC-噬菌体具有高度活性(它们具有同一宿主),而且pmoC基因与噬菌体组装相关的基因同时具有很高的转录活性,表明pmoC 可能在pmoC-噬菌体感染的后期具有重要作用。有趣的是,在Lake Rotsee发现的所有好氧甲烷氧化菌中,被上述pmoC-噬菌体感染的宿主,具有最快的生长速度。
讨论与Thaumarchaeota 的噬菌体基因组中只发现了amoC 基因的情况一致,pmoC-噬菌体基因组也只编码了pmoC 基因。我们认为,一开始pmoC-噬菌体可能从其宿主同时获得了其他的pMMO亚基,但是仅pmoC 基因就可以显著提高噬菌体的适应能力,因此才被保留了下来。此前的研究表明某些好氧甲烷氧化菌除了编码pMMO操纵子之外,还会编码额外的pmoC 基因,而且这些额外的pmoC 基因是甲烷氧化菌在依赖甲烷氧化生长过程中必不可少的。因此,我们推测,pmoC-噬菌体编码的pmoC 基因可能与甲烷氧化菌编码的额外的pmoC 基因,具有相似的功能;然而,我们需要更多更直接的证据来证实这一推测。
来源:meta-genome 宏基因组
原文链接:https://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247491691&idx=1&sn=8d22564c215cc3df0474a53ea8a40582&chksm=faba0adacdcd83cc146a0a10bfea3d4e743d82b41fd76ac512e2a6daed33da90bfaf040bd4cc#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

噬菌体消毒剂部署“十亿小兵”抗菌

气温升高,水体甲烷将增加或加剧温室效应
宏基因组公共数据挖掘基因组集再发Nature
姜还是老的辣:噬菌体VS超级细菌

细菌休眠机制抑制噬菌体蔓延

小麦D基因组完整图谱首绘成功

中国人类基因组研究有望“弯道超车”

数字基因组

华中农业大学教授赵书红:在基因组上“跳舞”

在细菌基因组中识别噬菌体