科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-08-31
来源:BioArt
近日,MIT的Christopher A. Voigt教授(现任中国科学院深圳先进技术研究院合成生物学研究所的陈业研究员,清华大学药学院张数一助理教授为共同作者)在Nature Microbiology杂志上发表了文章Genetic circuit design automation for yeast。该文章在发展酿酒酵母转录调控元件定量设计的基础上,首次实现了真核生物中基因回路的自动化设计,并实现了大规模基因回路长时间(包含11个转录因子,大于两周时间)的稳定状态切换和动态过程预测。
“转动”生命的密匙
自下而上构建生物基因回路
生命是世界上最复杂难解的命题,长久以来无数生物学家都想要探索生命的奥秘,解构生物本身复杂的调控机制。DNA读写技术的产生和进步催生了新的生物学研究范式:合成生物学家通过编码DNA序列,构建人工生物系统,实现预期功能。由此,自下而上(bottom up)的工程学方法被引入,生物体系可按照从DNA序列、元件、回路、系统被自下而上逐层构建。
人工基因回路的构建,能够对天然调控回路进行简单化处理和重新编程,或引入自然界不存在的人造法则,设计符合目标需求的人造生命体,可以为医药健康、农业环境和工业发酵等领域提供全新的解决方案。然而,研究人员手动设计生物基因回路费时费力且容易出错,因此,研究团队希望开发出基因回路设计工具以改变这种状况,自动化和程序化地完成基因回路的设计、构建以及测试的过程。
将基因回路和电子电路的相似之处进行类比,发现两者都将输入信号经过元件的处理运算,从而控制下游输出信号。在电子电路中,不同类型的输入信号被转化为电信号进行处理运算;而在基因回路中,信号可以通过生物分子的相互作用进行传递和运算,转化为下游输出信号。在这些相互作用中,基于DNA——蛋白质相互作用的转录调控有多种形式可以实现不同的运算,同时兼有信号放大的作用,因此最常被用来构建基因回路【1】。
从基因回路到DNA序列
真核系统升级CAD平台
为实现基因回路的程序化设计,MIT的Christopher A. Voigt团队将工程与生物相结合,把广泛应用于工程设计领域的计算机辅助设计软件(Computer-aided-design,CAD)引入基因回路设计,构建了第一代Cello平台。
Cello是一款自动化设计基因回路的软件(图1),基于输入传感器(例如化学分子诱导系统)、回路功能(例如逻辑真值表)、以及相应底盘细胞中的用户约束文件(user constraint file,UCF),自动将目标功能基因回路编译成DNA序列【2】。其中UCF文件包含对应底盘细胞的门电路传递函数、元件搭配和排布规则等内容。因此对于任意底盘细胞,实现CAD需要实现对于单个元件(包括输入传感器和门电路)的定量构建和测试。在大肠杆菌中,Voigt组实现了一系列程序化扩展门电路种类【3】、传递函数【4】和元件排布【5,6】的策略。这是实现计算机辅助设计基因回路的基础,并且用相似策略实现非模式原核生物——拟杆菌(一种肠道主要定殖菌)的基因回路的自动化设计【7】。然而,相同的策略却不能直接应用于真核生物,进而团队将Cello架构升级为Cello 2.0,以适应更多场景的应用。
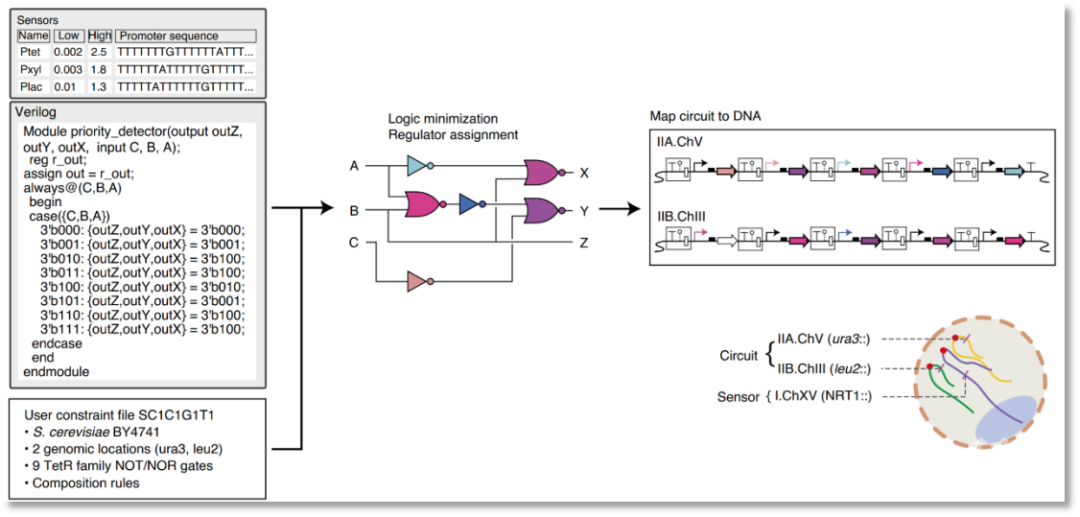
图1. 基因回路自动化设计软件:Cello 2.0
真核生物的调控机制更加复杂。构建传感器和门电路所需的启动子强度受染色体整体结构、核小体占有率、表观遗传修饰等因素的影响。组装回路时,相邻的转录单元中核小体的定位会相互影响导致传递函数改变。此外,各转录模块的终止子也会对上下游基因表达强度产生显著影响。目前有研究在天然启动子中引入调控序列,实现简单回路的构建。但天然启动子经常受到多重因素调控,受生长环境影响较大;若在构建中重复使用可能导致同源重组,降低系统稳定性;强度和调控范围输出也可能不符合需求。
因此,要实现真核系统的基因回路自动化设计需要做到两点:1)设计一系列序列可变可调控启动子,并实现定量输出;2)开发基因元件绝缘和组装策略。
元件、传递函数、正交性
实现真核系统自动化基因回路构建
基因回路中的元件,其输入——输出对应的数量关系(即传递函数),是成功连接回路中不同元件,以实现功能的核心参数。陈业博士等人的前期工作在原核生物中开发了通过模块化构建启动子的方法,成功预测传递函数【8】。本研究中,研究团队将该策略推广到酿酒酵母(Saccharomyces cerevisiae)系统,通过对启动子进行模块化设计,构建了一系列只有100多碱基长度的可调控合成启动子(天然启动子~500碱基),可以系统引入转录调控信号、进行参数优化,并以此为基础构建了一系列传感和逻辑元件。
在构建逻辑元件时,考虑到逻辑回路的设计需求,对传递函数中的调控范围(ymax/ymin)、转录因子表达量相关系数(K)进行优化以避免信号损失,而构建时使用的高协同系数(n>=2)的转录因子则可以保证信号的数字化传递。同时仿照电子电路设计原理,将不同元件的传递函数同一化并满足正交性(图2),最大限度提高元件间的可连接性。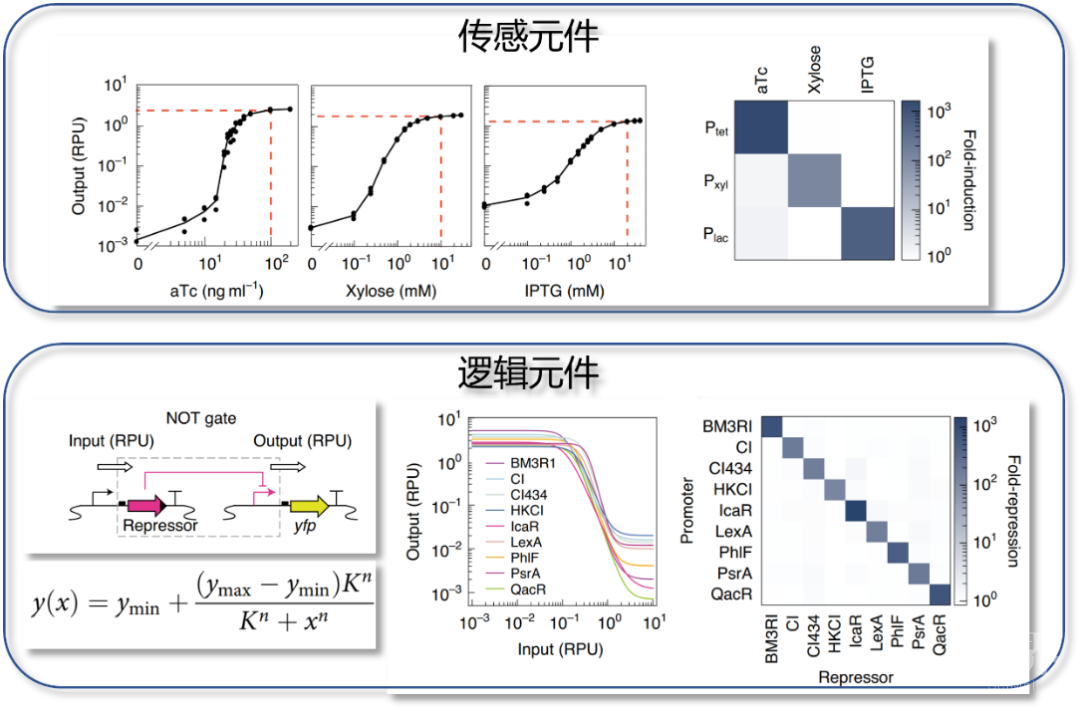
图2. 酿酒酵母元件的设计、传递函数、正交性
在获得了一系列优质元件的基础上,组装需保证各个部分参数不变。为此,研究进而开发了一系列基于核酶和终止子的绝缘序列,这种绝缘化设计避免了基因转录过程的各个核心元件间的功能干涉效应,显著提高了元件的模块化属性,实现了基因表达回路组装时的定量可控。
基于以上元件和组装策略,创建了酿酒酵母的UCF文件,以此在Cello 2.0中构建了一系列3输入的复杂逻辑回路(不少于6个逻辑门元件)。其中最复杂的回路包含11种调控蛋白,16个转录调控单元,33kbp大小。并且所有回路都可以在长达2周的时间内稳定切换状态,其中的动力学过程也可以被模型准确预测。
仿照电路设计基因回路
推动生物系统的自动化设计
这项工作历时4年,其中所包括的开发无串扰信号系统,定量构建对应调控元件,组建大规模基因回路(多达11种调节蛋白),并以此在酿酒酵母中设计出复杂的回路,为将来在其他真核细胞中实现回路设计的标准化和自动化提供了指导。同时,仿照电子电路的设计准则,从理论设计出发,分析需要的元件和参数;通过编写DNA序列,构建符合需要的元件;将元件整合成基因回路并测试,这三步自下而上设计并构建过程,为今后理性设计不同需求的生物系统打下了基础。
随着基因工程规模的上升,需要构建复杂调控网络以协调系统中的不同功能模块:例如,在适当的生长时期转换底盘细胞的能量和代谢途径,或者让细胞群落分工协作完成复杂任务。同时这些调控网络必须能在不同状态稳定工作,并实现精确的动态响应。随着构建基因回路规模的增长,手工设计对多个因素的综合判断越来越难以进行,并且因为回路需要对于多个状态响应,而无法通过定向进化进行优化。因此,需要自动化设计程序来平衡多种约束条件并组合所需的DNA元件。目前研究仍需要在新的底盘细胞中重新设计元件以实现自动化设计(例如本文使用的酵母UCF是重新构建的),但未来,这些都将纳入CAD设计的流程,在不同物种中元件参数可以根据更高层次的设计原则进行编译,实现完全的自动化设计。
原文链接:https://www.nature.com/articles/s41564-020-0757-2
参考文献
1.Brophy, J.A. and C.A. Voigt, Principles of genetic circuit design. Nat Methods, 2014. 11(5): p. 508-20.
2.Nielsen, A.A., et al., Genetic circuit design automation. Science, 2016. 352(6281): p. aac7341.
3.Stanton, B.C., et al., Genomic mining of prokaryotic repressors for orthogonal logic gates. Nat Chem Biol, 2014. 10(2): p. 99-105.
4.Salis, H.M., E.A. Mirsky, and C.A. Voigt, Automated design of synthetic ribosome binding sites to control protein expression. Nat Biotechnol, 2009. 27(10): p. 946-50.
5.Tamsir, A., J.J. Tabor, and C.A. Voigt, Robust multicellular computing using genetically encoded NOR gates and chemical 'wires'. Nature, 2011. 469(7329): p. 212-5.
6.Chen, Y.J., et al., Characterization of 582 natural and synthetic terminators and quantification of their design constraints. Nat Methods, 2013. 10(7): p. 659-64.
7.Taketani, M., et al., Genetic circuit design automation for the gut resident species Bacteroides thetaiotaomicron. Nat Biotechnol, 2020.
8.Chen, Y., et al., Tuning the dynamic range of bacterial promoters regulated by ligand-inducible transcription factors. Nat Commun, 2018. 9(1): p. 64.
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652497681&idx=7&sn=9397b18a14702b9e9f511fc51dd98f73&chksm=84e274a5b395fdb33e63933a6ff0fe9849daaafa2ff2fef454936f2412641c82a857d084746b#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

PBJ:四川大学开发出高效的双向绿色组织特异型启动子

中国科学家首揭灵长类动物发育和寿命调控的关键通路

神经领域中「特异性启动子AAV」的妙用
Nat Comm :科学家捕获代谢适应过程增强子启动子网络变化

【前沿资讯】编辑“启动子”:玉米增产新方法

苹果着色谁说了算?科学家有新发现
Cell子刊: 脑袋和肚子长得不一样, 胚胎发育学家基因启动子上找答案
Nat Microbiol:新研究揭示双向启动子在生命三界中普遍存在
中国科学院揭示热纤梭菌转录调控因子σI的独特启动子识别机制

上海交大揭示早期果蝇胚胎hunchback基因的多启动子差异性调控和信号整合的动力学机制