科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-04-25
来源:BioArt
原标题:专家点评Cell | 陈海威博士等利用大规模筛选揭示了肠道微生物代谢物可以影响宿主的生理功能
点评:朱书 中国科学技术大学
人体肠道微生物通过产生成千上万个小分子代谢物,影响几乎所有的宿主生理活动,从调节肠道免疫到影响宿主情绪和行为【1-5】。这些肠道微生物产生的小分子代谢物可以局部作用于肠道,或者在血液中富集达到几个毫摩尔(mM)【1,6-8】。最近发表的一系列工作通过最新的基因组和代谢组方法,揭示了复杂的肠道微生物代谢物可能影响宿主生理活动,同时肠道微生物也会影响食物来源的小分子和化学药物的消化吸收【9-14】。这些研究都强调继续发开新方法来寻找有生物活性的肠道微生物代谢物的重要性。
G蛋白偶联受体(GPCR)是最大的膜蛋白家族,是感受许多小分子的受体并且多方位调节人的生理活动,包括视觉、情感、痛觉和免疫等等【15】。肠道内的GPCR可以感受细菌代谢物,比如短链脂肪酸可以被GPR41/GPR43感受,丙酮酸和乳酸可以被GPR31感受,以及最近的一系列研究持续发现肠道微生物来源的GPCR配体可以调节宿主生理活动【2,4,16】。因此,肠道微生物代谢物是一个潜在的丰富的GPCR配体库。
但是对于大部分的肠道微生物代谢物,它们的功能未知,其中主要原因之一是缺少一个可以对所有肠道微生物代谢物进行系统性筛选的体系,从中寻找可以调控宿主生理活动的肠道微生物代谢物。
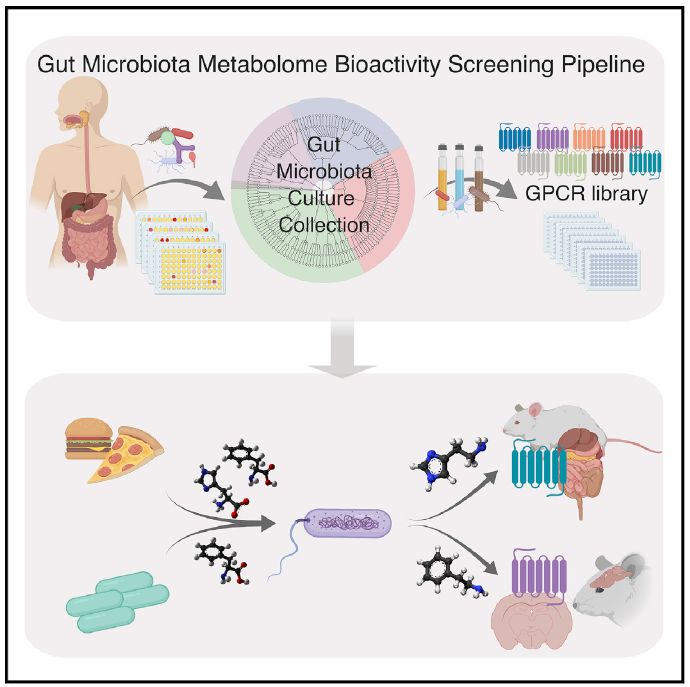
2019年4月18日,耶鲁大学医学院Noah Palm团队(第一作者为陈海威博士)在Cell杂志上发表题为A Forward Chemical Genetic Screen Reveals Gut Microbiota Metabolites That Modulate Host Physiology的长文,利用大规模筛选揭示了肠道微生物代谢物可以影响宿主的生理功能。

这篇研究借助了GPCR领域的一种高通量筛选工具(PRESTO-Tango)【17】,通过GPCR活化来寻找可以调控宿主生理功能的肠道微生物代谢物。研究者通过对一百多种肠道微生物的代谢物对几百种人GPCR之间进行筛选,主要由以下几方面的成果:
(1) 发现多种肠道微生物可以产生激活GPCR的代谢物。
(2) 揭示了肠道内“食物-肠道微生物-宿主轴”和“肠道微生物-肠道微生物-宿主”轴。
(3) 显示肠道微生物代谢物可以局部和系统性影响宿主生理活动。
专家点评

朱书(中国科学技术大学教授,主要从事免疫与肠道微生物相关研究工作)
我在耶鲁免疫系Richard Flavell实验室博士后研究期间曾与本文通讯作者Noah Palm共事4年。Noah Palm在耶鲁免疫系Ruslan Medzhitov实验室进行博士研究期间主要研究蜂毒诱导的II型过敏反应(N Palm, Immunity, 2013),在Richard Flavell实验室进行博士后研究期间Noah曾和另外一位博士后Marcel一起利用致炎性肠道细菌更易结合IgA的特性发明了IgA sequencing,使用IgA标记先进行细菌的流式分选,然后再进行16S测序来高效筛选致病性细菌(N Palm, Cell, 2014)。
在耶鲁成立自己的实验室之后,Noah主要的研究兴趣在于研究肠道微生物如何影响免疫反应进而影响生理和病理进程。Noah实验室除了进一步把发明的IgA seq应用到人类疾病炎症性肠病(IBD)和二型糖尿病(T2D)的致病性菌株的发现当中,也发展和应用了一系列新的筛选系统研究肠道微生物和微生物代谢物对宿主免疫系统的影响。本研究就利用了PRESTO-Tango系统来筛选能激活肠道GPCR的细菌代谢物(H Chen and N Palm, Cell, 2019)。PRESTO-Tango 是2015年开发出来的基于β-arrestin-recruitment assay来筛选全新激活GPCR的配体的平台。Noah Palm实验室的陈海威博士从11个IBD病人的粪便标本中分离出来144个肠道菌株,随后使用这些菌株的培养上清(代谢物混合物)去刺激PRESTO-Tango体系中314个表达GPCR的reporter细胞,不仅验证了这些代谢物可以激活一些Well-Characterized GPCR,同时也发现可以激活一些全新的尚未找到配体的Orphan GPCRs。通过screening,海威博士发现一类肠道菌M. morganii 可以将食物中的histidine转化为histamine(组胺,1957诺贝尔生理学或医学奖授予抗组胺),并调节肠道蠕动。海威博士还发现M. morganii可以将必需氨基酸L-Phe 转化为可穿透血脑屏障的精神类药物主要成分苯乙胺(phenethylamine), 使用苯乙胺且同时抑制单胺氧化酶( monoamine oxidase)的情况下会造成人的死亡。
这个工作使用一个独特的筛选工具来发现全新的细菌代谢物对于宿主生理过程的调节,对于剖析和加深理解细菌代谢组及其与宿主的联系有着极大的帮助。本工作得到了耶鲁大学研究代谢产物化学性质的Jason Crawford、使用yeast display研究微生物和宿主相互作用的Aaron Ring、以及哈佛大学研究GPCR结构的Andrew Kruse的帮助,体现了当前肠道微生物研究当中越来越多的免疫学、微生物学、结构生物学以及化学的交叉融合。
原文链接:
https://doi.org/10.1016/j.cell.2019.03.036
参考文献
1.Fischbach, M.S.D.a.M.A. (2015). Small molecules from the human microbiota. Science 349, 1254766.
2.Husted, A.S., Trauelsen, M., Rudenko, O., Hjorth, S.A., and Schwartz, T.W. (2017). GPCR-Mediated Signaling of Metabolites. Cell Metab 25, 777-796.
3.Smith, P.H., MR; Panikov, N; Michaud, M; Gallini, CA; Bohlooly-Y, M; Glickman, JN; Garret, WS. (2013). The Microbial Metabolites, Short-Chain Fatty Acids, Regulate Colonic Treg Cell Homeostasis. Science 341, 569-573.
4.Tan, J.K.M., C.; Mariño,E; Macia, L and Mackay, CR. (2017). Metabolite-Sensing G Protein-Coupled Receptors-Facilitators of Diet-Related Immune Regulation. Annu RevImmunol 35, 371-402.
5.Yano, J.M., Yu, K., Donaldson, G.P., Shastri, G.G., Ann, P., Ma, L., Nagler, C.R., Ismagilov, R.F., Mazmanian, S.K., and Hsiao, E.Y. (2015). Indigenous bacteria from the gut microbiota regulate host serotonin biosynthesis. Cell 161, 264-276.
6.Fischbach, M.A. (2018). Microbiome: Focus on Causation and Mechanism. Cell 174, 785-790.
7.Pedersen, H.K., Gudmundsdottir, V., Nielsen, H.B., Hyotylainen, T., Nielsen, T., Jensen, B.A., Forslund, K., Hildebrand, F., Prifti, E., Falony, G., et al. (2016). Human gut microbes impact host serum metabolome and insulin sensitivity. Nature 535, 376-381.
8.Perry, R.J., Peng, L., Barry, N.A., Cline, G.W., Zhang, D., Cardone, R.L., Petersen, K.F., Kibbey, R.G., Goodman, A.L., and Shulman, G.I. (2016). Acetate mediates a microbiome-brain-beta-cell axis to promote metabolic syndrome. Nature 534, 213-217.
9.Dodd, D., Spitzer, M.H., Van Treuren, W., Merrill, B.D., Hryckowian, A.J., Higginbottom, S.K., Le, A., Cowan, T.M., Nolan, G.P., Fischbach, M.A., et al. (2017). A gut bacterial pathway metabolizes aromatic amino acids into nine circulating metabolites. Nature 551, 648-652.
10.Guo, C.J., Chang, F.Y., Wyche, T.P., Backus, K.M., Acker, T.M., Funabashi, M., Taketani, M., Donia, M.S., Nayfach, S., Pollard, K.S., et al. (2017). Discovery of Reactive Microbiota-Derived Metabolites that Inhibit Host Proteases. Cell 168, 517-526 e518.
11.Henry J. Haiser, D.B.G., Kelly Chatman, Gopal Sirasani,, and Turnbaugh, E.P.B.a.P.J. (2013). Predicting and Manipulating Cardiac Drug Inactivation by the Human Gut Bacterium Eggerthella lenta.pdf. Science 341, 295-298.
12.Larsbrink, J., Rogers, T.E., Hemsworth, G.R., McKee, L.S., Tauzin, A.S., Spadiut, O., Klinter, S., Pudlo, N.A., Urs, K., Koropatkin, N.M., et al. (2014). A discrete genetic locus confers xyloglucan metabolism in select human gut Bacteroidetes. Nature 506, 498-502.
13.Milshteyn, A., Colosimo, D.A., and Brady, S.F. (2018). Accessing Bioactive Natural Products from the Human Microbiome. Cell Host Microbe 23, 725-736.
14.Zimmermann, M., Zimmermann-Kogadeeva, M., Wegmann, R., and Goodman, A.L. (2019). Separating host and microbiome contributions to drug pharmacokinetics and toxicity. Science 363.
15.Wacker, D., Stevens, R.C., and Roth, B.L. (2017). How Ligands Illuminate GPCR Molecular Pharmacology. Cell 170, 414-427.
16.Morita, N., Umemoto, E., Fujita, S., Hayashi, A., Kikuta, J., Kimura, I., Haneda, T., Imai, T., Inoue, A., Mimuro, H., et al. (2019). GPR31-dependent dendrite protrusion of intestinal CX3CR1(+) cells by bacterial metabolites. Nature 566, 110-114.
17.Kroeze, W.K., Sassano, M.F., Huang, X.P., Lansu, K., McCorvy, J.D., Giguere, P.M., Sciaky, N., and Roth, B.L. (2015). PRESTO-Tango as an open-source resource for interrogation of the druggable human GPCRome. Nat Struct Mol Biol 22, 362-369.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652469130&idx=1&sn=5265b7e61069ba4f50c5f50d5f1ee232&chksm=84e2e43eb3956d2859ecc85f284b523971b8ff137dee19a2d3554c7982e4d7c0222a04ad1ba2&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn