科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-06-20
来源:中科院水生所
原标题:徐国良/唐惠儒/黄开耀等团队合作研究发现衣藻中去甲基化酶CMD1催化以维生素C为底物的5mC去甲基化修饰
DNA甲基化是指DNA序列上特定的碱基通过共价键结合的方式获得一个甲基基团的化学修饰过程,是一种普遍存在于生物体的DNA修饰方式。DNA甲基化能够在不改变DNA序列的前提下改变遗传表现,是表观遗传学的核心研究领域之一。目前的研究表明,DNA甲基化与基因组印记、X染色体失活、转座因子抑制、衰老和癌症发生等密切相关,因此是表观遗传学研究的重点和热点之一[1]。
CpG二核苷酸中的胞嘧啶上第5位碳原子在甲基化转移酶(DNMTs)的催化下,发生甲基化的形成5mC。目前发现5mC广泛分布于原核与真核生物中。在哺乳动物中催化5mC去甲基化主动修饰(AM,active modification)功能的是一种名为TET的双加氧酶。如图1所示,TET双加氧酶可以将5mC逐步氧化为5hmC,5fC和5caC,最终在主动移除基团(AR, active removal)或是被动稀释(PD,passive dilution)的作用下实现5mC的去甲基化[2]。
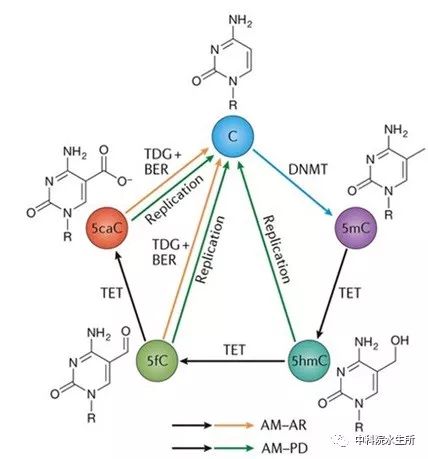
图1 TET酶介导的5mC去甲基化(Nature Reviews Genetics, 2017)
目前,在大多数真核动物中对TET同源蛋白的研究仍是一片空白。中国科学院上海生化与细胞生物学研究所的徐国良研究员课题组,复旦大学生命科学学院的唐惠儒教授课题组和中国科学院水生生物研究所的黄开耀研究员课题组综合运用分子生物学、生物化学、遗传学等技术发现在单细胞绿藻—莱茵衣藻中,TET的同源基因CMD1是如何行使其对5mC中甲基基团的修饰作用的。首先研究人员在莱茵衣藻中鉴定到CMD1等8个TET的同源基因,其共同特点是具有双加氧酶中保守的HxD二价铁结合模体,但不具有酮戊二酸(2-OG)的结合位点。由于酮戊二酸是目前已知的双加氧酶的共同底物,因此说明莱茵衣藻中的TET同源基因可能涉及到全新的DNA甲基化修饰机制。通过体外酶活分析实验,研究人员检测到5mC在CMD1酶的催化作用下甲基基团被修饰,生成两种未知物质。应用核磁共振技术,研究人员最终确定了两种未知物质是互为异构体的5gmC,即在5mC的甲基上通过C-C键加上了一个甘油基团。哺乳动物中的5mC修饰底物来自于酮戊二酸,而CMD1酶催化添加的甘油基团经过一系列同位素标记实验被鉴定出来自于底物维生素C。因此,莱茵衣藻中具有TET的同源蛋白CMD1所催化的,以维生素C为底物对5mC的甲基进行甘油基修饰的全新机制,如图2所示。
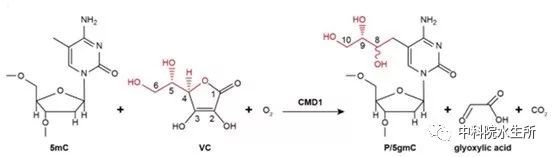
图2 在维生素C与O2的条件下,CMD1酶催化5mC发生甘油基修饰(原文图3c)
为阐明此机制在莱茵衣藻生理过程中的功能,研究人员通过CRISPR/Cas9技术获得莱茵衣藻的cmd1突变体,并且发现突变体具有不耐受高光的表型,如图3所示,此表型与突变体npq4相同。npq4突变体中缺失捕光复合体胁迫相关基因LHCSR3,从而使得非光化学淬灭(NPQ),一种在高光照时抵御光损伤的重要机制受到抑制。进一步研究表明LHCSR3的表达水平在cmd1突变体中下调,而与此现象一致的是LHCSR3序列5 '端相比野生型发生了超甲基化。此实验结果表明CMD1基因介导的5gmC修饰机制能够通过去甲基化调控LHCSR3基因表达,从而影响莱茵衣藻对高光强的适应性。

图3 高光照下,cmd1突变体与npq4突变体的生长受到抑制(原文图4b)
这一研究应用单细胞绿藻—莱茵衣藻作为实验材料,揭示了真核生物中的TET同源蛋白具有一种全新的DNA甲基化修饰机制,并且此修饰机制能够直接调控基因的表达,从而影响光合作用等至关重要的生理功能。本研究对TET蛋白在莱茵衣藻中作用机制的阐明在真核生物的DNA甲基化研究中具有重要的参考价值。此外,基于莱茵衣藻的甲基化修饰机制有望建立新的真核生物全基因组甲基化图谱的绘制方法,而且CRISPR/Cas9等技术的全面应用是莱茵衣藻中新方法发展与成熟的巨大推力。早前芝加哥大学的何川教授课题组已在《Cell》上报道N6mA(N6甲基腺嘌呤)在莱茵衣藻基因组上的独特分布,展示了6mA在真核生物中基因调控的功能[3]。可见莱茵衣藻作为经典的研究光合作用和纤毛的真核单细胞模式生物,由于其易于培养、生长周期短、遗传背景清晰等优越性,在表观遗传学中的研究前景也备受关注。
相关论文“A vitamin-C-derived DNA modification catalysed by an algal TET homologue”已于2019年5月1日在线发表于《Nature》杂志上。原文链接:https://www.nature.com/articles/s41586-019-1160-0
参考文献
●Pastor W A, Aravind L, Rao A. TETonic shift: biological roles of TET proteins in DNA demethylation and transcription[J]. Nature reviews Molecular cell biology, 2013, 14(6): 341.
●Wu X, Zhang Y. TET-mediated active DNA demethylation: mechanism, function and beyond[J]. Nature Reviews Genetics, 2017, 18(9): 517.
●Fu Y, Luo G Z, Chen K, et al. N6-methyldeoxyadenosine marks active transcription start sites in Chlamydomonas[J]. Cell, 2015, 161(4): 879-892.
来源:ihb_152342 中科院水生所
原文链接:http://mp.weixin.qq.com/s?__biz=MzI5MjcwODM0NA==&mid=2247484078&idx=1&sn=7ad50e5e7b4daffefbfceab7a352f2a8&chksm=ec7c0839db0b812f1c5ca87c99a60fc28b442a551211ca386bc741f2e38100da3fee4e267435&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn