科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-06-21
来源:ScienceAAAS
中国科学院青年创新促进会 李家堂
(中科院成都生物研究所)
评述论文:Genetic basis of ruminant headgear and rapid antler regeneration (Science 21 June 2019: Vol 365, Issue 6446)
反刍动物是一类特别的哺乳动物,与其他哺乳动物相比,几乎所有反刍动物在其头部都具有骨质角,而在不同的类群中,角的形态、组成结构以及生理性质存在差异(例如,长颈鹿科的角(ossicones)是表面有皮肤和毛发覆盖的骨质凸起;叉角羚科的角(pronghorn)和牛科的角(horn)都有分叉的角鞘,叉角羚科的角鞘每年脱落而且分叉,而牛科的角鞘不脱落不分叉;鹿科的角(antler)可以每年脱落和再生)。尽管差异巨大,它们仍共享一些特质,例如,相同的生长位置,两侧对称生长以及具有覆盖物的骨质核等。
本期Science杂志,西北工业大学邱强教授和西北工业大学暨中国科学院昆明动物研究所王文研究员团队,与西北农林科技大学姜雨教授团队联合攻关,对反刍动物角这一器官的遗传演化基础进行了深度解析。有角的反刍动物属于有角下目(pecorans),这一支系物种在大约20.8到23.3个百万年前发生了辐射进化。由于角形状及诸多特性存在较大差异,因此其起源问题一直存在争议。本研究中,依托“反刍动物基因组计划”获得的反刍动物广泛的系统发育关系(family-level)拓扑结构表明,角的发生应当为单次起源和后续的两次丢失事件(图 1),随后通过对三种无角的物种(獐子(Hydropotes inermis)和麝科两物种(Moschus chrysogaster和M. leucogaster))进行比较基因组分析,研究人员发现RXFP2基因的趋同假基因化可能是导致这一支系内物种角丢失角的关键因素。虽然对于起源问题以及造成丢失的问题进行了较为明确的解析,这一支系内物种角在短短~2.5个百万年内(20.8~23.3)就发生了如此多样性的分化,造成这种差异的关键原因仍未可知,这可能仍需要更精细以及丰富的数据来支持回答,而解决这一问题的关键点可能需要更加关注于非编码区的差异与改变。
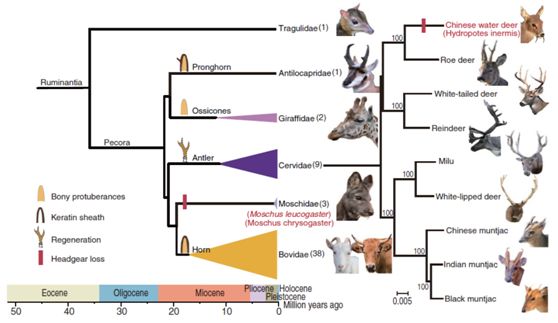
图1. 基于基因组大数据构建的系统发育关系表明反刍动物角的单次起源和两次丢失事件
通常,演化产生新的器官通常需要招募一些在其他组织和器官中表达得基因[1]。为了探究角的遗传和细胞学基础,研究人员收集并分析了袍子(代表鹿科)和绵羊(代表牛科)的221个转录组数据。通过对转录组数据进行比较,研究人员发现在羊角和鹿茸组织特异高表达的基因分别有624个和761个,并且这些基因主要从骨,皮肤,大脑以及睾丸组织中募集(图 2)。为了进一步明确角演化的遗传基础,研究人员通过分支点似然比检验等方法筛选出受到正选择的分别与成骨和诱导神经嵴细胞分化相关地两个关键基因,OTOP3和OLIG1,这一结果暗示它们对于有角下目物种角演化极为关键。在随后进行的保守序列分析和差异比较中,研究人员又发现了一系列与角演化相关的基因,而它们则大多与神经嵴细胞迁移及转化相关(图 2)。
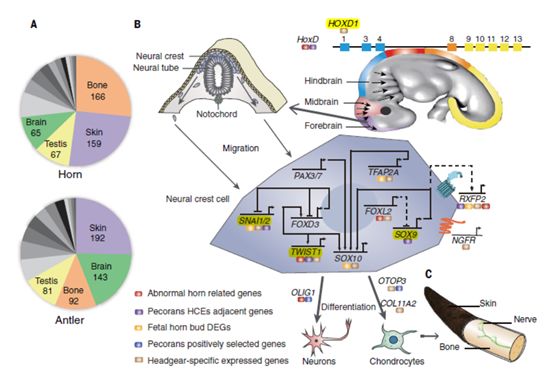
图2. 基于牛科(bovid)和鹿科(cervid)转录组数据发现反刍动物角起源的遗传基础
鹿茸是哺乳动物中唯一可完全再生的器官,是再生医学研究的优秀动物模型。鹿茸的再生是以干细胞为基础的发育过程,而这些干细胞可能起源自头盖骨的神经嵴细胞。同时,鹿茸生长速度极快,在生长阶段(由春到夏)平均每天可生长1.7厘米并且可以重达30千克[2]。这一过程中,细胞快速增殖的能力甚至超越了癌症细胞,因此研究人员对鹿茸生长通路表达基因与癌症表达通路的基因进行了比较,发现两者在细胞生长调节上具有一定的相似性:包括FOS、FAM83A和EGFR在内的一系列促进细胞快速增殖的基因在鹿茸中受到正选择,这与癌症细胞增殖类似(图3)。从比对结果上看来,尽管两者具有较高的相似性,但仍具有一定程度的差异性,从差异性入手,可能对于发掘癌细胞增殖但不受限制的原因具有一定启示。

图3. 癌细胞增殖通路相关基因参与鹿茸快速再生过程
快速增殖的相似通路似乎暗示了鹿类可能遭受更高强度的癌症威胁,不过有统计结果表明,鹿科物种患癌症的概率低于其他哺乳动物5倍。研究人员通过进一步分析,发现抑癌基因PML在鹿类中发生了特异性非同义突变并带有强烈的自然选择信号;与TP53(抑癌通路)相关的基因(MPL,NMT2,ISG15……)在鹿物种中也受到了强烈的正选择;同时,调控肿瘤微环境相关的基因ADAMTS18和DNA损伤响应相关的SLF1等基因也受到了自然选择(图 4)。
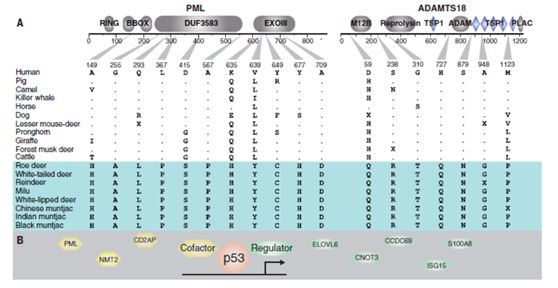
图4. 鹿科物种PML和ADAMTS18基因的特异性非同义突变,以及P53通路受到自然选择的基因
以上诸多信息表明,鹿科物种抑制癌症发生的一些基因经历了较强的自然选择,与大象通过增加抑癌关键基因TP53的拷贝数不同,鹿科物种“抑癌”通过调节TP53上下游基因实现以上结果,特别是与鹿茸快速再生生长和抑癌的相关基因,能够为人类再生医学和癌症相关研究提供重要借鉴。
作者简介:李家堂中国科学院成都生物研究所研究员,中国科学院青年创新促进会优秀会员,生命分会会长。主要从事两栖爬行动物系统学,进化遗传学与基因组学方面的研究工作。
参考文献
1. Griffith O W, Wagner G P. The placenta as a model for understanding the origin and evolution of vertebrate organs [J]. Nat Ecol Evol, 2017,1(4):0072.
2. Fennessy P F. Deer Antlers: Regeneration, function and evolution[J]. Journal of the Royal Society of New Zealand, 1984, 14(3):2.
来源:Science-AAAS ScienceAAAS
原文链接:http://mp.weixin.qq.com/s?__biz=MzI3NDY3NzQ2Mg==&mid=2247487333&idx=3&sn=9cac237a296f6b7afaca59328c1b11a4&chksm=eb1124dcdc66adca3b1c0a497fde1a9d990f1eb39722e355df3df57f750dd7c9806c416c7225&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
动物饲料

像鹿又像羊!它是全球最神秘的鹿科动物丨Science • 活物誌

美国新研究:植食动物灭绝风险高于肉食动物和杂食动物
动物学: 蚯蚓应对植物防御体系的办法
CURRENT ZOOLOGY(动物学报)影响因子2.181,继续稳定在Q1区期刊行列
物种保护:中国喀斯特地貌中的生物多样性
海龟是最长寿的海洋生物?这些动物表示不服
哺乳动物“变”温血动物之谜揭开
化石:气候变化是鱼龙灭绝的幕后黑手

动物所揭示外来动物和本土动物面对极端天气的响应差异