科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-06-24
来源:X一MOL资讯
药物影响下的代谢产物可直接反映出细胞所处微环境的变化,是药物研发,耐药性研究,等多种生物研究的重要手段。传统的代谢组学分析方法例如液相色谱或气象色谱与质谱联用,需要将样品均一化,从而失去了样品的重要的空间分布信息。质谱成像技术,由于其高灵敏性,高通量性,已应用在多种生物样品的代谢组学分析, 其中包括动植物组织和肿瘤样品。多细胞肿瘤球(简称肿瘤球)是一种3D 培养的肿瘤模型,与传统2D 细胞培养相比,它们可以更加真实地模拟肿瘤的3D结构及其内部微环境。通过结合机器学习来分析3D多细胞肿瘤球的质谱成像数据,一些重要的细胞代谢产物被发现分布在细胞球的不同位置,为药物筛选,抗药性研究提供了重要的信息。
近日,俄克拉荷马大学(University of Oklahoma)杨志柏团队提出了质谱成像(mass spectrometry imaging)结合机器学习(machine learning)的快速分析手段。首先通过对不同条件下抗癌药物处理过3D肿瘤球分别进行质谱成像数据采集,而后以一种重要的多变量数据分析方法(多元曲线分辨,multivariate curve resolution)为模型,区分出肿瘤球外层与内层部分。之后利用机器学习的方式寻找外层和内层部分代谢产物的分布规律,最后在此基础上建立和优化一系列的数学模型,用来预测整体肿瘤球的代谢产物空间分布规律(图1)。
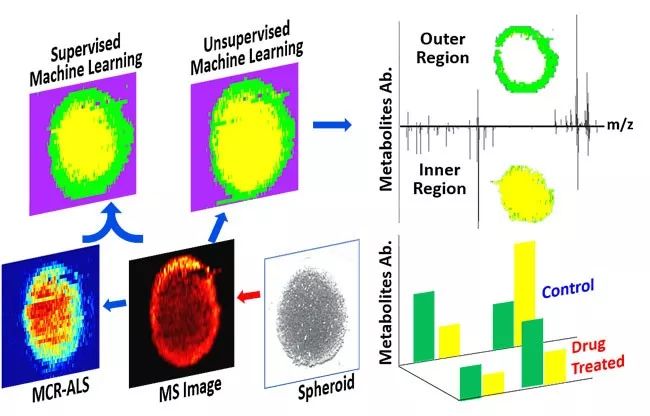
图1. 质谱成像与机器学习结合展示了其在代谢组学分析中的能力(X. Tian, G. Zhang, Z. Zhou, Z. Yang, Analytical Chemistry, 2019, 91, 5802-5809, Analytical Chemistry授权转载)。
实验结果表明,有监督机器学习(随机森林,random forest)模型经过训练后在极短的时间内就可以提供可靠的预测结果(图2)。为证明此模型的正确性,作者还将无监督学习机器学习(CLARA 和DBSCAN)算法来分析整体质谱成像数据,通过对比监督下与非监督下的机器学习,作者发现很高的图像重合度,由此证明机器学习的准确性。

图二:监督下的机器学习与非监督下的机器学习得出非常相似的图像结果。(X. Tian, G. Zhang, Z. Zhou, Z. Yang, Analytical Chemistry, 2019, 91, 5802-5809, Analytical Chemistry授权转载)
来源:X-molNews X一MOL资讯
原文链接:http://mp.weixin.qq.com/s?__biz=MzAwOTExNzg4Nw==&mid=2657614098&idx=3&sn=b2323bb1fa4355c222fe646f29fb78dd&chksm=80f7d0c2b78059d4df6f7c7c6c5785b13f4a43d4bb2fa8c412033545da915e01bb03bdd3bb91&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

机器学习诞生新型“研究员”

机器会学习,但它会遗忘吗?
机器学习算法能预测癫痫发作
DeepMind新论文:给侧面照片,AI给你脑补出正面


IBM研发出无监督式机器学习算法

机器学习与人工智能将协助人类破解宇宙中最大奥秘

机器学习在空间物理的应用

医疗数据非结构化广泛存在 医疗AI落地待解
机器学习揭示材料的“隐秩序”
机器学习软件可以预测细菌行为