科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-07-16
来源:BioArt
蛋白质工程(protein engineering)技术在优化抗体、生物酶、基因编辑工具酶等有重大作用。但若要大规模改造并筛选出最佳的蛋白质变体并非易事。现有的方法中,定点突变因工作量限制很难作高通量筛选;基因合成造价高昂且存在每千碱基有1至10个错误的风险;其他方法,比如同源重组,虽然可以同时构建不同的突变体,然而后期却很难实现高通量表征每一个突变体。
针对这个难题,2019年7月15日香港大学的黄兆麟(Alan S. L. Wong)博士课题组在Nature Methods上发表了一篇题为Combinatorial mutagenesis en masse optimizes the genome editing activities of SpCas9的文章,通过其命名为CombiSEAL的技术成功筛选出了一个SpCas9变体,Opti-SpCas9,使其在不损失活性及靶点选择范围的前提下降低脱靶效应(off-target effect)。

CombiSEAL技术首先将编码目标蛋白的DNA序列分切成若干部分,在每部分的序列中引入多个突变以形成子文库(sub-library),并在序列末端合成条码(barcode)以区分各个突变。其次,在第一部分子文库的基础上通过Type IIS限制性内切酶将第二部分子文库无缝插入到第一片段基因序列与barcode之间。以此类推,最终形成包含各个突变随机组合的蛋白突变体文库,每一个组合都由一个对应的组合条码标识(图1)。基于CombiSEAL合成的蛋白突变体文库不但满足了高通量筛选的要求,并且可以通过barcode对每一种可能的突变组合进行表征。
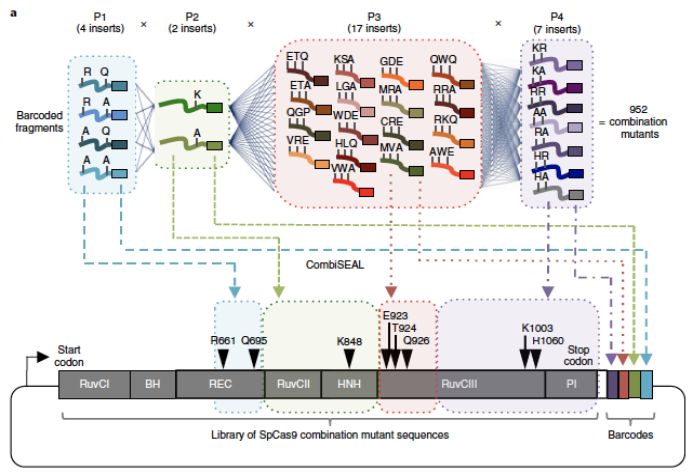
图1.CombiSEAL改造SpCas9示意图
黄博士团队利用CombiSEAL技术构建了包含948种突变组合的SpCas9文库。旨于筛选出比野生型SpCas9有更低脱靶效应且不限制其活性的突变体,作者合成了两对靶向红色荧光蛋白(RFP)的sgRNA,部分与靶向DNA序列完全互补(on-target),而另一部分包含一个或多个不匹配碱基(off-target)。利用分别包含这些sgRNA的报告细胞系,作者对SpCas9突变文库进行了高通量筛选。通过比较RFP敲除水平,作者筛选出Opti-SpCas9(图2)。
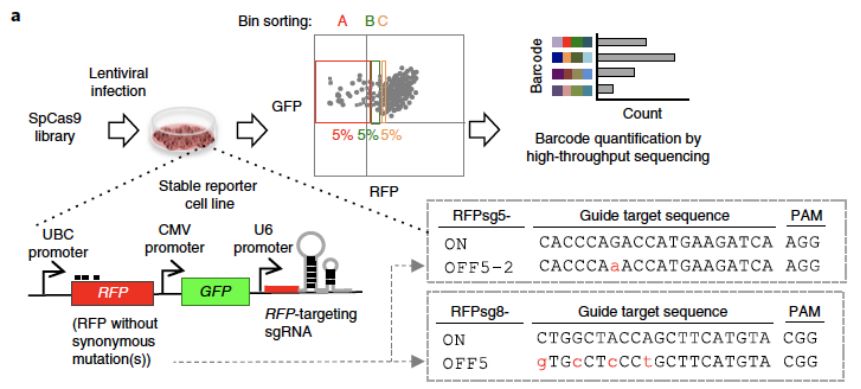
图2.高通量SpCas9突变体筛选策略
作者随后将Opti-SpCas9与当下Nature、Science等前沿期刊中报道的若干SpCas9突变体进行了比较与表征。首先,作者考察了sgRNA 5’-G对各个SpCas9突变体的活性影响。5’-G是广泛应用于sgRNA表达的启动子,U6,的转录起始信号。野生型SpCas9可以兼容5’端多一个G的sgRNA(即,GN20),而eSpCas9(1.1),SpCas9-HF1, HypaCas9,evoCas9 和Sniper-Cas9等突变体却不能与之兼容或活性较低,从而限制了这些突变体的应用。然而作者发现,Opti-SpCas9的活性并不受其影响,与野生型无异。同时,当sgRNA是以5’-G起始时(即,GN19),虽然Opti-SpCas9,eSpCas9(1.1)和HypaCas9均与野生型活性相差无几,但是GN19这类sgRNA却大大限制了CRISPR-Cas9的靶点选择范围。此外,作者还考察了另一项决定Cas9临床应用的指标,脱靶效应。研究发现Opti-SpCas9的脱靶效应显著低于野生型Cas9,并与上述诸多突变体保真性相当。
综上,Opti-Cas9在不损失野生型SpCas9的活性及靶点选择范围的同时大幅提高了其保真性,为后继实验提供了一个新的选择。黄博士课题组通过CombiSEAL技术成功地筛选出了Opti-SpCas9,充分地展示了该技术在蛋白质工程中的潜力,为以后改造其它蛋白质的工作提供了一个新颖高效的平台。
以下是黄博士早前于Nature Biotechnology及PNAS等发表开发其他高通量筛选平台(CombiGEM and CombiGEM-CRISPR)的相关文章:
1 Wong, A.S.L., Choi, G.C.G., Cheng, A.A., Purcell, O. & Lu, T.K. Massively parallel high-order combinatorial genetics in human cells. Nature Biotechnology 33, 952-961 (2015).
2 Wong, A.S.L. et al. Multiplexed barcoded CRISPR-Cas9 screening enabled by CombiGEM. Proc Natl Acad Sci U S A 113, 2544-2549 (2016).
3 Wong, A.S.L., Choi, G.C.G. & Lu, T.K.Deciphering Combinatorial Genetics. Annual Review of Genetics 50, 515-538 (2016).
原文链接:
https://www.nature.com/articles/s41592-019-0473-0
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652472589&idx=3&sn=58d1e0433e89a4e05091d64af3989228&chksm=84e216b9b3959faf753a987834370f0c42ea81070bda7c071bba4c78896bc34c742f866fed3a&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn