科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-07-28
来源:BioArt
原标题:Nat Biotech丨效率更高、兼容性更好,David Liu研发噬菌体辅助持续演化的单碱基基因编辑器
基因组编辑,是生物医学科学领域的一项重大革命,为很多人类遗传病的治疗提供了无限可能。一个理想的基因编辑工具应该可以高效率的编辑任何所需位点、具有高度特异性并几乎不产生其他副产物,当然,这种理想的试剂还没有被开发出来,而且不太可能存在于自然界中,因为自然发生的基因组编辑蛋白的进化只是为了实现部分相关的功能,例如调节基因表达或保护自身免受病毒感染。因此,研究人员认识到开发新的工具来增加基因组编辑的范围和有效性、特别是在真核细胞和人类疾病动物模型中的应用的重要性,近些年的努力也使这一目标在相对较短的时间内取得了显著进展。
从早期的基因编辑技术——锌指核酸酶(Zinc-finger nucleases,ZFNs)技术和转录激活因子样效应物核酸酶(Transcription activator-like effector nucleases,TALENs)技术,到具有跨时代意义的CRISPR/Cas9系统的发现,2016年4月,哈佛大学生物化学家 David Liu 实验室又率先在Nature杂志上报道了一种新的基因编辑工具——单碱基编辑系统(base editors, BEs),它以一种RNA程序化方式引入点突变,而不依赖于同源重组修复或双链DNA断裂【1】。如果说CRISPR/Cas9技术改变的是“字符串”,那么单碱基编辑技术更改的就是一个个精确的字符。
单碱基编辑技术利用一个催化受损的Cas9在特定的基因组位点打开一个单链DNA环,在这个区域内的编辑窗内(通常为5个核苷酸)的碱基被一个融合的碱基修饰酶所修饰,实现碱基转换(图1)。目前的单碱基编辑技术包括两种——嘧啶碱基编辑技术(CBEs, Cytidine base editors),可将C/G转换成T/A,以及嘌呤碱基编辑技术(ABEs, Adenine base editors),将A/T转换成G/C。
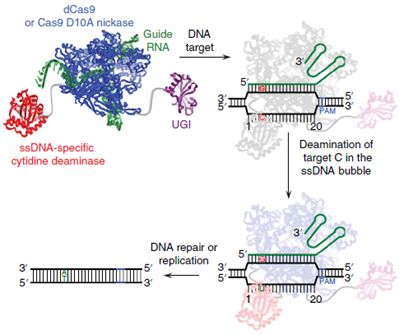
图1 CBE工作示意图
与利用程序化核酸酶产生双链DNA断裂从而引起同源重组修复的基因编辑方式相比,单碱基编辑技术的效率更高,而且产生意想不到的插入缺失、易位或其他染色质重排的几率变小。但是其局限性也不容忽视,以CBE为例,其缺点主要体现在以下几个方面:1.脱靶效应;2.适用范围有限,要求NGG PAM序列与目标C有一个特定距离;3.识别位点特异性有限,编辑窗内的C都有可能被编辑【2】。因此,改造CBE以发挥其最大优势成为目前的研究趋势。
研究发现,通过改变CBE的融合蛋白中的胞嘧啶脱氨酶可以有效增加编辑效率,例如基于CDA1脱氨酶的CBE在HEK3细胞中编辑GC3的效率比第一代基于APOBEC1脱氨酶的CBE效率明显增加。CBE的这种可变性使得其可以不考虑靶序列背景而根据应用需要进行改造,而且,一个可以定制碱基编辑器性能的通用平台将有利于理想的适用于特定应用的编辑器的开发,这些编辑器在效率、序列兼容性和特异性方面可能存在很大差异。
基于CBE的复杂程度以及现阶段单碱基编辑器开发的需求,2019年7月22日,来自美国哈佛大学的David Liu课题组在Nature Biotechnology上在线发表题为Continuous evolution of base editors with expandedtarget compatibility and improved activity 的文章,开发了一个噬菌体辅助持续演化的单碱基编辑系统(BE-PACE),可以根据应用需求,在快速进化CBE的同时,大大提高CBE的编辑效率和靶序列兼容性。

2011年4月,David Liu实验室在Nature上报道了一种能在实验室内快速进化蛋白质的方法,他们利用由噬菌体辅助进行的连续演化体系,这个体系能使基因编码的分子连续定向进化,而这些分子可以与大肠杆菌中的蛋白质生产相联系,被称为噬菌体辅助持续演化(PACE)。在PACE过程中,进化的基因通过一个改良的噬菌体M13生命周期在宿主细胞中传递,其方式取决于所关注的活性,而且一天之内就能进行几十轮的演化,无需人为干预【3】(图2)。
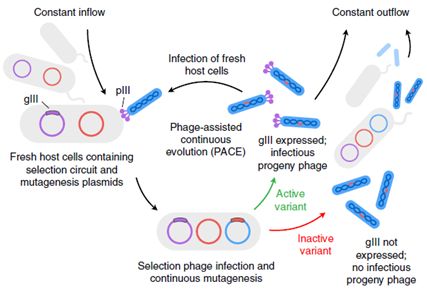
图2 PACE模式图
在本研究中,研究人员期望将胞嘧啶单碱基编辑活性与PACE偶联,在大肠杆菌宿主细胞中产生遗传回路,即BE-PACE系统,在这个系统中,遗传回路必须由一个单一的碱基变化激活,对少量的编辑事件作出响应,并且足够快地启动以支持噬菌体在连续稀释下的繁殖。为了满足这些要求,研究者设计了一个基因回路,在转录模板链上发生胞嘧啶脱氨基作用以恢复蛋白质的失活突变,进一步优化后,只有一部分碱基编辑器由噬菌体编码,其余的由宿主细胞质粒提供,每一半融合到一个快速剪接的分裂内含肽中,使得全长碱基编辑器蛋白在感染后形成(图3),进而使得噬菌体的繁殖和选择性都大大提高,并可用于基于荧光素酶报告系统的表达评估细胞内脱氨酶的动力学。
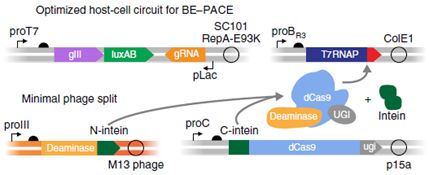
图3 优化的BE-PACE系统
随后,研究者们利用BE-PACE系统,克服传统CBE的靶序列背景限制的缺陷,演化产生了三种类型的胞嘧啶单碱基编辑器,在体外荧光素酶报告系统和哺乳动物细胞中均证实,演化的CBE系统在一定程度上增加了序列背景兼容性和编辑活性。
首先,针对野生型APOBEC1脱氨酶的序列背景限制(只支持TC靶序列而不支持GC靶序列的识别),演化的evoAPOBEC1-BE4max与传统的APOBEC1相比,在GC背景下编辑胞嘧啶的效率提高26倍,并且维持了在其他序列背景下的编辑效率。而另一个演化的脱氨酶evoEFRNY-BE4max,在所有检测的序列背景中都表现出较高的编辑效率,并且它比APOBEC1小29%,使得其可能对受有效载荷大小限制的病毒传递应用有特别作用。与此同时实验证实,与野生型BE4max相比,上述两种演化的CBE的编辑窗宽度和脱靶效应均未有太大改变。而本研究中的另一个演化的CBE——evoCDA1-BE4max,虽然对于较难编辑的靶位点有较高的编辑效率,但是考虑到该CBE表现出了较高的插入缺失水平和脱靶效应,作者提示我们,要根据其特点仔细筛选其应用,在脱靶效应和周围的意外编辑不被考虑而只关注高效率的情况下可以选择evoCDA1-BE4max,例如单碱基基因编辑的高通量筛选。
最后,为了评估PACE演化的CBE在实际应用中的潜力,本文研究人员在原代细胞和细胞系中,用演化的CBE编辑三种疾病相关突变位点,分别为引起耳聋的TMC1突变、高阿尔兹海默症风险相关的APOE4位点和Wolfram综合征1的WFS1突变,结果显示演化的CBE的编辑效率显著提高,从而突出显示了evoAPOBEC1-BE4max和evoEFRNY-BE4max改进的GC编辑效率和窄窗口,也强调了最大限度地提高找到符合特定应用要求的CBE的可能性。
综上所述,本研究应用PACE平台,开发了一个新的单碱基基因编辑优化系统,使得单碱基基因编辑的效率更高、兼容性更好,而本文开发的优化CBE的方法——BE-PACE平台可将继续用于丰富单碱基基因编辑的性能,扩大碱基编辑的应用范围,加深人们对碱基编辑结果决定因素的影响的认识,为单碱基基因编辑系统的优化提供强大工具。
原文链接:
https://doi.org/10.1038/s41587-019-0193-0
参考文献
1. Komor AC,Kim YB, Packer MS, Zuris JA, Liu DR. Programmable editing of a target base ingenomic DNA without double-stranded DNA cleavage. Nature. 2016; 533:420–424.
2.Alexis C.Komor, Ahmed H. Badran, David R. Liu. CRISPR-based technologies for themanipulation of eukaryotic genomes. Cell. 2017 January 12; 168(1-2): 20–36.
3. Kevin M.Esvelt, Jacob C. Carlson, David R. Liu. A System for the Continuous DirectedEvolution of Biomolecules. Nature. 2011 April 28; 472(7344): 499–503.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652473260&idx=2&sn=655bd1d290a41dc54eb9a6eaa7396e3d&chksm=84e21418b3959d0ec91fbc6cff577a72d50f2c760db0e56d392b079bc3e27be0feba2d1d1fdb&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn