科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-08-12
来源:BioArt
基因组的三维结构是由拓扑相关结构域(TAD)基本单元构成,而这些TAD又被称之为边界的序列分隔开,TAD和边界的形成过程中,锌指转录因子CTCF以及多亚基复合体黏连蛋白Cohesin起很关键作用(图1)。
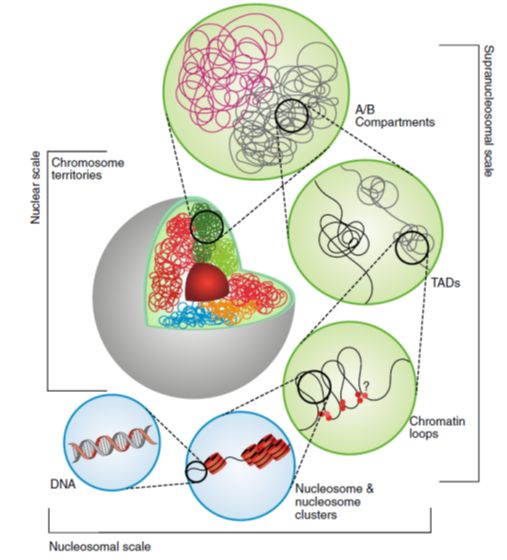
图1. 基因组的三维立体结构【1】
尽管已有研究报道基因组TAD的重组装会引起基因的异常表达和疾病,但条件性全基因组水平敲除CTCF并不显著影响基因的表达水平【2】。2019年7月28日,来自德国马普分子遗传学研究所的Daniel M. Ibrahim课题组在Nature Genetics杂志上发表文章Functional disp of the Sox9–Kcnj2 locus identifies nonessential and instructiveroles of TAD architecture。研究者们聚焦在Sox9-Kcnj2基因座,研究该基因座内不同序列元件(边界序列和CTCF结合序列)对TAD结构重构的影响以及对肢端发育的影响。

Sox9-Kcnj2是肢端发育的重要基因,在位置上紧密连锁。作者首先利用capture Hi-C(cHi-C)技术【3】鉴定了Sox9-Kcnj2 基因座的拓扑相关结构域(TAD)及它们之间的边界,同时利用ChiP-seq鉴定了CTCF的结合序列,值得注意的是这些CTCF结合序列有方向性。ATAC-seq和H3K27ac 的ChiP-seq预测了52个可能的增强子序列(图2)。
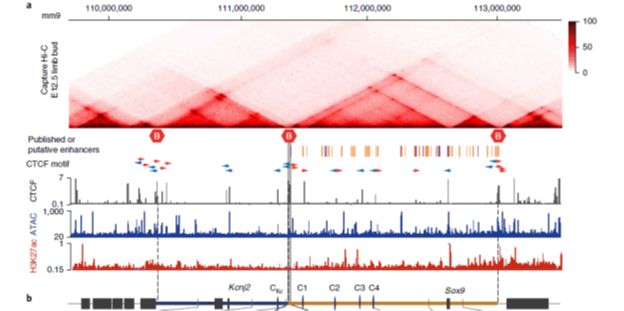
图2. Sox9-Kcnj2 基因座的拓扑相关结构域以及对应的CTCF/H3K27ac ChiP-seq ATAC-seq 峰值图:a图中红色深度显示不同序列间的相互作用强度。垂直的虚线分隔开不同的TAD(红色三角区域),六边形红色框中间字母B标记边界(boundaries)。cHi-C下面的竖线指示预测的增强子。b图: 基因由黑框显示,蓝色横线区域指示KcnjTAD, 橘色横线区域指示Sox9 TAD;灰色区域是这两个TAD中间的边界区。
接下来作者敲除了Sox9 和Kcnj2 TAD之间的约8Kb长边界序列(ΔBor),以及同时敲除边界和TAD区域内的CTCF结合位点(ΔBorC1–4),结果发现单独敲除边界序列并没有导致TAD结构的较大改变,仅稍微增强了两个TAD的接触。但同时敲除边界序列和CTCF结合位点,则大大增强了这两个TAD的融合。在此ΔBorC1–4敲除基础上同时敲除Kcnj2 TAD 中的主要CTCF结合位点进一步促进了融合,甚至这两个TAD已发生重构。这些数据说明TAD结构的建立和维持不仅需要边界序列,还依赖于边界和CTCF结合位点的协同作用。
那么TAD融合和内部重构对基因表达以及肢端发育的影响如何呢?它们利用qPCR 检测这两个基因的表达量以及Wish检测E12.5 时期在肢端的表达范围,结果发现引起TAD融合的敲除(ΔBorC1–4、ΔCTCF)并没有显著影响Sox9的表达量,但使得Kcnj2的表达量略微上升并且表达范围呈现类似Sox9的表达谱(图3),但并没有引起表性变化。
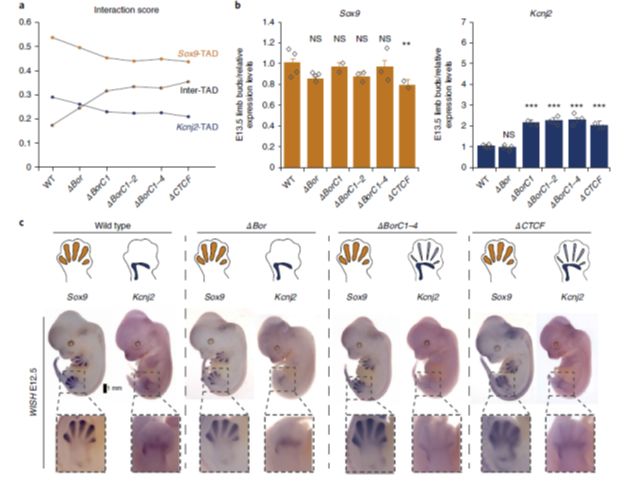
图3. TAD融合以及CTCF结合序列的敲除对基因表达和表型的影响。
因为CTCF对结合序列有方向性,那么翻转CTCF的结合序列会造成什么影响呢?作者进一步翻转了TAD内部主要的CTCF位点C1-4,发现对TAD结构影响不大,但连同边界区域一同翻转(InvC)则导致了TAD的重构,Kcnj 和翻转区域之间形成更大的TAD,sox9 被独立出去形成小的TAD。在Sox9基因之前加入一个边界序列也将Sox9独立成新的TAD。在(InvC)基础上删除边界序列(InvCΔBor)导致TAD的融合,和Sox9 TAD的重新形成。作者进一步研究了翻转、重置等这些突变对基因表达和表性的影响。结果显示:InvC 下调了Sox 9的表达量,InvC and InvCΔBor这两种突变则导致了Kcnj2的异位表达,获得了类Sox9的表达模式,并产生了突变表型。
那么怎么理解TAD融合和分离对基因表达的影响呢?作者认为TAD结构的建立和维持与启动子和增强子的相互作用是两个层面,基因的表达更依赖于增强子和启动子的相互作用。TAD的融合对基因的表达影响并不像想象中的那么显著,可能由于除CTCF之外Cohesion仍能维持内部的TAD,作者同时发现TAD内部CTCF结合位点对TAD结构维持的重要性;另一层面,TAD分离和重构则会导致启动子和增强子的分离,这就解释了为什么CTCF位点的翻转和边界序列的重置会明显影响基因表达量和表达范围。
总之,这篇文章在TAD结构调节基因表达的强度和准确性上给我们新的启示。
原文链接:
https://doi.org/10.1038/s41588-019-0466-z
参考文献
1. Dogan, E.S.,and Liu, C. Three-dimensional chromatin packing and positioning of plantgenomes. Nat Plants 4, 521-529.(2018)
2. Nora, E. P. etal. Targeted degradation of CTCF decouples local insulation of chromosomedomains from genomic compartmentalization. Cell 169, 930–944.e22 (2017)
3. Franke, M. etal. Formation of new chromatin domains determines pathogenicity of genomicduplications. Nature 538, 265–269 (2016).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652473831&idx=4&sn=499579332143d7445421d11c37dcfd3c&chksm=84e21a53b39593453405022c3f27fc5510ebbaf588d5978782a289022b8d02dfab33d4d0f26f&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

2018国内重磅科技新闻盘点

中国科技新闻学会倡议书

2020年国内十大科技新闻
苹果WWDC 2017干货亮点全在这里
神经科学:协同工作的大脑

【前沿】一周科技新闻

中国科技新闻学会祝您元旦快乐!
动物学: 侦察蚁能够引导搬运食物的同窝蚂蚁回家

【纵览】一周科技新闻
化学:以分子为基础的隐形墨水