科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-08-18
来源:中国高科技
原标题:植物表观遗传学前沿领域取得重要进展,朱健康院士团队揭示植物DNA主动去甲基化的分子机制
2019年7月30日,中国科学院上海植物逆境生物学研究中心朱健康研究组在PNAS在线发表了题为"HistoneAcetylation Recruits the SWR1 Complex to Regulate Active DNA Demethylation in Arabidopsis"的研究论文,该研究发现了植物DNA主动去甲基化的完整调控途径,详细阐述了植物DNA主动去甲基化的最新机制。该研究还揭示了拟南芥SWR1复合体通过识别常染色(组蛋白乙酰化)和异染色(DNA甲基化)标记物而被招募到染色质上的靶向机制。

DNA甲基化修饰发生于胞嘧啶的第五位碳原子上,是一种很重要的、保守的表观遗传学标记。DNA甲基化与DNA去甲基化决定了生物体内的甲基化水平和图谱。在植物中,DNA主动去甲基是一个在去甲基酶的作用下甲基基团被移去的过程(Zhu, 2009)。朱健康教授实验室于2002年报道了首个生物体内的去甲基化酶ROS1(Gong et al., 2002);随后的研究表明IDM1蛋白介导了ROS1的主动去甲基化过程 (Qian et al., 2012)。然而,尽管进行了深入的研究,DNA去甲基酶是如何被招募到基因组上的靶位点进行去甲基化的详细机制仍然不清楚。
为了鉴定新的DNA去甲基化因子,研究人员通过正向遗传筛选体系以寻找ROS1介导的DNA去甲基化和抗沉默所需的细胞因子。研究人员鉴定到ARP6和PIE1,两个染色质重塑SWR1复合体中的组分蛋白,以及一些已知的DNA去甲基化因子,如ROS1、IDM1和MBD7。研究结果表明,含bromo结构域的蛋白NPX1和ATMBD9能够识别由IDM1建立的乙酰化组蛋白标记,乙酰化的组蛋白标记物将SWR1复合体招募到染色质上去沉积H2A.Z;最终,在这些特定基因组DNA区域中,H2A.Z和ROS1直接相互作用从而招募ROS1去执行DNA主动去甲基化,以防止高甲基化和/或DNA甲基化的传播(图1)。因此,该研究确定了由IDM复合物起始的植物DNA主动去甲基的完整调控途径。
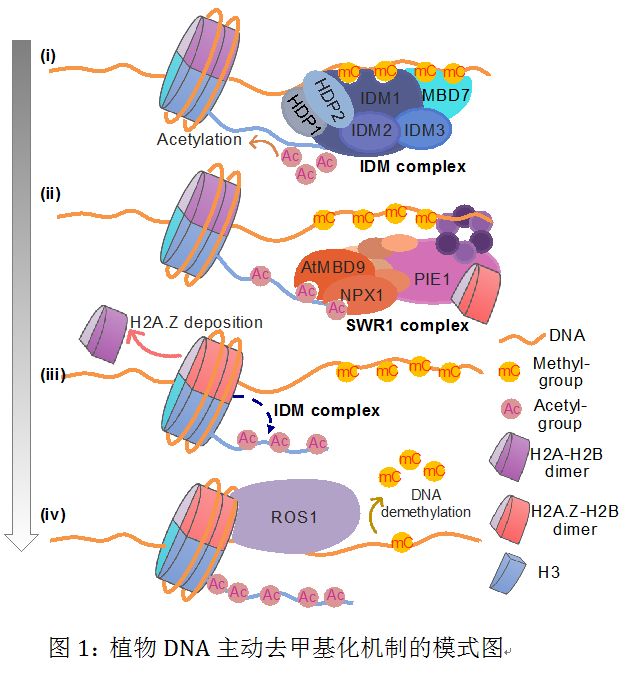
研究还发现甲基DNA结合蛋白ATMBD9和SCBDF1同系物NPX1通过识别特定染色质标记在DNA主动去甲基化过程中具有冗余功能。ATMBD9作为拟南芥SWR1复合物的一个新成分的发现,通过识别常染色质(组蛋白乙酰化)和异染色质(DNA甲基化)标记 (图1),提供了对SWR1复合物到特定染色质区域的募集机制的重要认识。
困扰生物学研究中的一个基本问题,就是活性的常染色质和沉默异染色质在不同基因组区域是如何建立和维持的。两个最为广泛研究的染色质标记,DNA甲基化和H2A.Z,在植物和哺乳动物中都是相互拮抗的(Zilberman et al., 2008)。然而,这一有趣的内在机制尚未得到解决。该研究结果揭示了在许多基因组区域中H2A.Z(常染色质标记)和DNA甲基化(异染色质标记)相互排斥的机制;此外研究结果还明确了DNA甲基化修饰与H2A.Z的协同作用,发现了二者在调控DNA去甲基化过程中在染色质上的共存规律。
DNA甲基化图谱对于生物体生长发育、癌症发生和许多其他疾病,甚至对于人类衰老都是重要的,已经发现基因组上的很多位点的DNA甲基化水平的变化与人类的衰老完全相关。DNA甲基化图谱如何改变以及基因表达的调控是受到广泛关注的。从应用科学的角度来看,DNA去甲基化机制在保持转基因生物中的转基因活性进而改善生长、耐受环境变化、或预防疾病等过程中有十分重要的指导价值。
该研究由上海植物逆境生物学研究中心联合美国普渡大学、浙江大学、浙江省农科院等国内外研究单位共同完成,朱健康教授为该研究论文的通讯作者,聂文锋博士和雷明光研究员是该文章的共同第一作者,朱健康教授和聂文锋博士设计了该研究工作,浙江大学园艺系喻景权教授和奥地利科学院Fred Berger教授等参与了该研究工作。该工作得到了中国科学院、中国国家留学基金委员会等单位的经费资助。
原文链接:
https://www.pnas.org/content/early/2019/07/29/1906023116
参考文献:
Gong, Z., Morales-Ruiz, T., Ariza, R.R., Roldan-Arjona, T., David, L., andZhu, J.K. (2002). ROS1, a repressor of transcriptional gene silencing in Arabidopsis,encodes a DNA glycosylase/lyase. Cell 111,803-814.
Qian,W., Miki, D., Zhang, H., Liu, Y., Zhang, X., Tang, K., Kan, Y., La, H., Li, X.,Li, S., et al. (2012). A histoneacetyltransferase regulates active DNA demethylation in Arabidopsis. Science 336, 1445-1448.
Zhu,J.K. (2009). Active DNA demethylation mediated by DNA glycosylases. Annu RevGenet 43, 143-166.
Zilberman,D., Coleman-Derr, D., Ballinger, T., and Henikoff, S. (2008). Histone H2A.Z andDNA methylation are mutually antagonistic chromatin marks. Nature 456, 125-129.
来源:cas-hitech 中国高科技
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MDczMTAzMA==&mid=2650047022&idx=2&sn=9a870910f31ccb7ffb9f646ff7228efe&chksm=87387657b04fff41ae1bf691c398c30157d1de33ff51e7ba960cea6be83017be9115791f8efd&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn