科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-09-04
来源:BioArt
原标题:NCB:何爱彬课题组开发新一代高灵敏度单细胞技术-sc-itChIP,整合单细胞转录组解析细胞命运决定的表观控制规律
染色质的表观修饰状态与基因表达和细胞命运决定密切相关。目前单细胞表观组学的研究,主要集中在DNA的化学修饰,染色质的开放程度以及3D构象等【1-3】。值得注意的是,染色体上组蛋白的各种共价修饰影响着顺式作用元件的功能,DNA结合蛋白的结合决定了基因的特异性表达。因此,在单细胞水平捕获DNA互作蛋白的结合信息尤为重要,但这一技术仍面临诸多挑战。
如上所述,蛋白质和DNA相互作用的染色质免疫共沉淀技术(ChIP-seq)是一种研究表观转录调控的重要方法。尽管核酸酶MNase的引入促进了STAR-ChIP【4】, μChIP-Seq【5】和CUT&RUN【6】等少量细胞ChIP-seq技术的实现和优化,然而ChIP-seq技术在单细胞水平的研究一直进展缓慢。目前报道的单细胞ChIP-seq技术一方面需要特殊的微流控【7】或者Takara ICELL8【8】装置,另一方面得到的单细胞测序数据质量欠佳【9】(DNA片段基因组比对率低,单细胞捕获的DNA片段少),这些因素极大地限制了单细胞ChIP-seq技术的广泛应用。所以,开发一种易操作,高质量,具有普适性的单细胞ChIP-seq技术,是单细胞表观基因组领域的科学家期盼实现的重大突破。
2019年9月3日,北京大学何爱彬课题组在Nature Cell Biology在线发表了题为Profiling chromatin state by single-cell itChIP-seq 的文章,报道了一种新的具有普适性,易操作的单细胞ChIP-seq技术,并将这一技术命名为itChIP(simultaneous indexing and tagmentation-basedChIP-seq)。不同于该课题组开发的CoBATCH单细胞ChIP-seq技术(Molecular Cell | 何爱彬组开发高通量、普适性单细胞ChIP-seq技术——CoBATCH)【10】,单细胞itChIP-seq的一大亮点是,这项技术不仅适用于上千个单细胞的捕获,同时也可用于捕获起始量只有几十个单细胞的样品。这为研究稀少细胞样品例如植入前胚胎等的表观调控异质性提供了新的技术手段。

在itChIP实验中,研究人员采用Tn5转座酶切割DNA,切割完的DNA直接带上可供PCR扩增的DNA序列。巧妙之处在于,研究人员用甲醛交联样品,然后在高温下用SDS处理细胞,这样能使全基因组染色体变得松散,同时不会影响蛋白质与DNA的结合。在这种处理下,Tn5能均匀地切割染色体,而且不会产生对开放区域的偏好性(图1和图2)。

图1 itChIP实验流程图
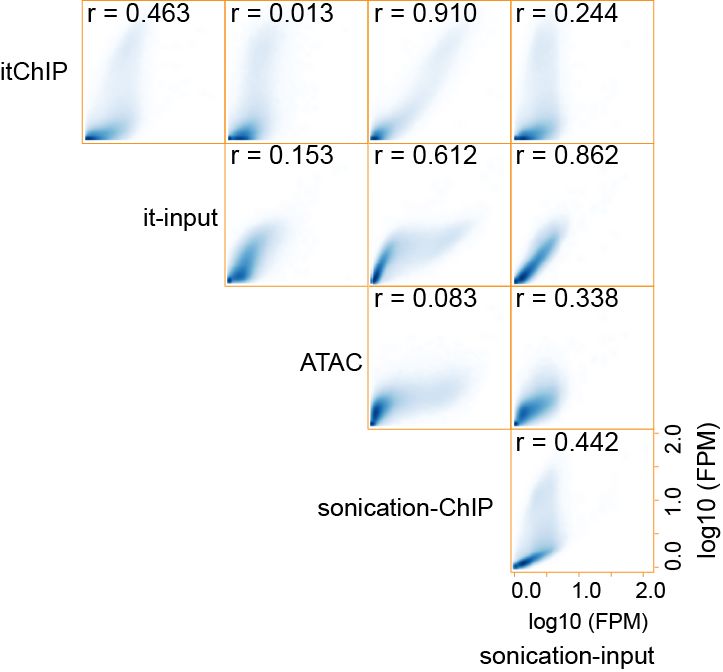
图2 全基因组水平分析验证itChIP实验中Tn5均匀切割染色体
研究人员首先用小鼠胚胎干细胞证明了少量细胞itChIP-seq的可行性。分别对不同细胞量级(100, 500, 10K)的细胞进行多种组蛋白修饰(H3K4me3和H3K27me3)和DNA结合蛋白(P300、RNA PolII和EZH2)的itChIP-seq,结果表明,itChIP比先前报道的少量细胞ChIP-seq方法具有更高的精确性、检出率和信噪比。
研究人员进一步拓展,巧妙地开发了一种新的单细胞ChIP-seq的方法,命名为single cell itChIP-seq。在单细胞itChIP实验中,研究人员用带有不同标签序列组合的 Tn5转座酶切割单细胞染色体,将切割完的单个细胞合并,然后裂解细胞核,释放带有不同标签序列的染色体片段,进行免疫共沉淀。(图3)。利用这一单细胞ChIP-seq方法,研究人员能在每一个单细胞中捕获平均9,000个DNA片段,并且基因组比对率能达到94%。这一单细胞方法的实验不需要依靠任何特殊设备。
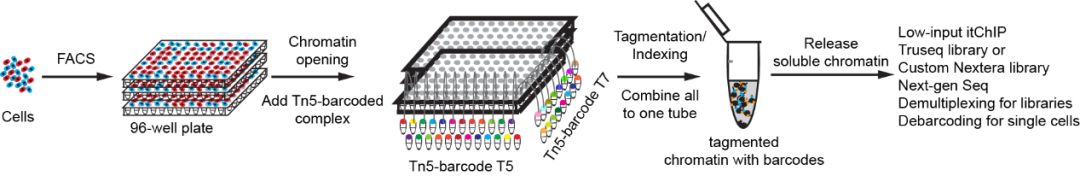
图3 单细胞itChIP实验流程图
值得一提的是,研究人员开发了一种新的单细胞Mosaic Truseq两步PCR的文库制备方法。Mosaic Truseq文库可以使用Illumina标准的测序方法和平台进行测序。与高成本特殊定制的单细胞测序方法相比,一方面,Mosaic Truseq文库制备方法大大降低了测序成本;另一方面,实验人员不需要精通专业的测序知识,即可进行大规模的单细胞测序。
在胚胎发育的早期,细胞退出全能性并向内、中、外三个胚层分化过程中,原始态多能性相关增强子活性逐渐减弱。然而,具有各胚层命运的细胞出现的时间和调控机制无法通过检测大量细胞的增强子活性获得。通过单细胞itChIP对近2,000个外胚层样细胞(epiblast-like cells, EpiLCs)的H3K27ac(一种反映增强子活性的组蛋白修饰)的异质性进行分析,研究者清楚地区分出处于不同分化状态的细胞群体,并揭示在发育过程中,外胚层和内胚层的命运可能优先被决定,而中胚层分化最后发生。
为了进一步证明单细胞itChIP适用于细胞数量很少的胚胎组织样品,研究人员对胚胎期第7.75和第8.25的NKX2-5谱系的心脏细胞同时进行了单细胞的RNA-seq和单细胞H3K27ac itChIP。通过对这两个不同维度的单细胞数据进行整合分析,研究人员发现了,在心脏干细胞向心肌细胞和内皮细胞两个不同谱系分化过程中,增强子表现出谱系特异性的激活和关闭。有趣的是,在这两个谱系分化过程中,虽然基因表达呈现出平滑的逐渐上调和下调(线性变化)的渐变趋势,但是增强子却表现出骤变式(指数级变化)的激活或者失活,这一现象说明增强子比其调控的基因表现出更强的时空特异性。综上,单细胞itChIP不仅可用于解析通量较高的细胞群体间的表观异质性,也可以用于鉴定低通量如特定分化谱系的胚胎样品或者早期胚胎样品细胞命运决定的表观调控机制。
此外,作者还建议将不同处理组样品利用barcoded Tn5切割后,混合在一起进行免疫共沉淀,这样可以消除不同组间由于实验引入的误差,同时节省大量的实验操作时间和成本。总之,这项全新的单细胞itChIP技术,将为研究重要的生物学过程,尤其是植入前胚胎细胞命运的分化和调控机制,带来新的曙光。
值得注意的是,该课题组近期(详见BioArt报道:Molecular Cell | 何爱彬组开发高通量、普适性单细胞ChIP-seq技术——CoBATCH)报道了另外一种截然不同的高通量的单细胞ChIP-seq技术—CoBATCH。本文介绍的sc-itChIP-seq方法,可适用于起始细胞数极少的珍贵样品(~100)的单细胞解析。
专家点评
颉伟(清华大学)
单细胞RNA-seq的广泛应用极大的推动了组织特异性研究,新的细胞类型的发现,以及基因表达时空性的模式研究。国内汤富酬、郭国骥等课题组在相关技术开发和应用上做出了出色的贡献。而另外一个重要的生物学问题是在单细胞水平这些基因表达是如何调控和实现的呢?许多单细胞表观组学技术比如单细胞甲基化scRRBS (Guo et al., 2013), scBS-seq (Smallwood et al., 2014), 单细胞染色质开放性scATAC-seq(Cusanovich et al., 2015; Buenrostro et al., 2015), scDNase-seq (Jin et al.,2015),单细胞三维基因组sciHiC-seq (Nagano et al., 2017)等的开发为我们了解细胞命运的调控和异质性提供了很多信息。这些技术在各个领域的应用已经逐步开始显现。另一方面,组蛋白修饰和转录因子也在基因调控中起到了至关重要的作用,但是能够在单细胞水平有效检测蛋白与DNA相互作用的技术仍然比较有限,已经报道的方法大部分还处于应用初期,尚未开始广泛应用。
北京大学何爱彬实验室在最新发表于Nature Cell Biology的文章中报道了一种新的能用于少量细胞甚至单细胞的ChIP-seq方法,即itChIP。研究人员将单个细胞用甲醛交联并用SDS处理使染色质松散之后,用带不同标签的Tn5转座酶来打断基因组,这个过程中通过优化来保证染色质打断的平均性。之后再将单个细胞合并到一起做后续的免疫共沉淀。这一方法使得研究人员最终能够拿到平均每个细胞9,000 条测序片段的信息。研究人员对约200个小鼠胚胎发育的第7.75和第8.25天的心脏细胞进行了H3K27ac itChIP-seq。结合约300个单细胞scRNA-seq的数据,研究人员找到了一些心脏前体细胞命运决定中潜在的谱系特异性增强子。近年来Steve Henikoff和Keji Zhao等实验室也开发了基于Tn5的CUT&RUN(Peter et al., 2017),CUT&TAG(Hatice et al., 2019), scChIC-seq(Wai et al., 2019) 等方法。与这些方法相比,itChIP-seq的mapping效率要更高,另外,itChIP-seq的起始细胞可以低至100个,而一些其他的单细胞技术的实验起始步骤仍然需要大量细胞。我们期待itChIP-seq和其他高灵敏染色质分析技术能够大大推动在单细胞水平研究基因表达的研究。
另附颉伟研究员对上周何爱彬组Molecular Cell文章的点评
动物发育过程中伴随着细胞的分裂和分化,最终发育成为不同的组织并构成器官。由于发育过程复杂性和细胞的异质性,在单细胞层面上理解不同细胞对于发育的贡献是生物学的基本问题之一。最近的许多工作通过单细胞RNA-seq的方法解析了多种物种关键发育时期的不同类群细胞的转录状态和发育轨迹。进一步解析不同细胞的转录调控则需要鉴定基因组中不同的调控序列(如启动子、增强子和绝缘子等)的表观遗传学特征,因此需要建立单细胞表观组学的分析方法。此前,单细胞DNA甲基化测序、高级结构解析、开放染色质鉴定等方法已经建立了起来,但蛋白(包括组蛋白、转录因子或其他染色质调控因子)和染色质互作的鉴定方法开发还相对落后。因此,建立单细胞水平的组蛋白修饰和转录因子结合的鉴定方法是非常重要的。
不同于经典的研究组蛋白修饰或转录因子结合位点的方法ChIP-seq,最近,包括Henikoff和Keji Zhao等课题组报道了依赖于免疫原位切割并建库测序的方法,包括CUT&RUN,CUT&Tag、scChIC方法等。依据类似的原理,北京大学何爱彬实验室开发了in situ ChIP-seq以及它的高通量、单细胞应用CoBATCH。在该方法中,作者构建了protein A和Tn5的融合蛋白(PAT);前者能识别结合在目标蛋白上的抗体,后者能对目标蛋白附近的DNA进行切割转座,并通过建库测序就能确定目标蛋白在基因组中结合的位置。通过这种方法,作者实现了在少量细胞中检测组蛋白修饰(包括激活形式的组蛋白修饰,也包括抑制性的组蛋白修饰)和一些转录因子的检测。值得一提的是,这种方法能直接应用于未经消化的胚胎组织,因此极大地扩展了可能的应用场景。为了实现单细胞、高通量的检测,作者将in situ ChIP和组合标签标记单细胞的方法结合起来。作者分析了E16.5小鼠不同器官的内皮细胞单细胞H3K27ac的数据,鉴定了其中不同的细胞类群并观测到其中激活增强子的异质性。同时作者也在E16.5的小鼠心脏中进行了Pol II和H3K36me3的单细胞检测,识别了不同的细胞类群和特征。这些结果建立了分析单细胞表观组学数据的基本模式,为挖掘其中的生物学规律提供了可能。
总之,该项技术提供了一种可操作、高通量和单细胞的方法研究蛋白和染色质互作的方法,为进一步研究打开了新的大门。我们期待通过进一步改进这些单细胞技术,或者基于其他原理开发新的单细胞表观组学测序的方法,使得我们能从单细胞中得到更加丰富的数据(coverage更高)、从更加少量的样品中获得数据(如文章中所提,CoBATCH的起始细胞仍需要10, 000),以及能应用于丰度更低的特异性转录因子。我们相信这些技术成熟之后将极大地促进相关生物学问题的研究,通过整合不同的单细胞组学数据(如单细胞RNA-seq的数据和单细胞表观组学的数据)来解析发育和疾病过程中基因的时空表达调控规律。
专家点评
周帆、汤富酬(北京大学)
单细胞组学测序技术的革新使得人们有机会在极限分辨率观察与理解细胞异质性、细胞命运决定与命运转变的重要调控模式与分子机制。
自2009年首次报道单细胞转录组测序技术(Nature Methods, 2009)以来,过去十年的多项技术革新的涌现与整合,使得在单细胞、单碱基的极限分辨率理解生命活动呈现出广阔的应用前景(Nature Method of the Year 2013)。表观遗传修饰与细胞命运决定以及功能特化密切相关。目前除了已经广泛开展的转录组与基因组分析外,表观基因组多个不同层面的分析也逐渐走进研究者的视野,例如DNA化学修饰(DNA甲基化、羟甲基化等)、染色质开放状态、以及基因组三维构象等。然而,表观遗传调控中的重要调控层面-组蛋白的共价修饰-能否实现可操作性强的单细胞分辨率的测定一直备受关注。近日, 北京大学何爱彬课题组在Nature Cell Biology在线发表了题为Profiling chromatin state by single-cell itChIP-seq的文章,研究者们开发了一种全新的具有广泛普适性、优异可操作性的单细胞ChIP-seq技术(itChIP,simultaneous indexing and tagmentation-based ChIP-seq),与同期国际上开发的几种单细胞ChIP-seq技术相比有突出的优点。在该技术探索中,研究者们创新性地利用甲醛和高温SDS使得染色质松散,随后再利用带有序列标签的Tn5酶均匀切割染色质并插入引物序列标签,多个细胞合并后再进行免疫共沉淀。相比于以往报道的少量细胞以及单细胞ChIP-seq技术,该实验过程无需特殊实验设备,体现出优异的用户友好性。
随后作者利用该技术在两种模型(多能性干细胞系EpiLCs、不同胚胎发育阶段的NKX2-5谱系心脏细胞)中清晰呈现了多能性状态以及细胞谱系特异性的组蛋白修饰特征。有趣的是,整合单细胞转录组测序数据后,分析结果显示增强子比其调控的基因表达特征表现出更强的时空特异性。
值得注意的是,不同于该课题组最近开发的CoBATCH单细胞ChIP-seq技术(Wang et al, Molecular Cell, 2019),itChIP尤其适用于捕获起始量只有几十个单细胞的稀有珍贵样品。这项重要的技术革新意义重大,极大地丰富了单细胞表观基因组学研究维度,为诸多生物学研究领域(例如早期胚胎发育与细胞命运决定的表观遗传调控机制、器官再生、肿瘤发生及转移等)提供了全新的探索手段。有了这样强大的新技术的出现,相信单细胞组学测序领域将延续过去十年(2009-2019)黄金期的辉煌,未来十年(2019-2029)将是又一个黄金期,而且会从过去十年的基础生物学研究的黄金期过渡到未来十年的深度临床医学应用的黄金期。
专家点评
朱冰(中科院生物物理所)
工欲善其事,必先利其器。方法学突破始终是科学研究突破的重要源泉之一。在发育特殊阶段研究和细胞异质性研究领域中,单细胞组学信息的获取尤为重要。近年来,单细胞转录组、单细胞DNA甲基化组、单细胞染色质开放性甚至单细胞三维基因组技术都取得了长足的进步。然而,单细胞ChIP-seq技术仍然未有重要的突破。北京大学何爱斌课题组近期在这一领域取得了两项重要成果。上个月,该课题组在Mol Cell发表了CoBATCH技术,可以对大量细胞同时进行ChIP-seq研究,并通过不同的barcode巧妙地拆分来自于单细胞的信息。BioArt日前已经做了介绍(Molecular Cell | 何爱彬组开发高通量、普适性单细胞ChIP-seq技术——CoBATCH),GoBATCH技术对于研究细胞异质性非常有用,但仍然需要较多的起始细胞数量。今天,何爱斌课题组又在Nature Cell Biol杂志发表itChIP-seq技术,该技术可以用数十个细胞作为起始材料,并获得有单细胞分辨率的ChIP-seq信息。这一将有效拓展单细胞ChIP-seq技术的应用面。该技术的一个巧妙之处是在甲醛交联后利用SDS在62摄氏度条件下处理,促进了染色质的开放,从而有利于更均匀地实现抗体结合,有效提升了数据质量。
何爱彬实验室简介:
何爱彬博士,北京大学分子医学研究所研究员,北大-清华生命科学联合中心研究员。实验室成立于2014年,主要研究方向为:1. 哺乳动物器官发育、再生(包括心脏和血液系统)的细胞命运调控机制。为此,课题组利用已有和开发全新单细胞表观遗传技术,高通测序技术和体内外功能基因组验证手段;2. 实时成像结合数字细胞谱系绘制阐明器官发育、再生的细胞与分子机制。“seeing is believing”, 课题组搭建适应哺乳动物胚胎发育的大视野光片显微镜,综合运用遗传标记小鼠,高通量图形图像处理和数字谱系,以期用更加直观方式和定量的手段研究在体全细胞(in vivo and in toto)行为规律。近期课题组发表了一系列相关文章:
1、2019年8月27日,北京大学何爱彬课题组在Molecular Cell在线发表了题为 CoBATCH for high-throughput single-cellepigenomic profiling的文章,报道了一种新的具有普适性、易操作、高通量和高质量的单细胞ChIP-seq技术,将单细胞表观组学新技术的研究、普及和应用往前推进了一大步。研究者把这一新型单细胞技术命名为CoBATCH(combinatorial barcoding and targeted chromatinrelease);
2、2019年5月,在Circulation Research杂志在线发表题为Replication-independent histone turnover underlines the epigenetic homeostasis in adult heart的研究成果。该研究发现与不增殖成体心肌细胞的较长寿命相比,复制非依赖型的核小体更新较快;该机制可控制调控区域的组蛋白乙酰化修饰水平,对心脏功能稳态起重要作用;
3、2019年6月,在Circulation Research杂志在线发表题为“Single-cell transcriptomics revealschemotaxis mediated intra-organ crosstalk during cardiogenesis”的研究成果。先天性心脏病心脏结构发育畸形主要是由于第二心区来源的干细胞发育缺陷所致,然而精确调控分配这波干细胞的机制和理论却不清楚。该研究首次发现趋化作用介导了原始心管对这一波心脏干细胞迁移的直接调控作用,为理解早期心脏不同心区的发育和先心病的治疗提供了重要的理论依据;
4、2017年5月17日,在Circulation Research杂志在线发表题为Divergent requirements for EZH1 in heart development versus regeneration 的研究b成果。该研究揭示同一表观调控因子EZH1在心脏发育和再生过程中,产生不同的组蛋白修饰,导致截然不同的分子机制,调控发育与再生两个关联的生物学过程;
5、2017年4月10日,在eLife上发表了题为EED orchestration of heart maturation through interaction with HDACs is H3K27me3-independent的研究论文。该研究回答了组蛋白修饰H3K27me3还是其修饰酶PRC2本身发挥抑制基因转录的直接作用这一关键科学问题。
原文链接:
https://doi.org/10.1038/s41556-019-0383-5
参考文献
[1] Guo, H., etal., Single-cell methylome landscapes of mouse embryonic stem cells and earlyembryos analyzed using reduced representation bisulfite sequencing. Genome Res,2013. 23(12): p. 2126-35.
[2] Nagano, T.,et al., Single-cell Hi-C reveals cell-to-cell variability in chromosomestructure. Nature, 2013. 502(7469): p. 59-64.
[3] Buenrostro,J.D., et al., Single-cell chromatin accessibility reveals principles ofregulatory variation. Nature, 2015. 523(7561): p. 486-90.
[4] Zheng, H.et al. Resetting Epigenetic Memory by Reprogramming of Histone Modifications inMammals. Mol Cell, 2016. 63, 1066-1079.
[5] Para,Alessia, Ying Li, and Gloria M. Coruzzi. "μChIP-seq for genome-widemapping of in vivo TF-DNA interactions in Arabidopsis root protoplasts."Root Development. Humana Press, New York, NY, 2018. 249-261.
[6] Skene,P.J., Henikoff, J.G. & Henikoff, S. Targeted in situ genome-wide profilingwith high efficiency for low cell numbers. Nat Protoc, 2018. 13, 1006-1019.
[7] Rotem, A.,et al., Single-cell ChIP-seq reveals cell subpopulations defined by chromatinstate. Nat Biotechnol, 2015. 33(11): p. 1165-72.
[8] Kaya-Okur,H.S., et al., CUT&Tag for efficient epigenomic profiling of small samplesand single cells. Nat Commun, 2019. 10(1): p. 1930.
[9] Ku, W.L.,et al., Single-cell chromatin immunocleavage sequencing (scChIC-seq) to profilehistone modification. Nat Methods, 2019. 16(4): p. 323-325.
[10] Wang,Qianhao, et al. "CoBATCH for high-throughput single-cell epigenomic profiling."bioRxiv, 2019. : 590661.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652474815&idx=2&sn=039cbdfe2b8c4b616b3025cdc062f79e&chksm=84e21e0bb395971d74f4ff6a1b8256b87f00cdea7d83bc3d601687cf38c7155f1b8862213a95&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn