科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-09-08
来源:BioArt
对于真核生物来说,将大量的基因组DNA包装到微米尺度的细胞核中是一个非常大的挑战。在细胞周期的任何一个时间点,染色质都必须允许转录能通过合适的空间和时间上调控的方式准确发生,并且保证DNA在细胞分裂的过程中能被准确的复制。在过去的二十年中,我们已经获得了染色质在三维空间组织调控的重要认识【1】。
Hi-C实验显示,基因组在超大规模尺度(mega-base scale)的组织形式主要为A和B两个区室(compartments),它们分别与基因的活性或者非活性形式相关【2】。基因组在亚超规模层次(sub-megabase scale)的组织形式则是拓扑相关结合域(TADs, topologically associating domains)。TAD是基因组中一段物理上联系比较紧密的区域,增强TAD内物理联系将导致其内基因的表达改变,这或许是通过改变基因的启动子区和增强子的联系来实现的【3】。
由黏连蛋白复合体(cohesin complex)挤压DNA形成DNA环是TADs形成的原因,在这个过程中,CTCF蛋白(CCCTC-binding factor)是DNA环的边界,它们将相邻的DNA环分隔开【4】。最近的研究显示,CTCF或者黏连蛋白的敲除都会导致DNA环的消失。
因为CTCF结合位点一般比较长(大约19bp),含有较高的信息量,因此基因组中一些随机突变一般不会导致CTCF结合位点提高很多倍。但有趣的是,在许多物种中,CTCF结合基序以及CTCF蛋白自身的相互结合都会随着SINE(short interspersed nuclear element) B2转座元件的扩张而增强【5】。这提示我们,SINE B2的扩张促进了新的CTCF结合位点的产生,并可能导致物种特异性的染色质环的形成。
2019年9月5日,来自瑞士弗里德里希·米舍尔生物医学研究所(Friedrich Miescher Institute for BiomedicalResearch)的Marc Bühler研究组在Cell杂志上发表了长文The ChAHP Complex Counteracts Chromatin Looping atCTCF Sites that Emerged from SINE Expansions in Mouse,发现作为ChAHP复合体(CHD4, ADNP, HP1)中的一个组分,ADNP能够与CTCF竞争性的结合SINE B2 元件,并因此对染色质的三维形态进行调控。

ADNP是ChAHP复合体的核心组分,ADNP敲除会导致ChAHP的无法组装,因此,ADNP敲除是ChAHP复合体失活的最直接方法。敲除ADNP会导致一些基因表达受到抑制,说明ChAHP复合体能够抑制一些基因的转录。但是,研究发现,ADNP在基因组中的结合位点只有约61%位于基因的启动子区,这说明,在基因启动子区之外的基因组中ChAHP同样发挥着重要的作用。
有趣的是,ADNP的结合位点看起来与CTCF的结合位点比较相像,那么,ADNP是否能够与CTCF直接结合呢?内源免疫共沉淀实验结果显示,ADNP能够与ChAHP的组分CHD4结合,但是ADNP和CTCF之间却不存在相互作用。这说明,ADNP并不是被CTCF招募,而仅仅是与CTCF有着相似的DNA结合位点。通过ChIP-seq实验,研究人员发现,几乎所有的ADNP峰都与CTCF峰重合。接下来,研究人员敲除ADNP,通过一系列的ChIP-seq实验,最终证实,ADNP与CTCF在小鼠基因组中的结合是竞争性的。
通过野生小鼠和Adnp敲除小鼠实验分析,研究人员发现,ADNP敲除并没有大量改变基因组的三维结构。同时,ADNP敲除之后,因为ADNP结合位点的大量暴露,增加了CTCF在基因组上的结合位点,导致CTCF结合模式发生改变,一些TADs形成了大的TADs,一些TADs则分割成了小的TADs。而由于TADs的改变,一些基因的启动子区和增强子的结合发生改变,导致这些基因的表达也受到了影响。
另外,通过对ADNP和CTCF结合位点的分析,研究人员发现,大约88%的ADNP峰与SINE B2元件重合,而仅有38%的CTCF峰与SINE B2元件重合。ADNP比CTCF具有更强的DNA识别能力。进一步的分析发现,ADNP更容易结合到SINE上具有较少分叉的序列上。
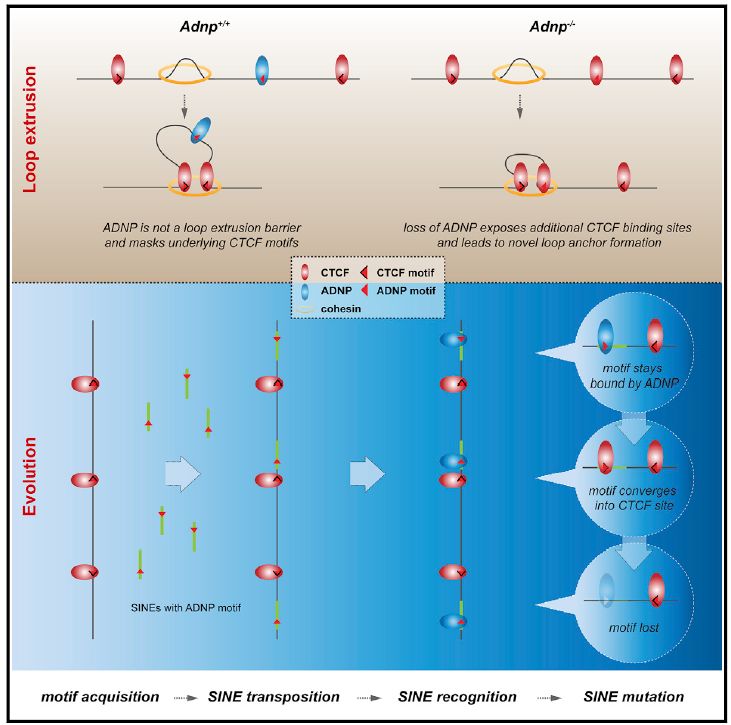
总之,本研究发现,ChAHP复合体能够与CTCF竞争性的结合基因组上的相应位点,通过对基因组三维结构的调控最终影响了基因组的包装以及基因的表达。不过,在基因组中还存在许多CTCF的结合位点不与DNA环的形成有关,因此,一定还存在一些与ChAHP复合体类似的调控因子,对CTCF的功能有着重要影响,这需要进一步的研究。
原文链接:
https://doi.org/10.1016/j.cell.2019.08.007
参考文献
1 Bonev, B., and Cavalli, G.(2016). Organization and function of the 3D genome. Nat. Rev. Genet. 17,661–678.
2 Lieberman-Aiden, E., vanBerkum, N.L., Williams, L., Imakaev, M., Ragoczy, T., Telling, A., Amit, I.,Lajoie, B.R., Sabo, P.J., Dorschner, M.O., et al. (2009). Comprehensive mappingof long-range interactions reveals folding principles of the human genome.Science 326, 289–293.
3 Dixon, J.R., Selvaraj, S.,Yue, F., Kim, A., Li, Y., Shen, Y., Hu, M., Liu, J.S., and Ren, B. (2012).Topological domains in mammalian genomes identified by analysis of chromatininteractions. Nature 485, 376–380.
4 Fudenberg, G., Imakaev, M.,Lu, C., Goloborodko, A., Abdennur, N., and Mirny, L.A. (2016). Formation ofChromosomal Domains by Loop Extrusion. Cell Rep. 15, 2038–2049.
5 Schmidt, D., Schwalie, P.C.,Wilson, M.D., Ballester, B., Gonc¸ alves, A., Kutter, C., Brown, G.D.,Marshall, A., Flicek, P., and Odom, D.T. (2012). Waves of retrotransposonexpansion remodel genome organization and CTCF binding in multiple mammalianlineages. Cell 148, 335–348.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652475001&idx=3&sn=4ac961a3a9175678a8de2372054f2bc7&chksm=84e21dcdb39594db858f55ee49da404822e89dbe67587638602dbc8cb8630b2a66951b9775de&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
metaProdigal:宏基因组序列中的基因和翻译起始位点预测
环境友好的农林害虫生态调控与生物防治技术——2017高级研修班总结

中国科学家团队《Cell》揭示人类大脑进化的基因组调控机制

今日科技话题:水稻高分辨率三维基因组、三千米高度、半远洋沉积、基因位点、北极海冰减少、巨型企鹅化石...

染色质架构蛋白CTCF结合人类基因组位点的机制研究取得新进展

人工种植桃没野生桃香?这25个基因组位点是原因
Molecular Plant 出版“植物激素”专辑
关于举办“环境友好的农林害虫生态调控与生物防治技术高级研修班”通知
metaProdigal:宏基因组序列中的基因和翻译起始位点预测
第一届材料基因组工程高层论坛通知