科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-10-11
来源:Nextomics
2019年9月17日希望组合作项目文章以Long-read assembly of the Chinese rhesus macaque genome and identification of ape-specific structural variants为题,在Nature Communications发表。该研究利用三代测序结合多种辅助技术组装了一个高质量的中国恒河猴参考基因组,极大地改善了当前版本恒河猴参考基因组的连续性和完整性。通过与已发表的猿类基因组比较分析,首次发现了17,000个猿类特有的结构变异(ASSVs),其中包含了一系列与猿类重要表型特征相关的ASSVs。该研究成果为非人灵长类模型在生物医学研究中的应用提供了重要数据,也极大地促进了包括人类在内的灵长类进化的研究。中国科学院昆明动物所和耀喜副研究员为论文的第一作者,博士研究生罗鑫、周斌,硕士研究生胡庭和博士研究生孟晓宇为该文的共同第一作者,宿兵研究员为该文的通讯作者。希望组承担了本研究中三代测序和部分分析工作。
研究背景
在进化上恒河猴属于旧大陆猴类(Old World monkey species),与人型总科的猿类(Apes)属于姊妹群,有着共同的祖先。与旧大陆猴类相比,猿类在进化过程中有着一系列创新,如无尾、巨大的体型、脑容量和复杂性的增加、手的灵活性提升等。结构变异(SVs)在灵长类进化和疾病中起重要作用,可以通过恒河猴与猿类基因组之间的比较分析来探索在恒河猴和猿类的共同祖先中发生的功能性遗传变化。然而,目前尚缺少一个高质量的恒河猴基因组,现有的几个恒河猴基因组序列连续性差(片段化)、完整性差(许多缺口),很难对序列进行系统的识别。
高质量恒河猴基因组
研究人员利用三代测序技术(100×PacBio)结合多种辅助组装策略(101×Bionano,105×Hi-C)构建了一个高质量的中国恒河猴基因组rheMacS,同时利用恒河猴10个组织的全长转录组数据改进了参考基因组的注释。rheMacS基因组大小2.95Gb,ContigN50为8.19 Mbp,BUSCO完整性评估达93.5%,与现有的印度恒河猴基因组相比,连续性提升了75倍,填补了2万多个缺口,极大地改善了当前版本恒河猴参考基因组的连续性和完整性。
三代测序技术
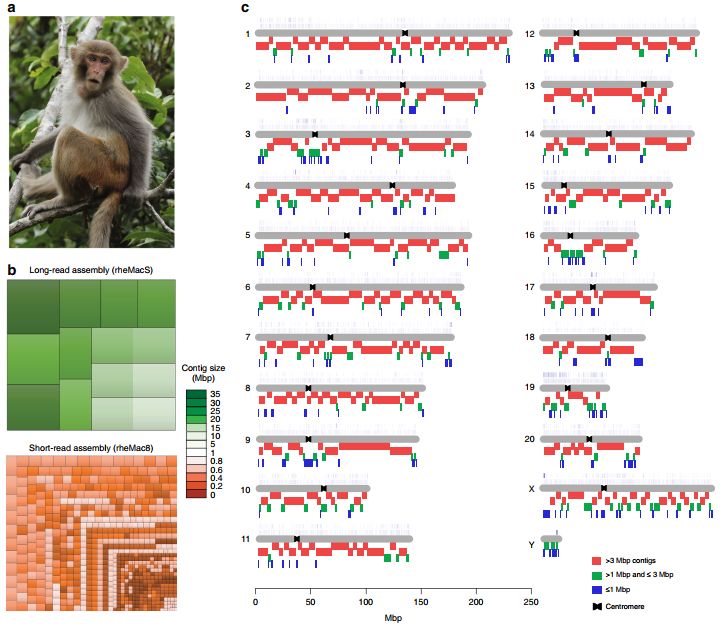
图1 rheMacS基因组与印度恒河猴组装质量对比
图1 中国恒河猴基因组的长读长组装
猿类特异性结构变异(ASSVs)鉴定
研究者在rheMacS中鉴定了53,916个SVs(图2a),其中96%(51,919/53,916)是全新的SVs,在之前使用array和NGS平台的研究中没有被观察到(图2e),可见长读长测序数据在全基因组SV检测中有巨大优势。
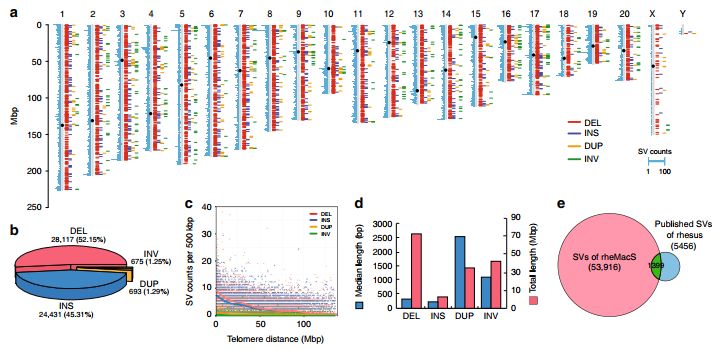
图2 rheMacS中的结构变异(SVs)
结合已经发表的高质量猿类基因组,首次有机会识别从共同祖先中分化以来出现在猿类谱系中的特异性结构变异(ASSVs)。通过比较基因组学方法,将rheMacS组装和三个已发表的猿类基因组、人类ZF1基因组以及狨猴基因组进行比较,筛选过滤后发现了17,000个候选ASSVs,包括13,456个删除和3,544个插入(图3a,b)。对这些ASSVs进行注释,有12,255个定位于3,412个编码基因内或附近(图3c)。其中,有25个ASSVs位于基因外显子上(共涉及32个基因),其余位于基因内含子上,提示其潜在的功能影响可能与基因表达调控有关。
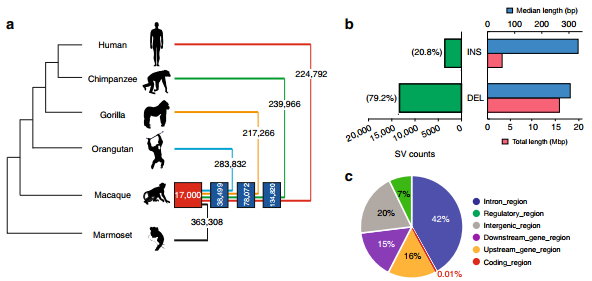
图3 猿类特异性结构变异(ASSVs)
大脑调节元件中的ASSVs
利用先前发表的来自人类、黑猩猩和恒河猴的脑组织不同区域的ChIP-Seq数据和RNA-Seq表达数据,发现大量ASSVs映射到猿类与恒河猴差异增强子区域(ADEs),筛选后其中21个ASSVs对应20个ADEs具有高可信度(图4a)。使用PCR和Sanger测序对两个ASSVs进行了实验验证,发现587bp的缺失破坏了5个猿类大脑区域的ADE(4b),其编码Intersectin-2,影响网格蛋白介导的内吞作用,对神经元突触囊泡的循环起关键作用。由于ASSV干扰了ITSN2中的增强子区域,ITSN2在人类和黑猩猩中的表达明显低于恒河猴(4d),这与其增强子活性减少一致。
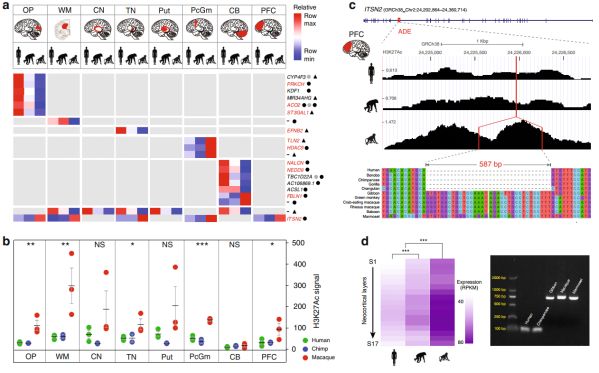
图4 与大脑调节相关的ASSVs
猿类特异表型性状相关ASSVs
猿类在进化过程中有着一系列重要特征创新,如无尾、巨大的体型、脑容量和复杂性的增加、手的灵活性提升等,通过进一步的分析研究人员找到了一系列与以上表型特征相关的候选ASSVs。例如位于与尾巴发育相关的CDH8基因的477 bp的缺失(图5e);与人类胎儿拇指内收相关的NALCN基因178bp缺失(图5f)等。这一系列与猿类特异性性状相关的候选ASSVs可作为研究猿类进化过程中出现的表型创新的遗传基础。
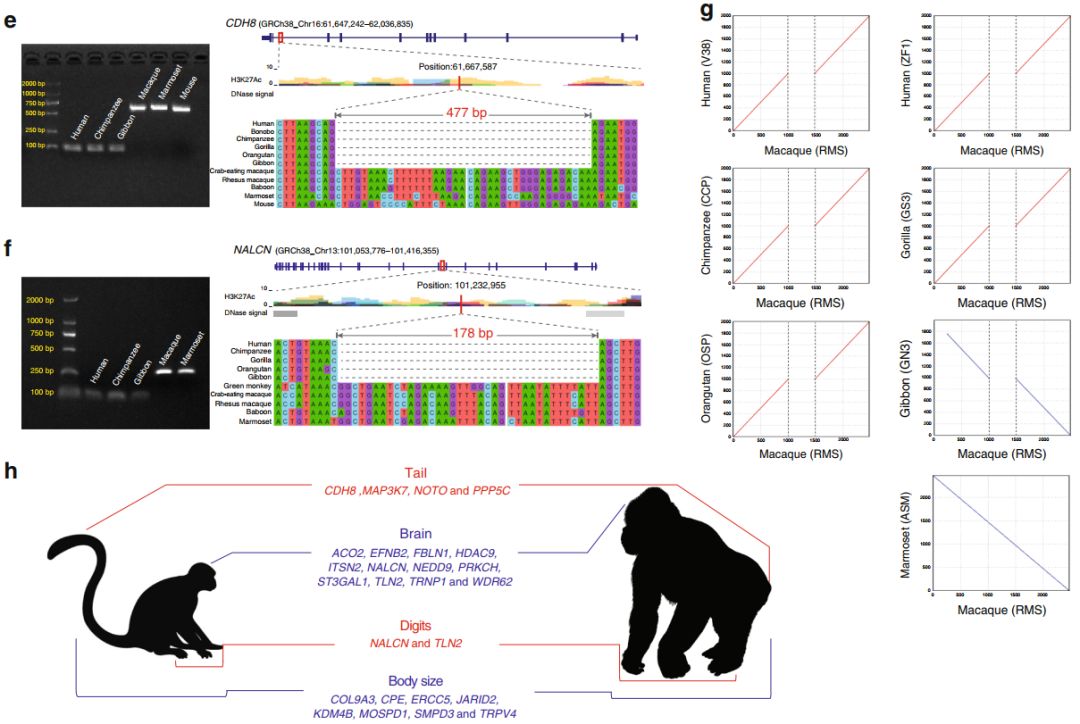
图5 猿类特异表型性状相关ASSVs
小结
该研究公布了一个高质量中国恒河猴参考基因组,极大地改善了当前版本恒河猴参考基因组的连续性和完整性。基于此恒河猴组装,通过与已发表的猿类基因组比较分析,使我们有机会识别从共同祖先中分化以来出现在猿类谱系中的特异性结构变异(ASSVs),首次发现了17,000个ASSVs,其中包含了一系列与猿类重要表型特征相关的ASSVs,如无尾、巨大的体型、脑容量和复杂性的增加、手的灵活性提升等。该研究成果为非人灵长类模型在生物医学研究中的应用提供了重要数据,也极大地促进了包括人类在内的灵长类进化的研究。
来源:gh_ef83defd2d1d Nextomics
原文链接:http://mp.weixin.qq.com/s?__biz=MzA5MzI3NDczNQ==&mid=2650985961&idx=1&sn=e53f053677c2baee8ca3f0f088f13c34&chksm=8b9622e9bce1abffc750ce9c91c13c47f7eff9d050b1fd976a110171209c9b685df8eb43b068&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
“垃圾DNA”对于染色体组连接至关重要
胰腺特异性抗原(PaA)
胰腺特异性抗原(PaA)
第一届材料基因组工程高层论坛通知

科研进展|昆明植物所在叶绿体基因组注释软件开发取得重要进展
遗传学:解密古埃及木乃伊基因组

3D生物打印血管动物试验成功

华中农业大学教授赵书红:在基因组上“跳舞”
种质库在叶绿体基因组注释软件开发取得重要进展

小麦D基因组完整图谱首绘成功