科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-10-30
来源:BioArt
原标题:Molecular Cell:伊成器/樊嘉合作组绘制人与小鼠m6A和m6Am甲基化图谱
m6A修饰是真核生物mRNA和lncRNA上分布最广泛的化学修饰。作为一种动态、可逆的RNA修饰,m6A由甲基转移酶复合物(METTL3/METTL14/WTAP/KIAA1429/RBM15等)催化形成,以及FTO和ALKBH5等“eraser”擦除,并可以被 “reader”蛋白(YTHDF1/2/3等)识别,从而参与包括RNA稳定性调节、剪接加工、转运等一系列mRNA代谢过程的调控【1】。大量研究表明,m6A修饰在调节昼夜节律、脂肪形成、精子生成、胚胎干细胞自我更新和分化、神经发育等生命活动中,以及许多癌症的发生与发展等病理过程中发挥重要作用【2-3】。
与位于RNA内部的m6A修饰不同,m6Am修饰主要位于真核生物mRNA 5’端 m7G帽子后的第一个碱基。最近研究发现FTO可以去m6Am修饰【4-5】;伊成器课题组及多个研究小组独立地发现帽子特异的甲基转移酶PCIF1可以介导m6Am的形成(Cell Research | 伊成器组鉴定新型RNA修饰m6Am甲基转移酶)【6-9】。
尽管一些重要的表观遗传学(例如DNA的5mC修饰)或表观转录组学图谱(例如A-to-I)已在组织或细胞水平被系统地绘制,但m6A和m6Am的甲基化图谱仅仅局限于有限数量的哺乳动物细胞系和小鼠组织水平。人体和小鼠组织水平的m6A和m6Am甲基化修饰图谱迄今尚未被系统地解析。
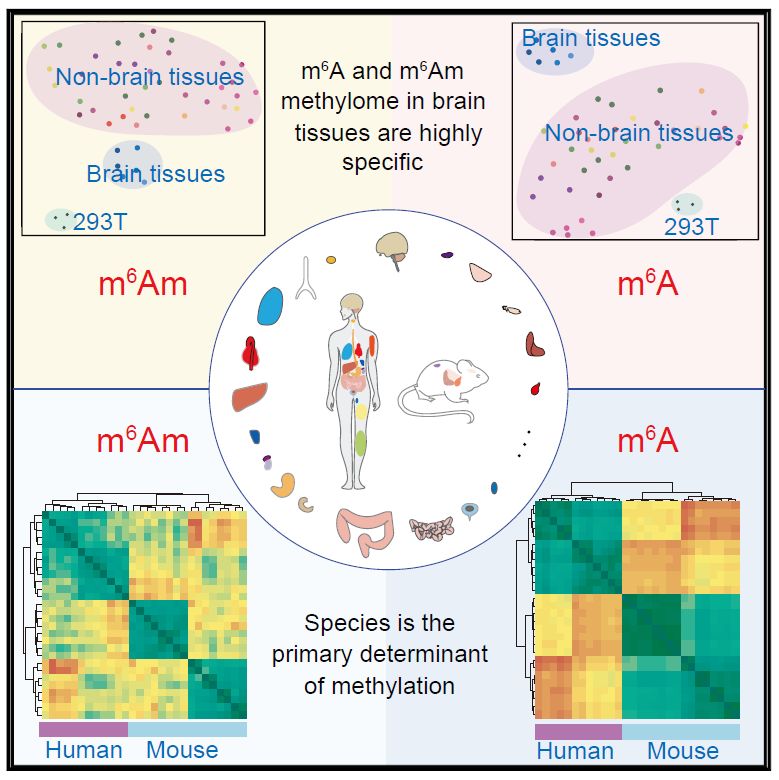
2019年10月29日,北京大学生命科学学院伊成器课题组与复旦大学附属中山医院樊嘉院士课题组合作在Molecular Cell杂志在线发表了题为 Landscape and regulation of m6A and m6Am methylomeacross human and mouse tissues的论文,研究人员通过对43个人体组织样本(来自7个人的26个身体部位),16个小鼠组织样本以及9种人类细胞系样本中的全转录组m6A和m6Am图谱进行测序及系统、全面的生物信息学分析,概括了其在人体和小鼠组织中的分布规律,揭示了m6A和m6Am在脑组织具有较强的组织特异。这一特异性最近在人类胚胎组织中也得到了证实【10】。

该研究鉴定了大量保守的和组织特异的m6A修饰位点,并可以利用组织特异性的m6A位点来区分不同的组织类型。此外,研究人员还发现m6A和m6Am修饰含量与其各自对应的甲基转移酶和去甲基化酶存在显著相关性;并且m6Av包含区附近富集了大量潜在功能性SNP位点(如数量表达性状(eQTL)SNP和脂代谢异常等疾病相关的GWAS SNP)及microRNA 靶向位点。该研究也发现,在人体组织中m6Am修饰与蛋白表达水平呈现负关联。跨物种的m6A和m6Am甲基化图谱分析表明物种才是m6A和m6Am修饰的主要决定因素。该研究为深入理解人体和小鼠的m6A和m6Am修饰的动态变化规律及调控机制提供了重要的生物数据资源。
原文链接:
https://doi.org/10.1016/j.molcel.2019.05.030
参考文献
1. Shi, H., Wei, J., and He, C. (2019). Where,When, and How: Context-dependent functions of RNA methylation writers, readers,and erasers. Mol Cell 74, 640-650.
2. Licht, K., and Jantsch, M.F. (2016). Rapid anddynamic transcriptome regulation by RNA editing and RNA modifications. J CellBiol 213, 15-22.
3. Liu, J., Harada, B.T., and He, C. (2019).Regulation of gene expression by N(6)-methyladenosine in cancer. Trends CellBiol 29, 487-499.
4. Mauer, J., Luo, X., Blanjoie, A., Jiao, X.,Grozhik, A.V., Patil, D.P., Linder, B., Pickering, B.F., Vasseur, J.J., Chen,Q., et al. (2017). Reversible methylation of m(6)Am in the 5' cap controls mRNAstability. Nature 541, 371-375.
5. Wei, J., Liu, F., Lu, Z., Fei, Q., Ai, Y., He,P.C., Shi, H., Cui, X., Su, R., Klungland, A., et al. (2018). Differential m(6)A,m(6)Am, and m(1)A demethylation mediated by FTO in the cell nucleus andcytoplasm. Mol Cell 71, 973-985 e975.
6. Sun, H., Zhang, M., Li, K., Bai, D., and Yi, C.(2019). Cap-specific, terminal N(6)-methylation by a mammalian m(6)Ammethyltransferase. Cell Res 29, 80-82.
7. Akichika, S., Hirano, S., Shichino, Y., Suzuki,T., Nishimasu, H., Ishitani, R., Sugita, A., Hirose, Y., Iwasaki, S., Nureki,O., et al. (2019). Cap-specific terminal N (6)-methylation of RNA by an RNApolymerase II-associated methyltransferase. Science 363.
8. Sendinc, E., Valle-Garcia, D., Dhall, A., Chen,H., Henriques, T., Navarrete-Perea, J., Sheng, W., Gygi, S.P., Adelman, K., andShi, Y. (2019). PCIF1 catalyzes m6Am mRNA methylation to regulate geneexpression. Mol Cell 75, 620-630 e629.
9. Boulias, K., Toczydlowska-Socha, D., Hawley,B.R., Liberman, N., Takashima, K., Zaccara, S., Guez, T., Vasseur, J.J.,Debart, F., Aravind, L., et al. (2019). Identification of the m(6)Ammethyltransferase PCIF1 reveals the location and functions of m(6)Am in thetranscriptome. Mol Cell 75, 631-643 e638.
10. Xiao, S., Cao, S., Huang, Q., Xia, L., Deng,M., Yang, M., Jia, G., Liu, X., Shi, J., Wang, W., et al. (2019). The RNAN(6)-methyladenosine modification landscape of human fetal tissues. Nat CellBiol.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652477152&idx=2&sn=880151fe6d91be6bc6138e19a209a1dd&chksm=84e20554b3958c42b1850e45b8b060fc05d9f586bf85105d5ba4b490f2b6972c81d8eae114eb&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
3-甲氧基-N-甲基苯甲基胺
Cell | 首次将大肠改造为转化为仅在甲醇上生长的甲基营养菌,在工业有极大价值
甲基硼酸
Cell:利用DNA甲基化图谱重构古人类解剖学特点
4-(羟甲基)环己基甲基乙烯醚

活动预告丨“CAAI进校园”系列活动走进电子科技大学

中文解读 | 基底膜在血脑屏障发育及维护屏障功能中的作用
浙大34岁“歌神教授”:科研也是在创造艺术品



Cell子刊:维生素C可以激活DNA去甲基化,促进浆细胞分化,增强抗体产生!

Cell :从头甲基转移酶丢失后,酵母DNA甲基化仍可维持数百万年