科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-11-08
来源:BioArt
基因及其表达模式是表征和决定细胞状态的基础,因而基因表达的调控机制也是分子生物学研究的核心大课题之一。在基因调控的多个层级中,转录调控(transcriptional regulation)是最早受到关注和研究得最为透彻的分支。经过数十年的研究(详见BioArt报道:致敬Robert Roeder & 纪念真核生物RNA聚合酶发现50周年——转录调控研究简史),人们已经知道,在原核和真核生物中,基因转录水平的决定因素都包括顺式调控元件(cis-regulatory element)和反式作用因子(trans-acting factor)两大要件【1,2】。在转录调控的语境下,前者主要由启动子、增强子和沉默子等非编码DNA序列构成;而后者则包含RNA聚合酶、转录因子、染色质重塑因子(chromatin remodeler)、甚至是一些RNA结合蛋白(RNA binding protein, RBP)等【3】。
尽管从研究范式上考虑,顺式元件和反式因子可以被认为在基因转录调控中各自做出独立贡献,因而获得同等的重视。但就生物机制的前后逻辑而言,顺式元件所蕴含的内在序列特征(intrinsic sequence pattern)往往直接决定了反式因子的结合与作用模式。一个经典的例子是,由ChIP-seq测定的人与小鼠肝细胞关键转录因子HNF1α、HNF4α和HNF6虽然具有几乎完全保守的DNA结合域,但却在两类物种间表现出极为不同的全基因组结合模式;然而,当研究者将人类21号染色体转入小鼠肝细胞后,却发现小鼠细胞的上述因子表现出与人同源因子几乎一致的对外源人DNA序列的结合模式;因而证实了小鼠与人之间转录因子的结合模式差异并不来自于因子本身或细胞整体环境的差异,而是几乎由物种间DNA同源序列的变异所决定【4】。当然,这并不意味着基因调控的根本逻辑是“序列决定论”:毕竟,拥有同一套DNA序列的不同组织和细胞类型、不同生理和病理条件,往往表现出高度差异化的基因调控模式。即使将各类表观遗传修饰差异,如DNA甲基化、染色质开放性等考虑进去,作为概念延展后的顺式元件,也不能完全预测和解释基因调控的多样性。也即是说,反式因子在不同生物场景下的差异性行为仍然是形塑异质化基因调控的重要元素,只不过它并非基于一个独立于顺式元件自行作用的逻辑,而是类似于“鸡”与“蛋”的相辅相成的复杂逻辑【5】。
因此,对各类顺式调控元件所遵循的序列逻辑的解析就显得极为重要。通常,这类信息可以由共识序列(consensus sequence)、结合基序(binding motif)和位置权重矩阵(position weight matrix)等形式表征。虽然这些方法在大致判断顺式元件的激活程度或反式因子结合模式方面具有一定的效果,但它们所容纳的信息量少,且灵活性差,无法用来精准预测顺式元件的行为。与基于短序列匹配的传统方法相比,机器学习和深度学习方法由于具有强大的模式提取(pattern extraction)能力,极大地提升了对顺式元件内在序列特征的认知能力,被成功运用在了包括DNA甲基化概率、RNA结合蛋白的结合位点和染色质三维关联等预测任务中【6-8】。
2020年9月9日,来自加州大学圣地亚哥分校(UCSD)在转录调控研究方面曾产出多项重量级结果的James Kadonaga【9】(其博士后导师为著名美籍华裔科学家钱泽南Robert Tjian,为转录调控研究方面的先驱)课题组在Nature上发表了题为Identification of the human DPR core promoter element using machine learning的研究,基于大规模DNA序列筛选数据建立了预测下游核心启动子序列活性的机器学习模型,首次证实了下游核心启动子序列的存在,再一次展现了机器学习方法在基因调控甚至是整个基因组学研究中的巨大潜力。
控制基因表达开关的启动子核心序列通常被认为是转录起始位点(transcription start site, TSS)上下游分别约40bp的DNA区域,而这一区域往往具有跨物种、跨基因的保守性序列特征,如最为著名的七碱基TATA盒(TATA box)。不过,TATA box本身只出现在约25%的人类基因启动子序列中。尽管在非TATA box启动子中也发现了诸如十基序元件(ten motif element)和下游核心启动子元件(downstream core promoter element)等,但和TATA box一样,各类元件均以不甚高的频率散布在部分基因的启动子中。因此,启动子序列的内在特征是否具有统一性和普适性,仍然是个未解之谜。另外,核心启动子序列中研究较为缺乏的位于+17至+35的下游核心序列DPR(downstream core promoter region)的存在性,也是一个悬而未决的问题。
为了回应这一挑战,该研究的作者希望解答的一个关键疑问是,DPR是否存在某种一般性的序列特征使其能够具有最优化的激活效应?为此,基于用于大规模DNA序列活性筛选的SuRE(survey of regulatory elements)方法【10】,作者首先合成了50万个携带随机序列的DPR,然后嵌入同一启动子框架(promoter cassette)中,并经由细胞外或细胞内表达系统进行转录,最后以带有特定序列的RNA数目与初始DNA数目的比例作为某一DPR激活程度的表征(下图)。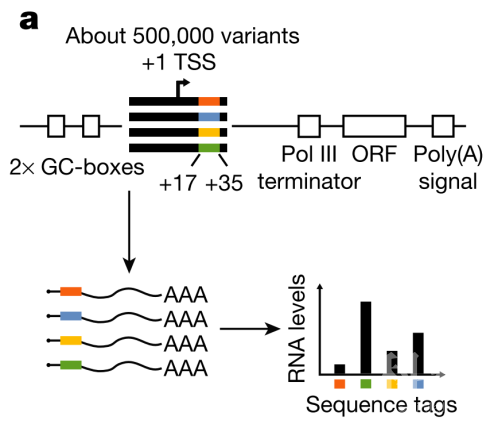
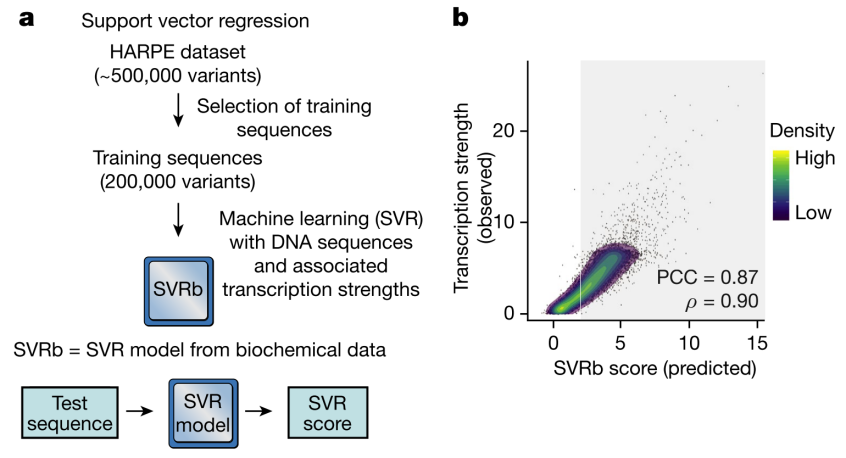
在获得了关于DPR序列对其激活程度影响的大规模筛选数据之后,作者考虑利用机器学习方法构建一个能够将DPR序列的内在特征进行精准识别和提取,并映射至对应激活程度的预测模型。经过一系列对比,作者选择了非线性机器学习模型支持向量机回归方法(Support vector regression, SVR)来实现这一目的,结果发现,在独立测试集上,该模型所预测出的转录强度与真实值具有高达0.9的相关系数,且呈较好的线性关系(下图)。在基于细胞内转录系统所产生的数据上重复这一模型构建,依然获得了相当良好的预测精度和复现性。因此,机器学习模型不仅能够实现对未知DPR序列的转录强度的精准预测,更重要的是,其在预测任务上的极佳表现直接反映了与转录强度相关的DPR本身的内在序列特征的存在性。
 利用上述构建的DPR序列到转录强度的映射模型,作者进一步分析了真实存在于人体细胞基因启动子序列中的DPR的分布状况。结果发现,约25-34%的启动子相应区域均被预测具有显著较强的DPR激活程度(定义为转录强度超过沉默DPR的6倍及以上);与之相反,利用传统的DPE位置权重矩阵进行扫描仅能在0.4%-0.5%的启动子中发现DPR。因此,这一结果证明了DPR是一个真实存在于基因启动子下游区域的调控元件,只是其内在序列特征较为复杂和“无序”,并不能由共识短基序等常规方法进行表征和预测,但却能够为机器学习模型所识别。
利用上述构建的DPR序列到转录强度的映射模型,作者进一步分析了真实存在于人体细胞基因启动子序列中的DPR的分布状况。结果发现,约25-34%的启动子相应区域均被预测具有显著较强的DPR激活程度(定义为转录强度超过沉默DPR的6倍及以上);与之相反,利用传统的DPE位置权重矩阵进行扫描仅能在0.4%-0.5%的启动子中发现DPR。因此,这一结果证明了DPR是一个真实存在于基因启动子下游区域的调控元件,只是其内在序列特征较为复杂和“无序”,并不能由共识短基序等常规方法进行表征和预测,但却能够为机器学习模型所识别。
总之,这项研究结合了大规模DNA序列活性筛选和机器学习模型两项分别在实验分子生物学和数据科学中处于前沿地位的方法,成功构建了在转录调控中扮演关键角色的启动子的部分区域及下游核心区域的序列特征与其转录强度之间存在的难以被人类直觉所探知的复杂非线性映射关系。其在解答DPR存在性问题上另辟蹊径,通过证明人类基因启动子序列显著富集了高激活DPR序列,传达出由自然选择所形塑的顺式元件的分子演化历程的确创造了DPR这一元件的关键结论。
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652507278&idx=3&sn=ada2e0985f87c99bd8084a68ca34ab53&chksm=84e19f3ab396162c06633db11d7836fc72cbc49119af1ea39c8aeada7e322effd53d7bcdc6f3#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

机器学习帮助精确分析黑猩猩行走特征
增强子选择启动子的新模型
IBM研发出无监督式机器学习算法
Cell子刊: 脑袋和肚子长得不一样, 胚胎发育学家基因启动子上找答案
神经科学: “快进”脑补看生活
通过目标启动子基因编辑及病原菌群体监控实现水稻白叶枯病持久广谱抗性
机器学习技术使得自动化原子级制造成为现实

医疗数据非结构化广泛存在 医疗AI落地待解

人工智能被用于重构量子系统,为量子研究提供新途径
中国科学院揭示热纤梭菌转录调控因子σI的独特启动子识别机制