科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-11-12
来源:Nextomics
原标题:中国科学院武汉病毒研究所刘翟课题组利用纳米孔测序技术成功解析一例非洲猪瘟病毒基因组
2018年8月,非洲猪瘟疫情在我国首次发生,非洲猪瘟具有较高的感病率和致死率,而国内生猪疫情防治水平较低。这导致我国生猪出现大面积死亡,生猪数量骤减,产能恢复压力较大。据农业农村部数据显示,截至2019年5月3日,我国累计发生非洲猪瘟疫情129起,合计扑杀生猪高达102万头。截至目前,全球共有60多个国家报道过非洲猪瘟疫情。
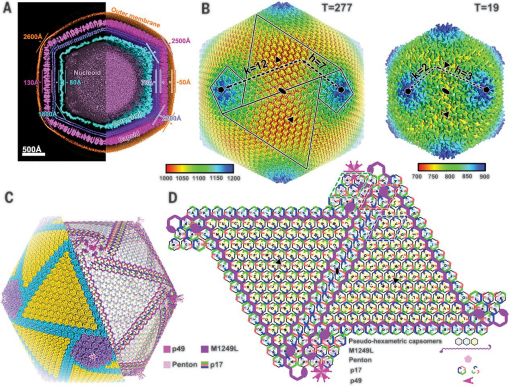
非洲猪瘟病毒结构图(饶子和院士团队解析)[1]
非洲猪瘟由大型双链DNA 病毒非洲猪瘟病毒(ASFV)引起。根据B646L基因可将非洲猪瘟病毒分为24种基因型。不同分离株的基因组长度在170~190kbp之间变化,基因组中央区是长约 125 kb 的保守区,两端基因区含有数个多拷贝基因家族因而序列长度变化较大。由于两端基因拷贝数不同,导致不同毒株间的基因长度差异较大。编码151~167个开放阅读框,170多种蛋白质。病毒颗粒是直径为 200 nm 的正二十面体, DNA 核心直径为 70~100 nm,外周是直径为 170~190 nm 的衣壳和含类脂的囊膜。非洲猪瘟病毒主要通过巨胞饮和网格蛋白介导的胞吞方式进入宿主细胞,主要感染猪网状内皮细胞和单核巨噬细胞,引发细胞凋亡,严重影响宿主免疫系统。
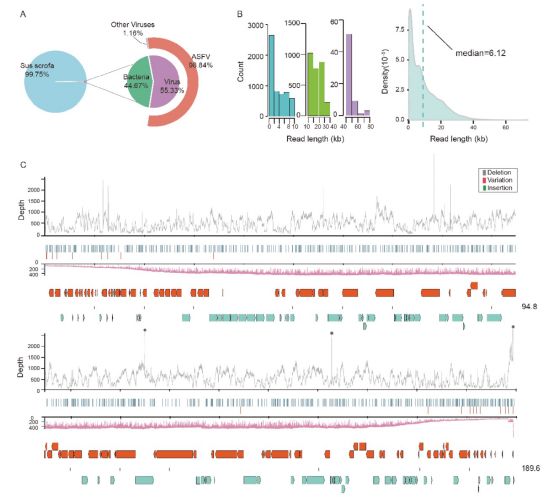
图1. CAS19-01的纳米孔测序[2]。A.测序样品宏基因组分类;B.Nanopore测序reads读长分布;C.Nanopore与illumina测序数据对比。
非洲猪瘟疫情在我国爆发以来,对相关行业造成了重大的影响,也得到了有关部门的高度重视。中国科学院武汉病毒研究所刘翟研究员团队在疫情爆发的第一时间对非洲猪瘟病毒进行研究,利用北京希望组Nanopore PromethION平台,对一株新分离的非洲猪瘟病毒株样本进行了测序,实验所使用的PromethION测序平台具有具有高通量、长读长的特点。测序共得到8,517 个病毒reads ,平均测序深度达到了330.08× ,其中最长的reads长度达到了73.78 kb,相当于直接覆盖了非洲猪瘟病毒基因组的 38.93% 。本次实验所得到的病毒基因组被命名为CAS19-01,通过二代测序技术对数据进行修正,发现该基因组全长189,405 bp ,编码 183个开放阅读框。对珠海CAS19-01病毒株的测序充分证明Nanopore测序技术具有快速、高通量、长读长的特点,在未来的疾病诊断与疫情防控方面能够发挥巨大的作用。
该研究成果以Nanopore sequencing of African swine fever virus为题于2019年11月5日在SCIENCE CHINA Life Sciences 发表。
参考文献:
[1] Wang N, Zhao D, Wang J, et al. Architecture of African swine fever virus and implications for viral assembly[J]. Science, 2019, 366(6465): 640-644.
[2] Jia L, Jiang M, Wu K, et al. Nanopore sequencing of African swine fever virus[J]. Science China Life Sciences, 2019: 1-5.
来源:gh_ef83defd2d1d Nextomics
原文链接:http://mp.weixin.qq.com/s?__biz=MzA5MzI3NDczNQ==&mid=2650986124&idx=1&sn=d604b0ac0aac186fb3670a95d47c78f2&chksm=8b96218cbce1a89a517d1d70f91910db07ae902e6dbae97d9200742faa6d0ba481b9af883bc7&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn