科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-12-09
来源:BioArt
DNA的精确复制对于维持基因组稳定性至关重要。然而复制叉时常会遭受来自细胞内外的复制压力而停滞,如果无法及时重新起始,将造成复制叉崩塌,影响基因组稳定性甚至导致细胞死亡。1976年Higgins N. Patrick等人在研究复制相关的损伤修复时,在哺乳动物细胞中观察到了一种Holliday Junction的结构。他们猜测该结构形成的原因是复制叉停滞后,两条新合成的DNA子链与亲代DNA链解开后发生退火而翻转,进而将正常的复制叉结构重塑成Holliday Junction结构(图一),该过程也被称为复制叉翻转。
由于Holliday Junction结构的形状与鸡爪相似,生物化学教科书中也通常将其称为“鸡爪结构 (chicken foot)”。受检测技术的限制,直到2002年José M. Sogo等人才在检验点缺陷的酵母细胞中观察到“鸡爪结构”,“鸡爪结构”也因此被认为是复制失败的副产物,其生理意义一直存在争议。近年来研究表明,在高等真核生物细胞中,复制叉翻转是由PARP1介导的应对复制压力的关键调控机制,酵母中由于缺乏PARP基因,因而在正常生理条件下极少能够观察到“鸡爪结构”的存在。
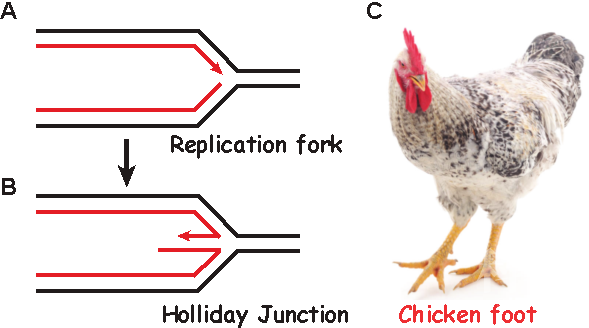
图一:复制叉翻转时“鸡爪结构”的形成
2020年12月8日,浙江大学黄俊课题组在Molecular Cell杂志上发表文章The ZATT-TOP2A-PICH Axis Drives Extensive Replication Fork Reversal to Promote Genome Stability,提出了复制叉翻转时“鸡爪结构”形成的机制以及在维持基因组稳定中的功能。

黄俊实验室在电镜下观察“鸡爪结构”时,注意到翻转的第四条链的长度可以达到数千碱基对。显而易见的是,在复制叉翻转过程中,新合成的链与亲代DNA链解开将会在位于复制叉后方新合成的DNA双链上产生拓扑张力,进而抑制复制叉的进一步翻转。因此他们猜测复制叉在翻转过程中需要通过拓扑异构酶释放拓扑张力,促进复制叉的深度翻转(图二)。他们通过EM (Electron Microscope)、single-molecular DNA fiber、iPOND (isolation of proteins on nascent DNA)、PLA (Proximity Ligation Assay)等实验手段发现拓扑异构酶TOP2A正是释放复制叉翻转过程中产生的拓扑张力的关键因子。TOP2A通过感应拓扑张力而被募集到停滞的复制叉处,在释放拓扑张力的同时招募SUMO化修饰的E3连接酶ZATT对自身进行SUMO化修饰,进而招募下游SUMO靶向的转位酶PICH对复制叉进行进一步翻转。
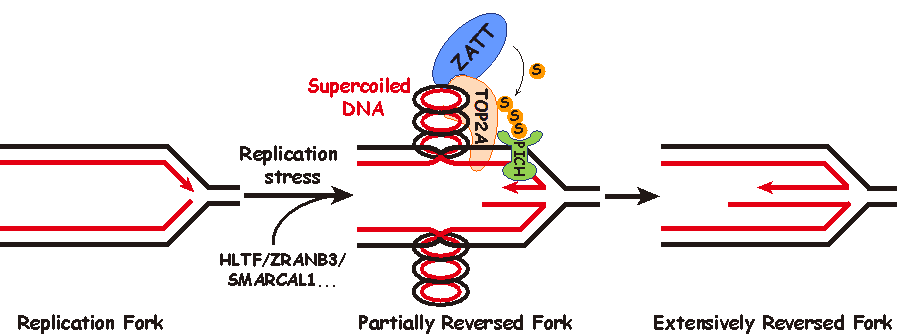
图二:新合成的链与亲代DNA链解开产生拓扑张力
该研究首次提出了“鸡爪结构”是通过两步级联反应而形成的新模型(图二)。第一步,在复制叉遭遇复制压力时,复制叉发生停滞,由转位酶HLTF、ZRANB3、SMARCAL1等蛋白催化复制叉的初始翻转,同时在复制叉后方新合成的DNA双链上产生拓扑张力;第二步,拓扑异构酶TOP2A释放拓扑张力,并通过SUMO化修饰招募转位酶PICH促进复制叉的深度翻转。进一步的功能研究表明,复制叉的深度翻转对于维持基因组稳定性至关重要。
浙江大学黄俊博士实验室的博士研究生田甜、卜敏、陈旭、丁林丽和阳玉兰为论文共同第一作者,黄俊教授为通讯作者。该研究工作也得到了北京大学李晴教授的大力支持。
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652513023&idx=6&sn=9530ed3397cedce123087cf2e3994660
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
【复材资讯】科学家首次实现陶瓷4D打印
警惕!这些鸡翅、鸡爪千万别吃

干货 | 美国空军实验室综述:高熵合金的特点及其研究现状
呼吸肌张力的测定
张力推平台
紧急提醒!这些鸡翅、鸡爪千万别吃
无张力轧制
位错线张力

戏水池看表面张力
肌张力低下型脑瘫