科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-11-20
来源:BioArt植物
原标题:Nature Plants:植物体内RNA 稳态调控新机制!连接RNA 质量控制与转录后基因沉默的桥梁被揭示
为了维持机体各项功能的正常运行,不论是动物还是植物都需要机体有一个相对稳定的理化状态,即是稳态 (homeostasis)。这对于植物体内RNA 水平的稳定状态也同样适用,在植物体内有两种调控RNA水平的系统:RNA质量控制 (RQC) 和转录后基因沉默 (PTGS)。
RQC 是一种进化上保守的RNA监测机制,它检测并去除内源性异常RNA,以防止潜在毒性蛋白的产生【1】。在植物中,异常的RNA会被5’-3’ XRN 核酸外切酶和3’-5’ 外切体复合物降解【2】。PTGS 是一种抑制侵袭性RNA (包括转基因和病毒RNA) 的调节机制,也参与调控逆境响应和发育过程中的内源性RNA。PTGS 通常是通过短干扰siRNAs (interfering RNAs) 和miRNAs (microRNAs) 来发挥作用。在植物中,SGS3 (SUPPRESSOR OF GENE SILENCING 3) 和RDR6 (RNA DEPENDENT RNA POLYMERASE 6) 将单链RNA 转化为双链RNA,接着,DCL2 (DICER-LIKE 2) 或DCL4 将双链RNA 加工成21-22 nt 的siRNAs,这些siRNAs最终被整合到AGO1 中以促进靶RNA 的切割【3】。
RQC 是植物抵御外源RNA 入侵和异常内源RNA 积累的重要防御机制。RQC 途径与PTGS 在植物体内相互竞争起作用:靶RNA 被RQC 的组分迅速降解,阻止它们转化为双链RNA,而双链RNA 是触发PTGS 所必需的【3】。不过,目前关于RQC 途径与PTGS 是如何相互调控的尚不清楚。
近日,韩国科技研究所 (DGIST) June M. Kwak 教授课题组在 Nature Plants 发表了题为 Proteasome subunit RPT2a promotes PTGS through repressing RNA quality control in Arabidopsis 的研究论文,揭示了蛋白酶体亚基RPT2a 能够通过抑制拟南芥的RQC,进而促进PTGS,从而来调控外源性RNA 的水平。

该研究首先构建了一个报告系统 (PORI system),用于检测和分析由miRNA 和siRNA 介导的PTGS,结果发现,siRNA 介导的PTGS 在GFP 转基因沉默中发挥主要的作用。在对PORI 植株研究的过程中,研究人员分离得到一个突变体rpt2a-6,其对应的基因编码一个26S 蛋白酶体的亚基RPT2a。对该突变体分析发现,PORI/rpt2a-6 能够恢复PORI 植株中GFP 的荧光 (如下图),这表明RPT2a 对PORI植株中GFP 的沉默具有一定的贡献。进一步的分析发现,RPT2a 能够促进产生转基因衍生的siRNA (transgene-derived siRNA),因此这也就解释了PORI/rpt2a-6 能够恢复PORI 植株中GFP 荧光的原因。
因为RPT2a 是26S 蛋白酶体的一个亚基,那么RPT2a 所介导的GFP 的沉默是26S 蛋白酶体的功能,还是RPT2a 所特有的呢?通过对其他亚基的分析发现,RPN12a 的突变能够部分的恢复GFP 的荧光,而RPT4a 的突变对GFP 的荧光则没有变化,这一结果表明26S 蛋白酶体是PTGS 所必需的,而其亚基RPT2a 在这方面则发挥着核心的作用。
RQC 途径与PTGS 是两个相互拮抗的生物学过程,为了进一步研究RPT2a 的作用机制,作者分析发现,RPT2a 能够定位于含有P-bodies (RQC 组分) 和siRNA-bodies (PTGS 组分) 的颗粒中,并且能够与RQC 组分DCP2,RRP41 和RRP45a 发生相互作用。而这种相互作用,则导致了RQC 组分蛋白质的降解,从而促进了PTGS 的发生。
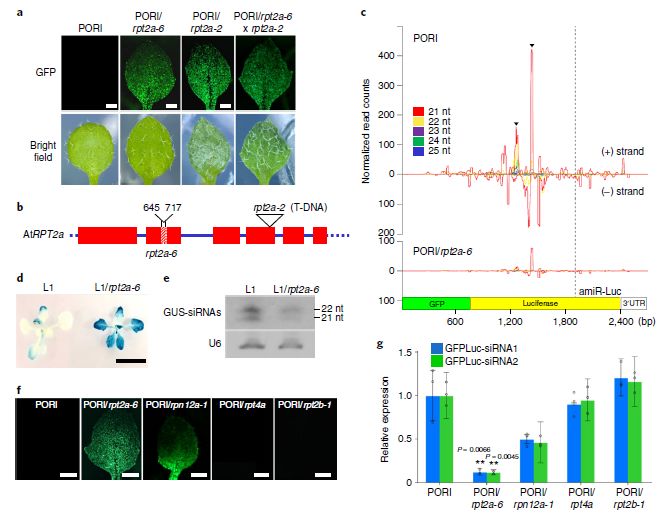
RPT2a 是产生转基因衍生的siRNA 所必需的
本文的研究主要揭示了26S 蛋白酶体亚基RPT2a 通过促进产生转基因衍生的siRNA 的积累进而增强了转基因的PTGS。RPT2a 能够与RQC 的组分相互作用,并下调RQC 组分蛋白质的水平。RQC 组分水平的下降则增强了PTGS,从而导致转基因的沉默。
总之,本文的研究为RQC 和PTGS建立了联系,使人们对植物体内RNA稳态的调控机制有了更清晰的认识。
参考文献:
【1】Schoenberg, D.R., and Maquat, L.E. (2012). Regulation of cytoplasmic mRNA decay. Nat Rev Genet 13, 246-259.
【2】Tsuzuki, M., Motomura, K., Kumakura, N., and Takeda, A. (2017). Interconnections between mRNA degradation and RDR-dependent siRNA production in mRNA turnover in plants. J Plant Res 130, 211-226.
【3】 Liu, L., and Chen, X.M. (2016). RNA Quality Control as a Key to Suppressing RNA Silencing of Endogenous Genes in Plants. Mol Plant 9, 826-836.
原文链接:
https://www.nature.com/articles/s41477-019-0546-1
来源:bioartplants BioArt植物
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3ODY3MDM0NA==&mid=2247493022&idx=1&sn=2047fdf429a37a0a3b09758ecdebf16c&chksm=fd737ff9ca04f6efa282e3f5e3ed3c0fe180242ba1b52cc4f268f93a1a6b621022d1f97d6386&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

【直播预告】转基因科普巡讲回来啦!聊聊转基因那些事
2016年全国植物生物学大会在湖北武汉召开
中国植物生理与植物分子生物学学会植物生物学女科学家分会第三届学术交流会成功举办
植物生理生态研究所科研人员发现植物抗虫调控新机制
第二届“分子植物”国际学术研讨会在清华大学成功召开
《生命科学》出版植物营养与人类健康专刊

转基因被误解的那些事:转基因食品会致癌?

华南植物园转基因烟草沉默机制研究获进展

利用高粱Rubisco小亚基在转基因水稻产生部分C4效果的高光合效率
从容看待转基因