科技工作者之家
加好友
科技工作者之家 2021-07-21
解析控制植物表型的各类编码和非编码因素是分子生物学研究的重要课题,也是高效遗传改良和分子设计育种的基础。
编码基因仅仅是决定植物表型的部分因素,基因组的非编码序列同样起着重要作用。植物基因组中绝大部分是由所谓的“垃圾序列”(重复序列)构成,编码区的占比很小。转座子是构成基因组重复序列的主要成分,它们像病毒一样在基因组中不断复制扩张,按其复制方式可分为逆转录转座子(Class I retrotransposons)和非逆转录转座子(Class II DNA transposons)。越来越多的研究表明,转座子往往导致基因组结构变异、序列突变、基因丢失、基因融合、新编码基因的形成,也通过提供各类非编码基因、新的调控序列或改变甲基化和染色质构象模式等影响基因的表达,转座子是控制植物表型变异中不可忽视的因素。除了直接的功能效应,转座子也与基因组大小有紧密关联,被认为是基因组变庞大的重要原因。
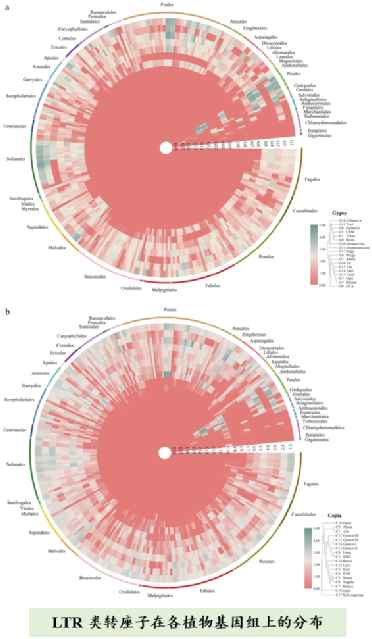
LTR 类转座子是植物基因组中占比最高的重复序列类型,它是逆转录转座子的一种。在玉米、小麦、松树等复杂基因组中,LTR 类转座子的比例超过或接近 70%,在其它植物基因组中占比也普遍超过了 50%。尽管 LTR 类转座子是基因组中的主导成分,起着显著的功能效应,然而由于不同物种中往往采用不同的注释算法,种间比较研究很难进行,这严重限制着对这类转座子扩张机制、功能效应的深入分析。
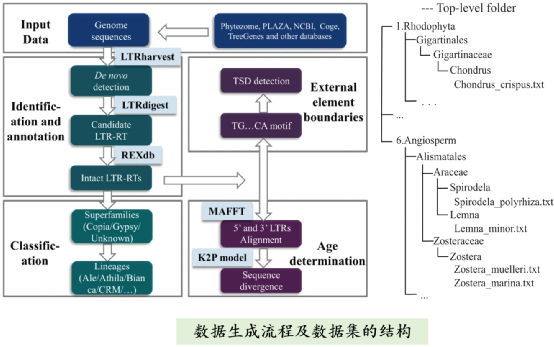
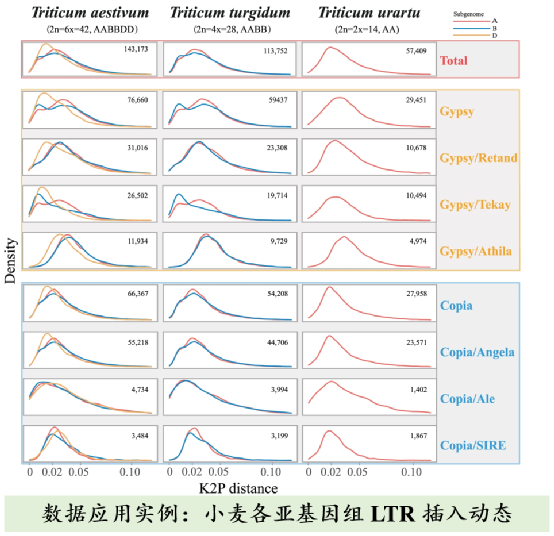
北京林业大学主导的研究团队,收集了已发布的植物基因组数据,整合先进的识别和注释算法,采用统一的策略完成了 300 种植物基因组的 LTR 注释。发布的信息包括:LTR 的基因组位置、所属亚类、LTR内编码区、TSD 序列、两端 LTR 序列的分化程度(LTR 的插入时间)等。该数据集可为后续 LTR 扩张及清除机制、LTR 转录及功能效应、LTR 形成基因重复、LTR 种间水平转移、LTR 对基因组大小变异作用等课题提供重要的基础,为解析松树等针叶树庞大基因组的构成和维持机制提供参考。
该研究以“A comprehensive annotation dataset of intact LTR retrotransposons of 300 plant genomes”为主题发表在重要的多学科期刊 Scientific Data 上。北京林业大学生物科学与技术学院、林木分子设计高精尖创新中心博士生周姗姗为论文的第一作者,博士生严雪梅、刘辉、徐杰、聂帅、贾凯华、焦思倩,博士后赵伟,西南林业大学赵友杰、张开福,加拿大UBC 大学 Yousry El Kassaby 和 Tongli Wang,加拿大 Université Laval 大学 Ilga Porth 等参与了该研究。北京林业大学毛建丰为该研究的通讯作者。
该研究得到国家自然科学基金(31670664)、北京林业大学“双一流”建设项目(2019XKJS0308)、北京林业大学基础科研业务费(2018BLCB08)等基金项目的支持。
来源:植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247519492&idx=2&sn=6bc620741e7ed52aed7c2316b371fb40
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
植物胁迫响应中非编码RNA间互作的研究进展
代号编码
2016年全国植物生物学大会在湖北武汉召开
中国植物生理与植物分子生物学学会植物生物学女科学家分会第三届学术交流会成功举办
2015上海辰山“药食同源与植物代谢”国际学术研讨会顺利召开
第二届“分子植物”国际学术研讨会在清华大学成功召开
植物所等发现植物生物钟的新调控因子
李新荣:沙漠书写草根论文
2013全国植物生物学大会

专刊征稿|植物钙生物学