科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-07-31
DNA甲基化可以被分为从头DNA甲基化和维持DNA甲基化两步。在植物中从头DNA甲基化的部位可以发生在对称性CpG、CpHpG(H = A、T 或者 C)和非对称性的CpHpHp等三种序列中。从头DNA甲基化转移酶酶DRM2主要负责催化CpG、CpHpG和CpHpHp序列的从头DNA甲基化过程,该基因是哺乳动物中DNMT3的同源基因。从头DNA甲基化转移酶DRM2通过RNA 介导 DNA 甲基化(RdDM) 途径来完成从头DNA甲基化。RdDM可以在转录水平抑制转座子和基因,并参与到植物生物和非生物胁迫、植株再生、植物生长和果实成熟发育等生物学调控过程。20多年来,经过全球各实验室的努力,已经鉴定了20多个蛋白参与RdDM途径,然而是否还有其他新的组分,仍不是很清楚!

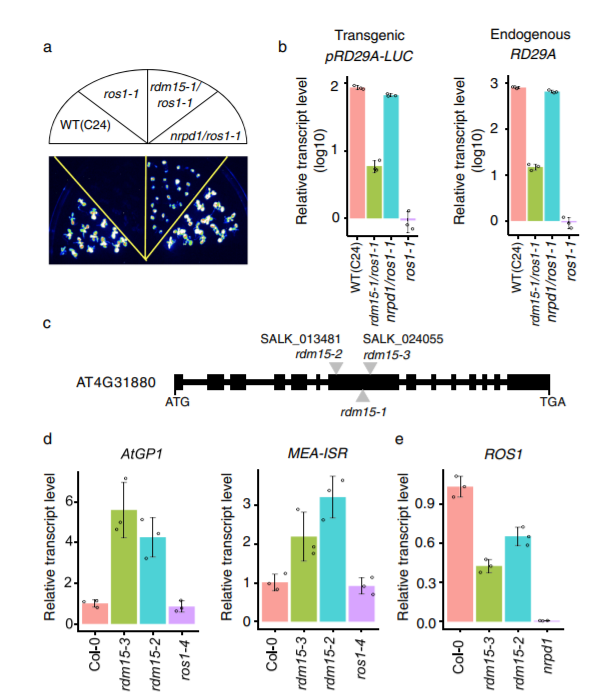
1. 该课题组通过正向遗传学筛选到了参与基因沉默的突变体rdm15,通过克隆发现是具有Tudordomain的基因RDM15功能缺失造成的。通过甲基化测序和DMR的分析发现,RDM15影响的甲基化位点是RdDM作用位点。在RDM15突变体中DNA甲基化水平的变化伴随着相同趋势的小干扰RNA水平的变化。研究结果确认了RDM15是RdDM途径中的一个组分。通过免疫沉淀和质谱联用的方法,进一步鉴定出RDM15是NRPE3B (PolV的亚基)的互作组分。通过BiFC和Split-LUC实验进一步确认了其互作关系。从而确认了RDM15是通过招募PolV来影响RDM15位点上的DNA甲基化。此外,该研究进一步发现RDM15可以特异性的结合H3K4me1。因此,该工作发现了一个新的H3K4me1识别蛋白RDM15,其通过招募PolV来参与RNA介导的DNA甲基化过程的新机制。
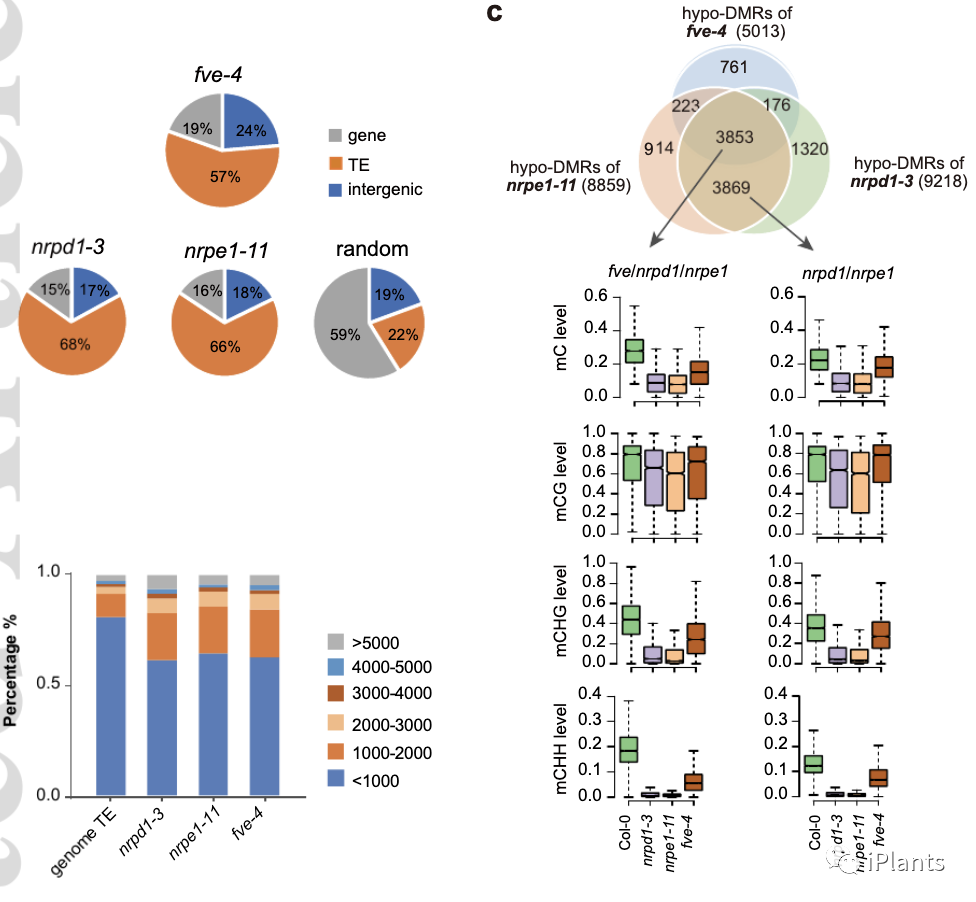
2. 该课题组进一步通过全基因组甲基化测序以及差异甲基化区域(DMRs) 分析发现,开花调控基因FVE能够影响部分RdDM通路调控位点的DNA甲基化水平。siRNA测序以及差异表达区域的分析说明FVE参与RdDM通路siRNA的调控,尤其是下游siRNA的累积。qRT-PCR实验分析说明FVE影响 PolV转录本的转录水平。此外FVE调节DNA甲基化水平的变化伴随着相同趋势的siRNA水平的变化。染色体免疫沉淀测序试验(ChIP-seq)进一步说明FVE倾向于结合RdDM通路下游调控位点,进而调节DNA甲基化。
更为重要的是,该研究通过FVE的免疫沉淀和质谱联用(IP-MS)IP-MS实验分析发现FVE与RDM15蛋白能够相互作用,通过酵母双杂交实验(Y2H)和Split-LUC实验进一步确认了二者的互作关系。利用RDM15的全基因甲基化数据以及DMR分析,证明FVE和RDM15共同调节部分RdDM位点的DNA甲基化水平。FVE与RDM15的研究分析进一步确认FVE参与RdDM通路下游功能调节的分子机制。
来源: iPlants
原文链接:http://mp.weixin.qq.com/s?__biz=MzUzNzczODE4Mg==&mid=2247519091&idx=2&sn=dec9179e333fa8ff81cac20eea0393cd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

DNA“甲基化指纹”鉴定出91种肿瘤
Nat Plants:植物如何防晒?抑制DNA甲基化

植物逆境中心朱健康院士在植物DNA主动去甲基化领域取得新进展

新研究揭开人类头发曲直的秘密

Science:揭示蛋白QSER1保护DNA甲基化谷免受新生甲基化
昆明植物所在DNA甲基化调控竹笋快速生长研究中获进展

Nat Plants:NIBS何新建组发现植物DNA甲基化动态调控的新机制
分子植物卓越中心发表植物DNA去甲基化的机理和功能综述文章
合成生物学: 一个用来控制转基因生物的内置毁灭开关

DNA机器人一小步 癌症治疗一大步