科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-09-09
传染性海绵状脑病(又称prion疾病)是由朊病毒蛋白(PrP)在体内发生错误折叠所导致的一种慢性并可在多种哺乳动物之间传播的致死性神经退行性疾病【1-4】。1997年诺贝尔生理或医学奖得主Prusiner S. B.最先描述了朊病毒(prions),即蛋白质感染因子【1】。根据发病特征,Prion疾病分为散发性、家族遗传性和感染性三种类型,家族遗传性prion疾病的发生与朊蛋白基因PRNP的突变密切相关,已发现引起家族遗传性prion疾病的PrP突变位点共33个,包含42种家族遗传性prion疾病【1,4】。这些不同的病理突变体可以形成朊病毒不同的株型,引起不同的临床症状,导致不同疾病的发生,如克雅氏病(Creutzfeldt-Jakob disease, CJD)、致死性家族失眠症(fatal familial insomnia, FFI)和格斯特曼氏综合征(Gerstmann-Sträussler-Scheinker disease, GSS)等。
Prion疾病给人类健康带来了重大威胁,自从Prusiner S. B.在1982年发现朊病毒之后,科学家们在试图阐释谜一般的朊病毒株型(prion strains)形成分化机制和测定朊病毒蛋白纤维结构两方面付出了巨大的努力【5-10】。
武汉大学梁毅团队和中国科学院生物与化学交叉研究中心刘聪团队长期合作聚焦朊病毒蛋白病理聚集的结构基础及致病分子机制研究,前期工作阐释了全长野生型朊蛋白纤维的组装机制,首次在原子水平上揭示了PrP由细胞型朊蛋白(PrPC)向病理型朊病毒蛋白(PrPSc)结构转变的机制【10】,启示不同的病理突变体在调节朊病毒蛋白构象转化中可能发挥着不同的作用(详见BioArt报道:NSMB | 梁毅/刘聪合作团队解析朊病毒蛋白纤维冷冻电镜结构)。然而,病理突变体是否以及是如何改变朊病毒蛋白结构的?不同的朊病毒蛋白病理突变体如何引起不同的病理临床症状?这些都是领域内尚未解决的重要科学问题。
2021年9月8日,武汉大学生命科学学院梁毅团队和中国科学院上海有机所生物与化学交叉研究中心刘聪团队在Science Advances上以长文(Research Article)形式在线发表了题为“Genetic prion disease—related mutation E196K displays a novel amyloid fibril structure revealed by cryo-EM”的最新研究成果,首次在原子水平上解析了朊病毒蛋白病理突变体E196K纤维的高分辨率冷冻电镜结构(图1和图2),揭示了朊病毒蛋白病理聚集多态性的分子机制(图3)。
图1:E196K淀粉样纤维的冷冻电镜结构
为了进一步揭示E196K淀粉样纤维的原子结构模型,研究组发现E196K纤维核心主要由其C端的175-217组成,E196K纤维形成了一个与野生型PrP纤维完全不同的结构。E196K纤维每股原纤维的核心包含5个β-折叠结构(从β1到β5)。在E196K聚集过程中,两股原纤维通过来自两个亚基的Lys194和Glu207之间以及Lys196和Glu200之间形成的4对盐桥发生相互作用,形成一个长的之字形相互作用界面,该界面同时包含亲水氨基酸和疏水氨基酸(图2)。
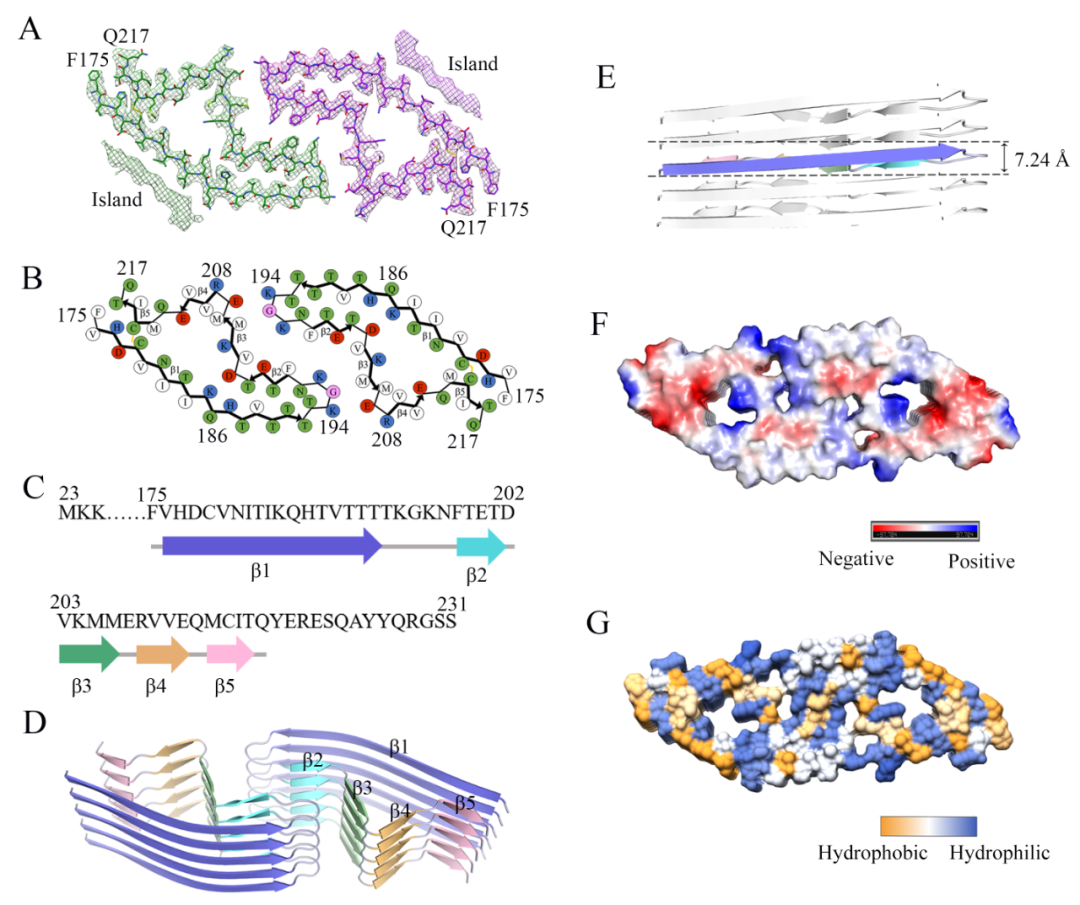
图2:E196K淀粉样纤维的原子结构模型
同时,对于E196K纤维每个亚基包含一个亲水腔这样新奇的结构,研究团队对于其病理学意义进行了探索,发现E196K纤维也能和刚果红结合,而且其蛋白酶抗性和构象稳定性均显著低于野生型PrP纤维。这项工作的实验结果表明,在E196K错误折叠过程中,E196K PrPC C端的两个α-螺旋转变成E196K纤维的五个β-折叠结构,Cys179和Cys214之间的二硫键起到了稳定淀粉样纤维结构的作用(图3)。
这项工作的研究结果首次揭示了E196K纤维和野生型PrP纤维之间的关键结构差异:(1) E196K纤维形成的是亲水空腔,而野生型PrP纤维则形成了疏水空腔;(2) E196K纤维形成的是既包含亲水又包含疏水的原纤维界面,而野生型PrP纤维则形成了短的亲水原纤维界面;(3) E196K纤维形成了5个β-折叠结构,而野生型PrP纤维形成了6个β-折叠结构;(4) E196K纤维相较野生型纤维具有更不稳定的构象(图3)。
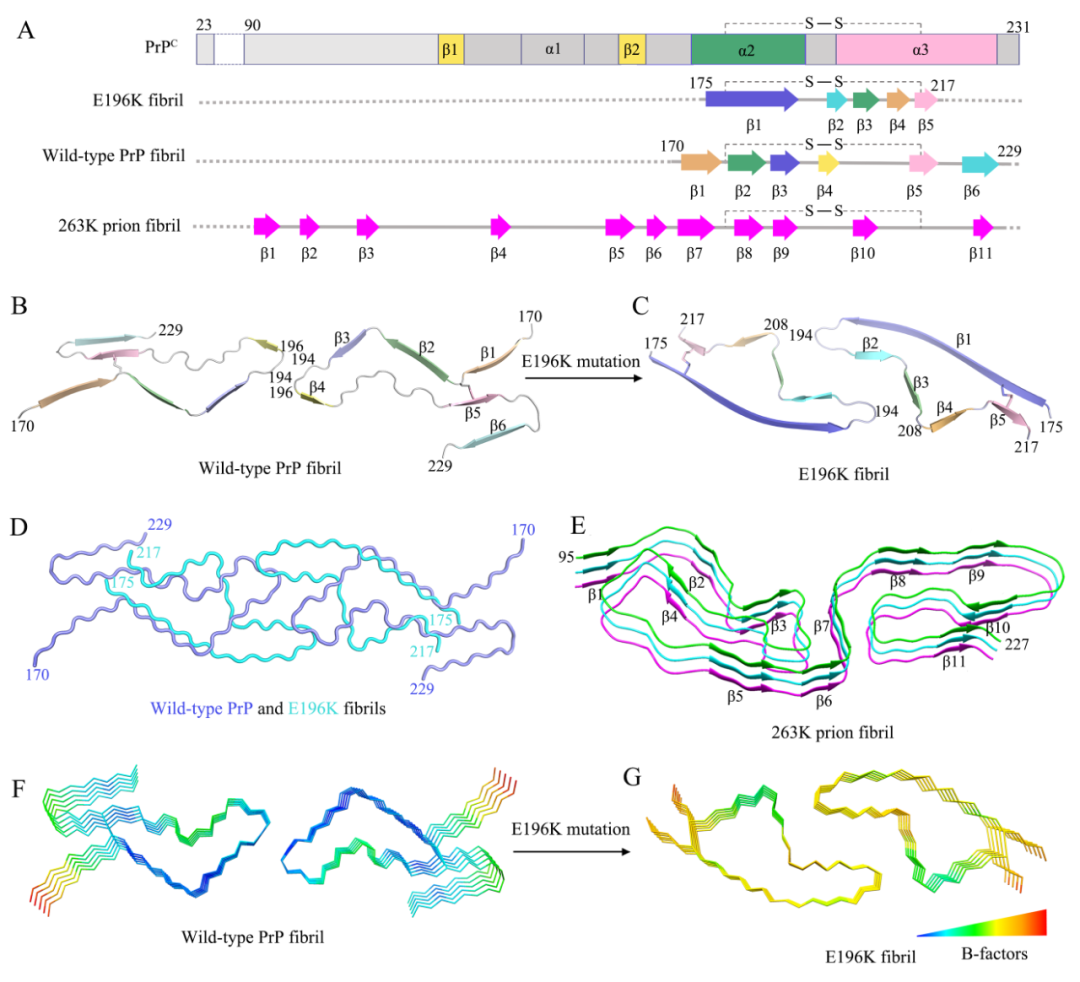
图3:人PrPC和E196K纤维、野生型PrP纤维以及263K朊病毒纤维结构比较
该研究首次在原子水平上揭示了朊病毒株型形成分化的机制,证明不同株型的家族性病理突变能够破坏朊病毒蛋白纤维中重要的相互作用(例如盐桥),使得这些病理突变体纤维结构形成了完全不同于野生型朊病毒蛋白纤维的构象,因此不同的病理突变体在调节朊病毒蛋白构象转化中可能发挥着不同的作用,并使得发展新的基于PrP纤维结构的prion疾病治疗药物成为可能,为PrPSc结构与致病功能的深入研究和回答“朊病毒株型是怎么形成的”奠定了分子基础,具有重要的科学意义。
梁毅教授和刘聪教授为通讯作者,梁毅课题组博士后王利强、刘聪课题组博士研究生赵焜和梁毅课题组博士研究生袁菡烨为共同第一作者,华中农业大学生命科学技术学院殷平教授等参与了该论文的研究工作。
原文链接:
https://www.science.org/doi/10.1126/sciadv.abg9676
参考文献
1. S. B. Prusiner. Prions. Proc. Natl. Acad. Sci. USA. 1998, 95, 13363-13383.
2. C. Soto. Prion hypothesis: the end of the controversy? Trends Biochem. Sci. 2011, 36, 151-158.
3. S. B. Prusiner. A unifying role for prions in neurodegenerative diseases. Science 2012, 336, 1511-1513.
4. C. Scheckel, A. Aguzzi. Prions, prionoids and protein misfolding disorders. Nat. Rev. Genet. 2018, 19, 405-418.
5. R. Diaz-Espinoza, C. Soto. High-resolution structure of infectious prion protein: the final frontier. Nat. Struct. Mol. Biol. 2012, 19, 370-377.
6. E. Vázquez-Fernández, M. R. Vos, P. Afanasyev, L. Cebey, A. M. Sevillano, E. Vidal, I. Rosa, L. Renault, A. Ramos, P. J. Peters, J. J. Fernandez, M. van Heel, H. S. Young, J. R. Requena, H. Wille. The structural architecture of an infectious mammalian prion using electron cryomicroscopy. PLoS Pathog. 2016, 12, e1005835.
7. G. Spagnolli, M. Rigoli, S. Orioli, A. M. Sevillano, P. Faccioli, H. Wille, E. Biasini, J. R. Requena. Full atomistic model of prion structure and conversion. PLoS Pathog. 2019, 15, e1007864.
8. A. Aguzzi, E. De Cecco. Shifts and drifts in prion science. Science 2020, 370, 32-34.
9. C. Glynn, M. R. Sawaya, P. Ge, M. Gallagher-Jones, C. W. Short, R. Bowman, M. Apostol, Z. H. Zhou, D. S. Eisenberg, J. A. Rodriguez. Cryo-EM structure of a human prion fibril with a hydrophobic, protease-resistant core. Nat. Struct. Mol. Biol. 2020, 27, 417-423.
10. L. Q. Wang, K. Zhao, H. Y. Yuan, Q. Wang, Z. Guan, J. Tao, X. N. Li, Y. Sun, C. W. Yi, J. Chen, D. Li, D. Zhang, P. Yin, C. Liu, Y. Liang. Cryo-EM structure of an amyloid fibril formed by full-length human prion protein. Nat. Struct. Mol. Biol. 2020, 27, 598-602.
本文转载自公众号“BioArt”(BioGossip)
中国生物物理学会官方订阅号,为BSC会员及生物物理领域专业人士服务。
投稿及授权请联系:bscoffice@bsc.org.cn。
微信号:BSC-1979

来源:BSC-1979 中国生物物理学会
原文链接:http://mp.weixin.qq.com/s?__biz=MzA4NjE1NTQ5Mg==&mid=2649510577&idx=2&sn=2a57be987e7bee1476d56053776cb9f4
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn