科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-05-07
来源:BioArt
撰文 | 十一月
责编 | 兮
在大多数的物种减数分裂过程中,同源染色体必须重组才能确保精准分裂【1】。如果重组过程是随机分布的,长度较短的染色体就有可能会发生错配的危险,因此减数分裂过程中引发的DNA双链断裂在染色体上并不是随机分布的【2】。虽然目前对于调控双链断裂的时间、定位以及数目的调节过程相关通路已有一些研究,但是双链断裂过程在染色体上的非随机分布是如何控制的研究还很不清楚。
2020年5月6日,美国斯隆-凯瑟琳癌症研究所Scott Keeney与Hajime Murakami研究组在Nature发文题为Multilayered mechanisms ensure that short chromosomes recombine in meiosis,对减数分裂过程中染色体是如何确保准确重组过程中多重保险机制进行了研究和总结。

同源染色体上至少含有一个DNA双链断裂位点就可促进重组过程的发生。在酿酒酵母中的研究表明,如果DNA双链断裂过程是随机分布的话,三条最短的染色体chr1、chr3和chr6就可能会存在错配和丢失的风险。同时前人的研究发现,染色体的长度与染色体互换的密度【3】、DNA双链断裂发生的密度【4】以及DNA双链断裂蛋白的结合【5】呈现明显的负相关性,说明这其中存在一定的调控机制来缓解减数分裂过程中较短的染色体错配和丢失的风险。
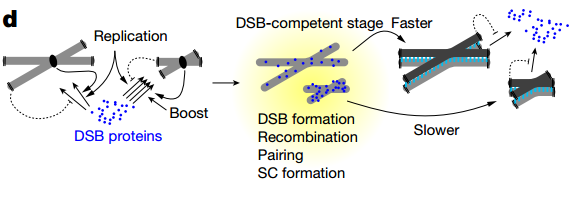
减数分裂过程中出现的DNA双链断裂过程是由Spo11和附属的DNA双链断裂相关的蛋白产生的,这其中就包括Rec114和Mer2。Rec114和Mer2会聚集在减数分裂的染色体上,并且这两种蛋白在真核生物中是广泛存在的【5,6】。Rec114和Mer2是如何调控减数分裂过程中重组过程的正常进行的呢?作者们首先对标签标记的Rec114和Mer2相关已发表的【7】以及新的减数分裂过程中不同时间点的ChIP-seq结果进行了整合和分析,从中得到了两个非常重要的结论:1)此两种蛋白在较短的染色体上富集存在;2)这两种蛋白在减数分裂的早期和晚期阶段不同时间点存在不同的密度分布。这一结果说明,在减数分裂过程中早期和晚期存在各自不同的调节机制。
总而言之作者们发现,在减数分裂过程中较短的染色体上,Rec114在早期阶段富集而在较晚的阶段解离下来。而且,Rec114在每条染色体上持续存在时间与染色体长度呈反比。并且作者们进一步确认了DNA双链断裂相关的蛋白通过染色体长度依赖的方式调控其富集和解离的过程,从而确保在非常短的染色体上具有大量的DNA双链断裂蛋白的存在。
那么Rec114蛋白在染色体上富集过程是如何被调控的呢?先前的研究表明Rec114在染色质上的结合受到复制过程的调控【7】。但是作者们发现,仅复制过程不足以对此Rec114在不同长度染色体上的富集程度不同进行解释,可能还存在其他的调控机制。为了找出可能参与其中的调控机制,作者们对ChIP结果进行了色彩编码图谱分析,发现在靠近着丝粒的区域DNA双链断裂相关蛋白的结合密度更高,而靠近端粒的区域结合趋势则与之相反。之前也有报道发现端粒存在延滞DNA双链断裂形成的效应【8】。因此,对于大多数的染色体来说,DNA双链断裂相关的蛋白是否在减数分裂前期结合在染色体上,同时受到复制时间以及与着丝粒或者端粒之间距离的影响。另外,作者们还发现,较短的染色体例如chr1本身具有促进早期Rec114蛋白富集结合的特点,而这一特点作者们通过对chr1染色体长度的操纵发现并不会有什么明显的影响。最后,由于减数分裂染色体会形成轴向结构锚定染色质环,作者们通过对轴向蛋白的敲除确认了环-轴结构(Loop-axis structure)对于较短染色体形成DNA双链断裂的潜在特性也具有一定的贡献。
总的来说,Scott Keeney与Hajime Murakami研究组的工作发现在酵母减数分裂过程中较短染色体上通过招募Rec114和Mer2调控DNA双链断裂的精准发生,从而确保较短染色体不会发生错配。同源染色体的相互结合调节减数分裂过程中Rec114和Mer2的解离,而复制时间以及与着丝粒和端粒距离则调控Rec114和Mer2在较短染色体上的招募和富集(图1)。该工作通过对酵母在减数分裂过程中较短染色体重组过程的 “多重安保机制”进行研究,为细胞如何确保每条染色体正确分配提供了重要见解。
制版人:珂
参考文献
1. Hunter, N. Meiotic Recombination: The Essence of Heredity. Cold Spring Harbor perspectives in biology 7, doi:10.1101/cshperspect.a016618 (2015).
2. Keeney, S., Lange, J. & Mohibullah, N. Self-organization of meiotic recombination initiation: general principles and molecular pathways. Annu Rev Genet 48, 187-214, doi:10.1146/annurev-genet-120213-092304 (2014).
3. Mancera, E., Bourgon, R., Brozzi, A., Huber, W. & Steinmetz, L. M. High-resolution mapping of meiotic crossovers and non-crossovers in yeast. Nature 454, 479-485, doi:10.1038/nature07135 (2008).
4. Pan, J. et al. A hierarchical combination of factors shapes the genome-wide topography of yeast meiotic recombination initiation. Cell 144, 719-731, doi:10.1016/j.cell.2011.02.009 (2011).
5. Panizza, S. et al. Spo11-accessory proteins link double-strand break sites to the chromosome axis in early meiotic recombination. Cell 146, 372-383, doi:10.1016/j.cell.2011.07.003 (2011).
6. Robert, T. et al. The TopoVIB-Like protein family is required for meiotic DNA double-strand break formation. Science (New York, N.Y.) 351, 943-949, doi:10.1126/science.aad5309 (2016).
7. Murakami, H. & Keeney, S. Temporospatial coordination of meiotic DNA replication and recombination via DDK recruitment to replisomes. Cell 158, 861-873, doi:10.1016/j.cell.2014.06.028 (2014).
8. Borde, V., Goldman, A. S. & Lichten, M. Direct coupling between meiotic DNA replication and recombination initiation. Science (New York, N.Y.) 290, 806-809, doi:10.1126/science.290.5492.806 (2000).
来源:BioGossip BioArt
原文链接:https://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652484082&idx=6&sn=ef83aaaab078484d5ab13f5fd47091aa&chksm=84e22246b395ab500b19f577c10d930750fae03a993c864718a0f87399c5d85215229ba23929#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

生殖细胞减数分裂百年之谜获解

Sci Adv :减数分裂前期向中期转变关键调控蛋白
减数分裂起始研究取得进展
演化:“最奇怪动物”身份揭秘
合成生物学: 一个用来控制转基因生物的内置毁灭开关
遗传发育所揭示氮营养与植物减数分裂起始的联系
植物前减数分裂过程中phasiRNA形成的新机制!
植物减数分裂着丝粒配对研究取得新进展
遗传发育所揭示减数分裂同源重组保障新机制
环境友好的农林害虫生态调控与生物防治技术——2017高级研修班总结