科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-05-18
来源:BioArt植物
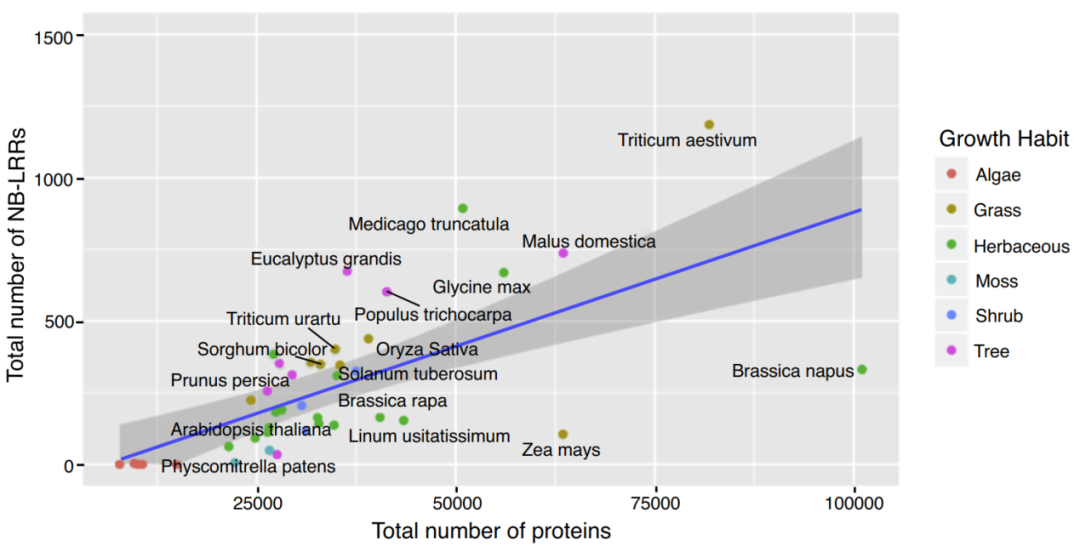 不同植物基因组中NLR的数量比较【3】
不同植物基因组中NLR的数量比较【3】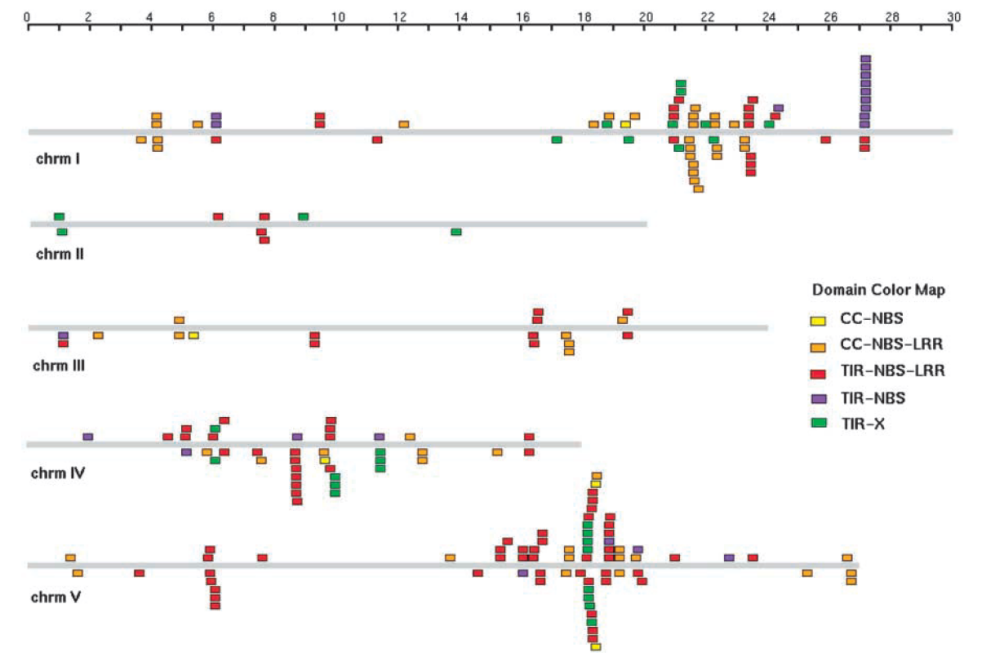 拟南芥基因组中许多NLR基因成簇分布【1】
拟南芥基因组中许多NLR基因成簇分布【1】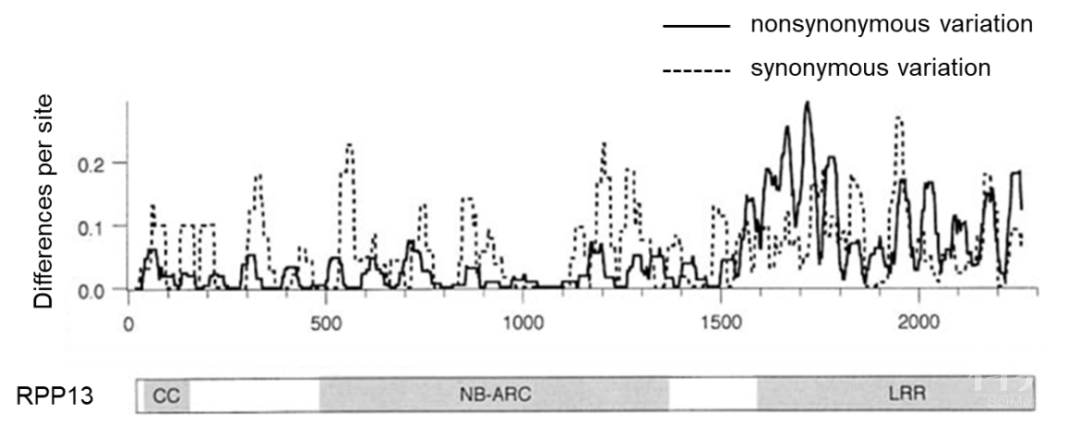 拟南芥RPP13中的变异分布情况【5】
拟南芥RPP13中的变异分布情况【5】 NLR整合其他结构域示例及整合结构域分布词云【8-9】
NLR整合其他结构域示例及整合结构域分布词云【8-9】参考文献
【1】Meyers, B. C., Kozik, A., Griego, A., Kuang, H., & Michelmore, R. W. (2003). Genome-wide analysis of NBS-LRR–encoding genes in Arabidopsis. The Plant Cell, 15(4), 809-834.
【2】Zhou, T., Wang, Y., Chen, J. Q., Araki, H., Jing, Z., Jiang, K., ... & Tian, D. (2004). Genome-wide identification of NBS genes in japonica rice reveals significant expansion of divergent non-TIR NBS-LRR genes. Molecular Genetics and Genomics, 271(4), 402-415.
【3】Baggs, E., Dagdas, G., & Krasileva, K. V. (2017). NLR diversity, helpers and integrated domains: making sense of the NLR IDentity. Current opinion in plant biology, 38, 59-67.
【4】Yang, S., Zhang, X., Yue, J. X., Tian, D., & Chen, J. Q. (2008). Recent duplications dominate NBS-encoding gene expansion in two woody species. Molecular Genetics and Genomics, 280(3), 187-198.
【5】Rose, L. E., Bittner-Eddy, P. D., Langley, C. H., Holub, E. B., Michelmore, R. W., & Beynon, J. L. (2004). The maintenance of extreme amino acid diversity at the disease resistance gene, RPP13, in Arabidopsis thaliana. Genetics, 166(3), 1517-1527.
【6】Mondragón-Palomino, M., Meyers, B. C., Michelmore, R. W., & Gaut, B. S. (2002). Patterns of positive selection in the complete NBS-LRR gene family of Arabidopsis thaliana. Genome research, 12(9), 1305-1315.
【7】Wicker, T., Yahiaoui, N., & Keller, B. (2007). Illegitimate recombination is a major evolutionary mechanism for initiating size variation in plant resistance genes. The Plant Journal, 51(4), 631-641.
【8】Kroj, T., Chanclud, E., Michel‐Romiti, C., Grand, X., & Morel, J. B. (2016). Integration of decoy domains derived from protein targets of pathogen effectors into plant immune receptors is widespread. New Phytologist, 210(2), 618-626.
【9】Sarris, P. F., Cevik, V., Dagdas, G., Jones, J. D., & Krasileva, K. V. (2016). Comparative analysis of plant immune receptor architectures uncovers host proteins likely targeted by pathogens. BMC biology, 14(1), 8.
来源:bioartplants BioArt植物
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3ODY3MDM0NA==&mid=2247495299&idx=1&sn=c9e376995192d051e54afdad8141fdda&chksm=fd7374e4ca04fdf27b5a6ba7c44f198842ed654b1bc51d71b52cc2f811494e2654a6ab40d69c&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn