科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-06-09
来源:顶尖植物科学研究
2020年6月8日,中国科学院分子植物科学卓越创新中心上海植物逆境生物学研究中心朱健康课题组于Nature Plants杂志在线发表了题为“Epigenetic memory marks determine epiallele stability at loci targeted by de novo DNA methylation”的研究论文。

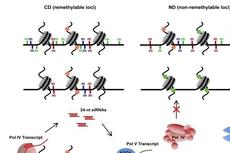
通常认为,从头RNA指导的DNA甲基化(RdDM)途径靶向的基因组区域的DNA甲基化变化是不稳定的。在这里,我们表明,在RdDM目标拟南芥能是否有再次甲基化NRPD1功能的恢复后的基础上分为两组nrpd1突变植物-可甲基化位点和不可甲基化位点。与可甲基化的位点相反,不可甲基化的位点在组蛋白H3(H3K4me3)的Lys 4处含有较高水平的三甲基化的常色标记,这会干扰RdDM分子机制的募集以及在组蛋白H3的Lys 18处的乙酰化。(H3K18ac),有助于募集DNA脱甲基酶ROS1拮抗RdDM。在这里,使用CRISPR–dCas9–TET1的靶向甲基化擦除,我们证明了甲基化CG(mCG)和mCHG(其中H代表A,C或T)是将RdDM机械靶向可甲基化位点所需的记忆标记。我们的结果表明,组蛋白和DNA甲基化标记对于确定RdDM靶基因座形成稳定等位基因的能力至关重要。
中科院分子植物科学卓越创新中心上海植物逆境生物学研究中心研究员朱健康为该论文的通讯作者,博士生李静雯为第一作者。该研究得到中科院战略性先导科技专项的支持。
来源:gh_14c52a57b006 顶尖植物科学研究
原文链接:https://mp.weixin.qq.com/s?__biz=MzI4MjkwOTE3OQ==&mid=2247490620&idx=3&sn=c55f035a5d4f3fc9edb66f3aa13e929d&chksm=eb939ca8dce415be7ed7d36eac654f7e9e09456313463d1b6864b50a5d1ba93281bbcfef9971#rd
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

Science:揭示蛋白QSER1保护DNA甲基化谷免受新生甲基化

DNA“甲基化指纹”鉴定出91种肿瘤
第二届“药食同源与植物代谢”国际学术研讨会在沪举行
李新荣:沙漠书写草根论文
Nat Plants:植物如何防晒?抑制DNA甲基化

当DNA甲基化遇上RNA甲基化:果实成熟的表观遗传调控 | Genome Biology
昆明植物所在DNA甲基化调控竹笋快速生长研究中获进展
郎曌博团队发表“植物DNA去甲基化的机理和功能”综述文章

研究发现植物DNA主动去甲基化新机制
DNA甲基化跨代遗传研究取得进展