科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-04-25
来源:BioArt
撰文:木兰之枻
当下以CRISPR-Cas9技术为基础的基因编辑技术面临的最大问题便是其脱靶风险。为此研究者在技术诞生初期便尝试用各种方法分析该技术在基因组水平的脱靶情况。
2013年,Feng Zhang & Gang Bao团队和Jeffry D. Sander & J. Keith Joung团队通过突变sgRNA序列的不同位点,在sgRNA/DNA杂交链的不同位置引入错配,以检测CRISPR系统对错配的容忍度【1,2】;David R. Liu & Jennifer A. Doudna团队的思路则殊途同归,通过体外筛选策略,检测sgRNA对携带有不同突变的DNA靶序列的切割,检测CRISPR系统的脱靶风险【3】。此时的脱靶检测方法尚处于摸索阶段,尚不具备在全基因组水平分析脱靶风险的能力。
2014-2015年,多个研究组开发出了可在全基因组范围内分析脱靶风险的方法如Guide-seq、Digenome-seq以及HTGTS,其基本原理是用Cas9 sgRNP在体外条件下切割基因组,之后通过捕获分析DNA双链断裂(DSBs)的位置判断脱靶位点【4-6】。在此基础上,研究者推出了相同原理的CIRCLE-seq和SITE-seq两种方法,改善了全基因组脱靶分析的特异性和灵敏度【7,8】。到2018年,研究者更是以CIRCLE-seq为基础,推出了“在体脱靶验证系统” VIVO(verification of in vivo off-targets)。该系统中,CRISPR-Cas9脱靶位点的检测依然是检测Cas9体外切割效应的CIRCLE-seq,通过CIRCLE-seq发现了潜在脱靶位点后,研究者在动物体内分析这些潜在脱靶位点的突变情况【9】。因此从严格意义上讲,该方法并不是严格意义上的“在体脱靶验证系统”。
2019年3月1日,中科院神经科学研究所杨辉研究团队等与中科院遗传发育所高彩霞研究团队在Science杂志上发表了两篇研究论文,通过精巧的设计和全基因组测序对CRISPR/Cas9以及单碱基基因编辑系统在小鼠胚胎及水稻中的脱靶进行了精准检测,发现良好设计的CRISPR/Cas9系统和ABE没有明显的脱靶效应,但BE3系统则导致大量的的脱靶性单核苷酸突变出现【10,11】(详见:专家热评Science丨杨辉组/高彩霞组“背靠背”首次发现单碱基编辑系统存在严重脱靶效应
)。这两篇文章没有去尝试预测脱靶位点,而是直接通过全基因组测序来无差别的分析脱靶情况。
2019年4月19日,来自加州大学伯克利分校的Jacob E. Corn(现已入职苏黎世联邦理工学院ETH Zürich)在Science上发表了题为Unbiased detection of CRISPR off-targets in vivo using DISCOVER-Seq 的研究成果,开发出了全新的CRISPR-Cas9基因编辑的全基因组脱靶效应的高效在体检测系统——Discover-Seq(discovery of in situ Cas off-targets and verification by sequencing)。研究者除了在iPS细胞和小鼠模型中测试了新方法的可靠性之外,还与上文提到的VIVO系统进行了对比分析,证实了新方法DISCVOER-Seq的优越性。

DISCOVER-Seq不同于之前的方法VIVO,其对脱靶位点的分析并不依赖于体外切割,而是实实在在的细胞内检测。该方法的原理在于,当Cas9在细胞内切割基因组引入DSBs之后,细胞会通过损伤修复途径对此加以修复,该过程中会有DNA修复蛋白结合于Cas9导致的DSBs位置。通过ChIP-Seq技术,研究者首先对Cas9靶位点处的多种DNA修复蛋白的分布进行了分析,发现MRE11的分布与Cas9在靶位点处的切割关系最为密切。考虑到MRE11表达的广谱性和商用抗体的有效性,通过分析Cas9发挥作用后MRE11在基因组上的分布便可有效的检测出潜在的脱靶位点(下图)。
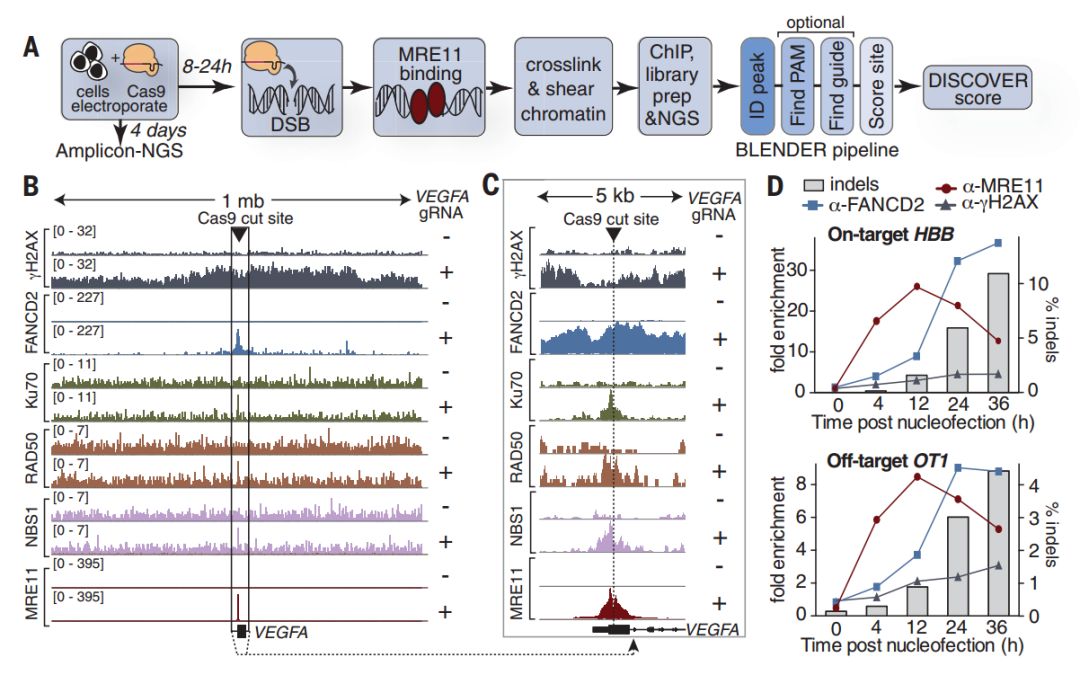
整个DISCOVER-Seq系统的检测不需要进行体外的基因组切割,只需要在细胞/动物体内的Cas9发挥作用引入DSBs后的合适窗口期,通过ChIP-Seq分析MRE11在基因组上的分布,便可以识别出脱靶位点。研究者首先在人源的K562细胞系和小鼠B16-F10细胞系中对新方法的有效性进行了确认,并与GUIDE-Seq进行了对比,指出DISCOVER-Seq虽在灵敏性上略有不如,但在准确性上较胜一筹。
之后研究者还在iPS细胞中对DISCVOER-Seq的适用性进行了验证,而GUIDE-Seq由于需要转染双链DNA寡核苷酸链(dsODN)作为标签而呈现出细胞毒性,无法用于iPS细胞内的脱靶分析。此处研究者选取的是CMT患者来源的iPS细胞,患者发病是由于其HSPB1基因的显性负效应(dominant-negative)杂合突变。研究者针对HSPB1的野生型位点和突变位点分别设计了特异性的sgRNA(WT-sgRNA vs Mut-sgRNA),并对两种sgRNA的特异性进行了比较分析,发现iPS细胞中Mut-sgRNA的特异性显著高于WT-sgRNA。基因编辑效率分析发现针对WT-sgRNA在突变位点同样有较高的编辑效率(~30%);而Mut-sgRNA在野生型位点的突变效率显著降低(~7%)。以上的结果证实了DISCVOER-Seq在脱靶分析上的可靠性,也表明在针对患者来源细胞的临床前分析中DISCOVER-Seq的前景令人期待。
最后,研究者在小鼠的肝脏中针对PCSK9基因检测了该系统的准确性,并与2018年Nature报道的VIVO系统进行了对比。2018年的VIVO系统发现了接近3000个潜在的脱靶位点,之后根据优先级挑选的45个验证位点中,19/45有显著的脱靶突变(发生率最低为0.13%,最高则为41.9%)。而DISCOVER-Seq系统则发现了36个潜在的脱靶位点,其中27个位点可以被扩增进行后续的验证,27/27均有显著的脱靶突变(发生率最低为0.9%,最高则为78.1%)。其中17/27个位点并未被VIVO系统识别为高风险脱靶位点。
作者认为,DISCOVER-Seq方法真正实现了在体全基因组范围内分析CRISPR系统的脱靶风险,且操作步骤得到了简化,在准确性方面也要明显优于VIVO系统,因此在临床应用上或有更大的可能。
总体而言,与2018年的VIVO系统相比,该文在方法的创新性上有很大的进步。虽然其检测脱靶的灵敏度上或有不如,但其操作的简捷性和分析的准确性上优势明显,因此或更有助于推动基因编辑从基础研究到临床的转化。
原文链接:
https://science.sciencemag.org/content/364/6437/286
制版人:半夏
参考文献
1.Fu, Y. et al. High-frequency off-target mutagenesis induced by CRISPR-Cas nucleases in human cells. Nat Biotechnol 31, 822-826 (2013).
2. Hsu, P.D. et al. DNA targeting specificity of RNA-guided Cas9 nucleases. Nat Biotechnol 31, 827-832 (2013).
3.Pattanayak, V. et al. High-throughput profiling of off-target DNA cleavage reveals RNA-programmed Cas9 nuclease specificity. Nat Biotechnol 31, 839-843 (2013).
4.Tsai, S.Q. et al. GUIDE-seq enables genome-wide profiling of off-target cleavage by CRISPR-Cas nucleases. Nat Biotechnol 33, 187-197 (2015).
5.Frock, R.L. et al. Genome-wide detection of DNA double-stranded breaks induced by engineered nucleases. Nat Biotechnol 33, 179-186 (2015).
6.Kim, D. et al. Digenome-seq: genome-wide profiling of CRISPR-Cas9 off-target effects in human cells. Nat Methods 12, 237-243, 231 p following 243 (2015).
7.Tsai, S.Q. et al. CIRCLE-seq: a highly sensitive in vitro screen for genome-wide CRISPR-Cas9 nuclease off-targets. Nat Methods 14, 607-614 (2017).
8.Cameron, P. et al. Mapping the genomic landscape of CRISPR-Cas9 cleavage. Nat Methods 14, 600-606 (2017).
9.Akcakaya, P. et al. In vivo CRISPR editing with no detectable genome-wide off-target mutations. Nature 561, 416-419 (2018).
10.Zuo, E. et al. Cytosine base editor generates substantial off-target single-nucleotide variants in mouse embryos.Science 364, 289-292 (2019).
11.Jin, S. et al. Cytosine, but not adenine, base editors induce genome-wide off-target mutations in rice. Science 364, 292-295 (2019).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652469130&idx=3&sn=c9995fb1171d9bb55043bccb6519dd4f&chksm=84e2e43eb3956d2809f8795cb3d82f8e8aac6a2b186da29d9b456b0024689bd513b940875a45&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
Prodigal:原核基因识别和翻译起始位点鉴定
唾液可检测天赋基因?专家:两三个基因点位“剧透”不了人生

中国科学家率先培育出带有基因剪刀的工具猪

蚊子怕怕了,利用转基因能破坏它们生育能力,必死无疑
揭秘基因编辑技术
5G商用进入倒计时,国产手机能否借机赶超苹果三星




一次转染同步编辑33个基因位点 抗病毒人类细胞系制备迈出第一步

基因编辑临床试验10原则出台
科技日报四问“基因编辑婴儿”

基因编辑立法箭在弦上