科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-05-04
来源:BioArt
在真核生物中,染色质在细胞核内相互缠绕、折叠、弯曲形成十分复杂的三维空间构象,这种高度结构化的空间构象即为三维基因组结构。诸多研究表明,基因组的空间结构与细胞中基因调控、DNA复制、转录以及染色体异位密切相关【1】。因此,三维基因组结构的解析对于染色体折叠机制的理解以及细胞内调控机理的研究具有非常重要的意义。近年来,基于染色体构象捕获(3C)技术来鉴定染色质相互作用图谱的方法发展十分迅速,其中与高通量测序技术相结合的Hi-C技术可以获得全基因组相互作用的图谱【2】。
然而,如何构建基因组的高分辨率三维结构一直以来都是一个难题。目前,一般的做法是根据Hi-C实验获取的染色体相互作用图谱利用优化或者统计的方法重构出染色体的三维结构,从而为染色体折叠机制和功能的研究提供宝贵的线索。
近日,清华大学交叉信息研究院曾坚阳课题组在Nature Communications 杂志在线发表题为Integrating Hi-C and FISH data for modeling of the 3Dorganization of chromosomes的研究论文,提出了基于FISH和Hi-C实验数据重构三维基因组结构的新方法。

曾坚阳课题组早在2014年便开始着手研究三维基因组结构建模问题,并先后开发出了基于贝叶斯推断和流形学习的三维基因组结构重构方法【3,4】,其中基于流形学习开发的建模方法GEM【4】被《基因组蛋白质组与生物信息学报》(Genomics,Proteomics & Bioinformatics,简称GPB)评为2018年国内生物信息学十大进展之一【5】。此次更是在基于流形学习重构方法的基础上首次提出了将FISH数据和Hi-C数据相结合的三维基因组结构重构方法(GEM-FISH)。在GEM-FISH中,模型首先利用FISH数据和Hi-C数据来确定拓扑结构域(TADs)的相对空间位置,然后利用Hi-C数据提供的几何约束以及多聚体的生物物理性质来确定TADs内的结构,最后将上述两部分进行融合,调整优化后得到最终的三维基因组结构。
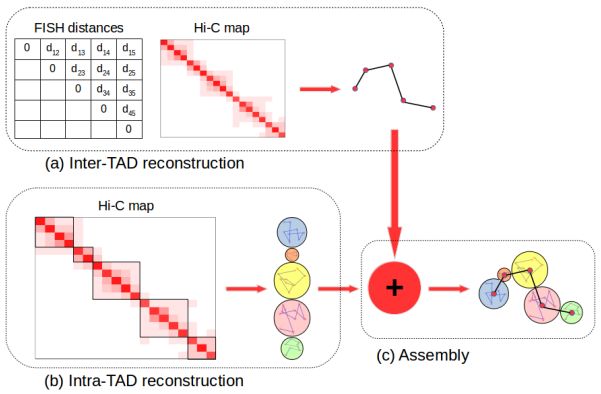
图1. 建模过程
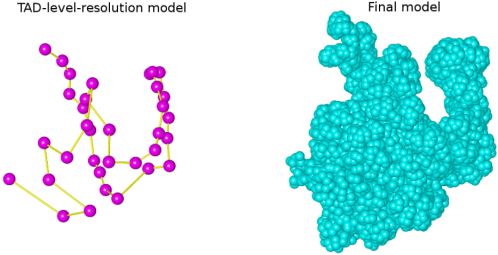
图2. TAD水平的结构和最终精细结构
在模型的评估过程中,相比之前建模方法,GEM-FISH将结构的平均相对误差降低了接近一半,获得了更为准确的三维基因组结构。另外,GEM-FISH可以准确地重构的基因组的compartments以及染色质相互作用形成的环(chromatin loop)。利用GEM-FISH还能帮助发现超级增强子(super-enhancers)新的空间分布模式。总而言之,结合FISH和Hi-C数据的GEM-FISH新方法可以更为准确地重构出三维基因组结构,并为下游分析提供帮助。
据悉,该文章的第一作者是来自埃及的博士后Ahmed Abbas,通讯作者是来自清华大学交叉信息研究院的曾坚阳副教授。合作者包括张奇伟教授和高军涛研究员等。
原文链接:
https://doi.org/10.1038/s41467-019-10005-6
制版人:小娴子
参考文献
1. Boyan Bonev and Giacomo Cavalli. Organizationand function of the 3D genome. Nature Reviews Genetics. 17, 661–678 (2016)
2. Lieberman-Aiden E, van Berkum NL, Williams L,Imakaev M, Ragoczy T, et al. Comprehensive mapping of long-range interactionsreveals folding principles of the human genome. Science 326: 289–293(2009).
3. Siyu Wang, Jinbo Xu and Jianyang Zeng.Inferential modeling of 3D chromatin structure. Nucleic Acids Research.2015.
4. Guangxiang Zhu, Wenxuan Deng, Hailin Hu, RuiMa, Sai Zhang, Jinglin Yang, Jian Peng, Tommy Kaplan, Jianyang Zeng.Reconstructing spatial organizations of chromosomes through manifold learning. Nucleic Acids Research. 2018.
5. https://mp.weixin.qq.com/s/sPI6X-iP2IY1a8iR4Vy6WA
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652469474&idx=4&sn=5651de0cc190d7358c40f7a75cf69e88&chksm=84e2eb56b39562406d058437c30b3532f98a3d894010c38d4141eb54e7dea72586e60ed8ab4f&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

宏基因组理论教程5宏基因组功能组成

中国研究团队破解中国种茶树全基因组密码
拟南芥基因组

Nature Plants封面文章:苏铁基因组——现存最原始种子植物基因组详解
北京基因组所开发基因组变异-表型关联知识库

乳腺癌有望实现精准治疗,科学家揭开基因组秘密

日本科研人员发现无呼吸微生物
北京基因组所等发现人类合子基因组激活起始于父源基因组
遗传学:解密古埃及木乃伊基因组

数字基因组