科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-05-22
来源:iNature
原标题:Nature子刊 | 同济大学刘琦团队开发基于人工智能模型的单细胞CRISPR筛选数据分析新方法
同济大学生物信息系刘琦教授课题组近日成功开发了基于人工智能主题模型(Topic Modeling)的单细胞CRISPR筛选数据的分析方法。相关研究论文《Model-based Understanding of Single-cell CRISPR screening》于5月20日在《自然·通讯》(Nature Communication)杂志在线发表【1】。
CRISPR筛选(CRISPRscreening)是揭示基因功能以及进行基因筛选的一项有效的高通量实验技术。近来,面向单细胞的CRISPR筛选技术(被称为Perturb-Seq, CRISP-seq和CROP-seq等)通过结合传统CRISPR筛选技术和单细胞测序技术的各自优点,使得研究者能够在单细胞水平上实施大规模的基因扰动,从而在更加精细准确的尺度下对基因扰动进行检测和评估,适合针对于高异质性的细胞类型(如肿瘤细胞,免疫细胞等)进行大规模的基因功能研究和靶点筛选。伴随这一技术的产生, 开发面向该数据类型的数据建模方法对于阐明复杂疾病的发生发展机制、有效进行药物靶点筛选等具有重要的研究意义。然而,该技术数据分析具有若干挑战:1)单细胞测序技术和CRISPR筛选技术各自存在巨大的噪音,二者结合所产生的单细胞CRISPR筛选技术使得数据本身的噪音加剧,迫切需要开发对噪音鲁棒的数据分析方法;2)面向单细胞CRSIPR筛选,如何设计有效的算法定量衡量sgRNA的敲除(knockout)/敲降(knockdown)的效率以及评估其潜在的脱靶(Off-target)效应?3)在单细胞层面,如何定量分析和评估扰动基因对细胞表型的影响以及两两扰动之间的相互关系?4)如何将扰动基因对细胞的影响进行直观有效的可视化展示?
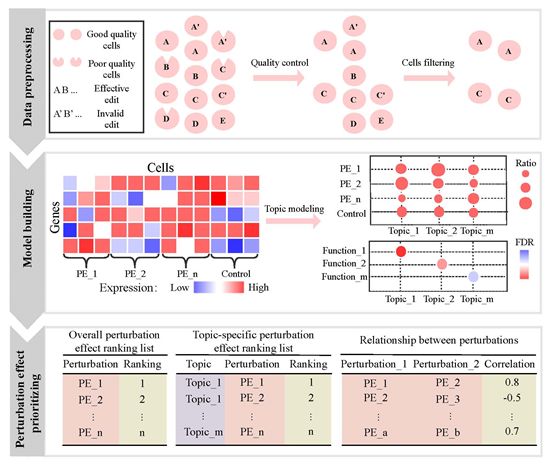
图1:基于主题模型的单细胞CRISPR筛选数据的分析流程图
针对上述单细胞CRISPR筛选数据分析中存在的问题,刘琦教授课题组提出了一种基于人工智能主题模型(Topic Modeling)的计算框架MUSIC(Model-based Understanding of SIngle cell CRISPR screening), 用以有效地对单细胞CRISPR筛选数据进行分析。MUSIC将单细胞CRISPR筛选数据分析类比于自然语言处理中的大规模文本处理,通过主题模型对数据本身所蕴含的隐变量(Latent variable)分布情况进行推断(Inference),用以进一步揭示该类数据本身所体现出来的生物学意义。其数据建模过程包含三个步骤:1)数据预处理。除考虑单细胞测序过程中普遍存在的噪音以及相应的预处理方法之外,MUSIC根据单细胞CRISPR筛选数据的特性设计了若干有效的数据预处理步骤,例如考虑sgRNA的敲除/敲入效率,最小干扰细胞数量等来进行噪音细胞样本的有效过滤;2)模型建立。运用主题模型对单细胞CRISPR筛选数据进行建模,获得扰动基因对细胞表型影响的主题分布,该主题分布可以用于对基因扰动所产生的细胞表型影响进行定量刻画; 3)基于主题模型定量和精准的刻画基因扰动后对细胞表型所产生的影响,并基于该影响对扰动基因进行功能排序,方便大规模的基因靶点筛选及基因功能注释等。MUSIC计算平台基于R语言开发,提供Docker版本方便用户进行快速有效的安装、部署以及使用,并在大规模的实验数据集上进行了系统的比较和测试。值得一提的是,近日(4月8日)发表在《自然·方法》(Nature Methods)上的一项工作开发cisTopic方法进行单细胞ATAC-seq数据分析,其核心算法同样基于主题模型,进一步体现了利用人工智能的隐变量模型对单细胞测序数据进行分析的优势【2】。
刘琦教授课题组近年来一直致力基于人工智能机器学习方法进行CRISPR基因编辑系统的优化设计研究。2018年,刘琦教授课题组基于深度学习方法,开发了CRISPR基因编辑系统的优化设计软件平台DeepCRISPR。相应的成果发表在国际著名期刊《基因组生物学》(Genome Biology)。该工作获国际权威论文评价体系F1000推荐为领域内Technique Advance【3】。本次工作是刘琦教授课题组继2018年DeepCRISPR工作之后在基因编辑系统数据分析领域的又一有益探索。该论文第一作者是刘琦教授课题组的博士生段斌,第一通讯作者是刘琦教授,上海交通大学第九人民医院孙树洋教授团队和同济大学医学院王平教授团队对本工作中单细胞CRISPR筛选实验层面的理解和数据解读提供了重要的支持。该研究成果得到国家科技部精准医学重点研发计划,慢病项目重点研发计划及国家自然科学基金面上项目经费的资助。
参考信息:
https://www.nature.com/articles/s41467-019-10216-x
参考文献:
【1】Bin Duan et al, Qi Liu#, Model based Understanding of Single-cell CRISPR Screening, Nature Communications, Advance Access, 2019.
【2】Carmen Bravo González-Blas et al, cisTopic: cis-regulatory topic modelingon single-cell ATAC-seq data, , Nature Methods, Advance Access,2019.
【3】 Guohui Chuai et al, Qi Liu#, DeepCRISPR: optimized CRISPR guide RNAdesign by deep learning, Genome Biology, Advance Access,2018.
来源:Plant_ihuman iNature
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3MTE3MjUyOA==&mid=2247501324&idx=6&sn=159173838efc94ac0ac25e04139d19c7&chksm=fce6b5d3cb913cc56834b33a9ee4d91face7234a9d7e16f96f761c168acb8ef10fd560c9ff02&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
构建云生态,深化云应用 第九届中国云计算大会闭幕
第九届中国云计算大会·科猫邀你免费入场
石油化工行业大数据分析应用平台

国家绿色数据中心试点工作座谈会顺利召开
P53大数据分析:超10000例癌症分析带来新见解

电信大数据分析支撑服务疫情防控阻击战

研招大数据分析:考研热背后的冷思考

收藏 | 如何运用大数据分析进行科研选题?
2015年新疆云计算大数据技术应用研讨会召开


2016制造业知识服务高峰论坛暨中国制造业知识服务联盟成立大会在北京举行


