科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-06-21
来源:ScienceAAAS
中国科学院青年创新促进会 李家堂
(中科院成都生物研究所)
评述论文:Large-scale ruminant genome sequencing provides insights into their evolution and distinct traits (Science 21 June 2019: Vol 365, Issue 6446)
反刍动物是一类包括六个科(鼷鹿科(Tragulidae),叉角羚科(Antilocapridae, Giraffidae), 麝科(Moschidae),鹿科(Cervidae)和牛科(Bovidae)),至少200个物种的重要食草动物1。牛科动物(至少143种)又是反刍动物里物种最丰富的一类2-3,不少重要的家畜(水牛,奶牛,牦牛,山羊,绵羊)就属于牛科动物4-5。在解剖学上,反刍动物因为与生俱来有多腔胃和头骨附件(如“角”)而与其他哺乳动物区别开来。反刍动物瘤胃以及除鼷鹿科动物外重瓣胃的获得,使反刍动物相较于其他食草哺乳动物(如马科动物),有对食物更高的利用率。这种利用率的提高被认为与这类动物在进化上的成功(分布广,种类多,数量大)有关系。在漫长的进化历程中,反刍动物演化出了多样化的行为和形态特征,不同物种的体型(体重<2 kg ~ >1200 kg),形态,生理具有明显差异。尽管反刍动物具有这些生物学优势且对人类文明发展有巨大价值,更多关于反刍动物的研究依然是迫切的,因为反刍动物内部,大到一些科之间的系统关系仍然存在争议,而且,反刍动物反刍行为及其他特征演化背后的遗传机制也是鲜为人知的。
在本期Science上,西北工业大学暨中国科学院昆明动物研究所王文研究员团队联合中国科学院昆明动物研究所,华大基因,哥本哈根大学等多家科研机构阐述了他们在反刍动物基因组进化研究领域的重要成果。研究团队通过对44个反刍动物基因组的从头测序组装,注释及与12个已公布的哺乳动物基因组作为外群比对分析鉴定到了221,166反刍动物特异性保守非外显子元件(RSCNEs)。继而,团队研究人员利用全基因组比对获取的373Mbp基因组直系同源共线序列,以虎鲸为外群构建了51个反刍动物的基因组系统发育树(nDNAtree)(图1A)。基于不同数据(中性位点(4D sites),CNEs, 完整线粒体(mtDNA))构建的系统发育树则进一步验证了基因组系统发育树拓扑结构的可靠性。尽管不同的基因组区域内检测到了不完全谱系分化(ILS)信号,基于收敛的系统发育分析方法(ASTRAL,MP-EST)说明了ILS对nDNAtree的拓扑结构是没有影响的,这些最终说明了这是一棵质量可靠的系统发育树。nDNAtree树表明叉角羚科与长颈鹿科(Giraffidae)为姐妹枝关系,这与既往基于线粒体的系统发育树支持叉角羚科是所有不同有角类哺乳动物外群的结论不同(图1A)。这一结果为之前叉角羚科与长颈鹿科为姐妹枝的提议提供了强有力的证据(100%支持率)。nDNAtree树确认麝科是牛科的一个姐妹枝而不是鹿科的一个姐妹枝;苇羚亚科(Reduncinae)是狷羚亚科(Alcelaphinae),羊亚科(Caprinae),短角羚亚科(Hippotraginae)祖先的姐妹枝;岛羚(Neotragusmoschatus)与黑斑羚(Aepycerosmelampus)的确是姐妹枝关系(图1A)。经过估算,反刍动物于39.1~32.3百万年前开始分化,而有角类反刍动物则分化于23.3~20.8 百万年前。研究人员比较反刍动物祖先基因组的进化速率后发现,反刍动物祖先进化速率(~1.5 × 10-9),明显高于其它哺乳动物(t-检验, P <0.01)而进化速率与体型大小呈现显著的负相关关系。种群历史动态分析(PSMC)结果表明, 相同食性和栖息地的反刍动物,它们种群大小变化规律却有明显区别,这可能预示不同反刍动物对环境中生物和非生物的变化有着不同的应对机制。研究还发现,晚更新世(Pleistocene)哺乳动物物种大衰退可能比之前的研究更为剧烈,因为40种反刍动物中就有25种在这一时期(10万~5万年前)出现有效种群数量减少。然而,与之不同的是,人类群体数量却是在增加的,这种物种减少在一定程度上可能与人类的活动有关(图 2A)。
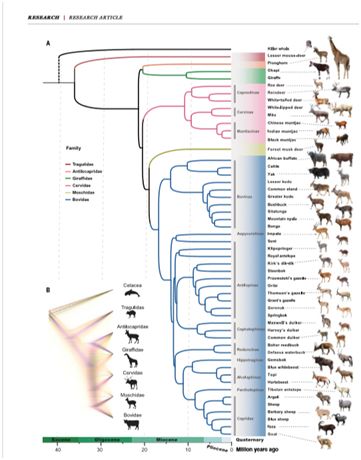
图1:(A)极大似然法反刍动物系统发育树(nDNA树);(B)WGTs中观察到的反刍动物系统发育混乱
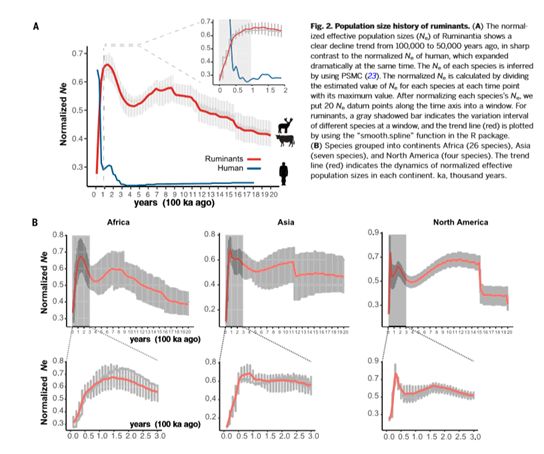
图2:(A)标准化的反刍动物和人的有效群体大小变化;(B)不同大陆反刍动物标准化有效群体大小变化
通常,基因组进化是表型进化的主要推动力,遗传的改变预示着表型的变化。本研究的科研人员还对反刍动物的基因组进化进行了详细的研究,并从基因组结构变异,编码基因及非编码调控元件进化角度,利用基因组及转录组数据建立了反刍动物表型变化与遗传差异之间的联系,为将来反刍动物基因功能研究提供了重要材料。基因组共线性分析表明,尽管反刍动物间基因组染色体数目差异明显((山羊(2n = 58);黑麂 (2n = 8/9);),其基因组相对于灵长类(Primates)和奇蹄类动物(Perissodactyla),基因组重排更少,基因组间相似性更高(图3A)。转座子(TEs)含量变化是反刍动物基因组大小变化的主要因素(图 3B,C)。反刍动物相对于其他哺乳动物基因组插入的片段里包括了妊娠相关基因簇(PAGs),干扰素基因以及嗅觉受体基因(图3D)。利用51种反刍动物以及12种非反刍动物(作为外群)的编码基因,研究人员构建了高质量的直系同源基因集,并基于该基因集,找到了反刍动物正选择基因(PSGs),快速进化基因(REGs)以及新演化的基因(图4A)。基因功能富集分析显示,PSGs,REGs以及反刍动物扩张的基因(EXGs)均在免疫相关的功能上有富集。其中,20个PSGs和12个REGs与白细胞跨血管有关(图4B),干扰素基因家族(IFNs),PAGs,组蛋白及丝氨酸蛋白酶家族基因在反刍动物中发生了显著扩张。特别地,研究人员还发现了一个瘤胃特异性表达的新基因(ENSBTAG00000038127),含有免疫蛋白的序列模块。白细胞跨血管通路中反刍动物PSGs的比其他哺乳动物多,这表明这一通路对于反刍动物来说非常重要。PSGs,REGs和EXGs也与脂肪代谢,糖代谢,氨基酸代谢以及氧化磷酸化相关 (图4C),这可能与反刍动物独特的消化系统相关(能量主要来自挥发性脂肪酸而不是糖类)。
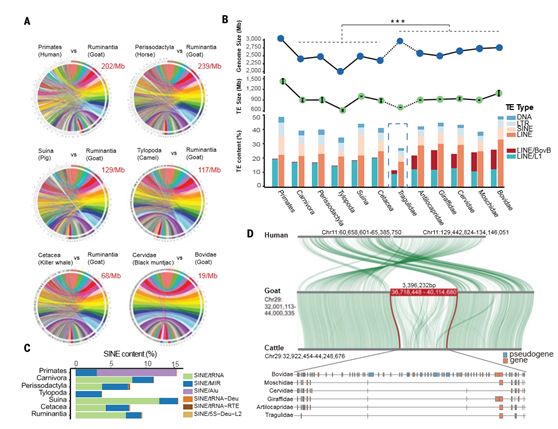
图3. 反刍动物基因组结构特征及演化。(A)以山羊基因组为参考的基因组共线性分析;(B)不同物种基因组大小及转座子种类,数量分布。(C)不同动物类群SINE基因组占比分布;(D)山羊及其它反刍动物3,396,232 bp基因组片段插入(相对于人)以及包含的编码基因和假基因(以山羊基因组为例)
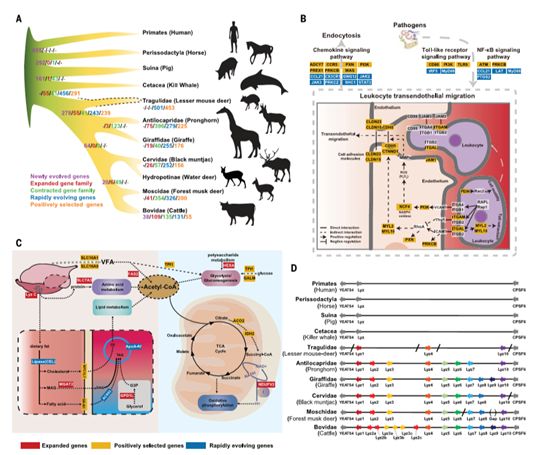
图4. 反刍动物中新演化的基因,扩张的基因家族(红色),收缩的基因家族,快速演化基因(蓝色)和受到正选择的基因(橘黄色)。(A)受到正选择的基因,快速进化基因,扩张的基因家族,新演化的基因均显示于系统发育树;(B)白细胞跨血管过程示意图及与之相关的受到正选择和快速演化的基因;(C)反刍动物营养代谢过程示意图及相关的扩张基因,正选择基因及快速演化基因;(D)反刍动物中扩张的溶菌酶c基因家族。
有角类反刍动物具有4腔胃(瘤胃,网胃,瓣胃,皱胃),那么多腔胃演化的遗传机制是什么呢?带着这个问题,研究人员使用来自绵羊50个组织的516个样品进行了转录组分析。分析结果发现瘤胃,网胃,瓣胃的基因表达谱与食管的基因表达谱相似,这表明,这些胃腔可能从食管演化而来,与之前研究一致(图5A)。另外,瘤胃中有七个新进化的基因发挥作用,其中,PRD-SPRRII 和TCHHL2是重要的瘤胃构成基因,三个丝氨酸基因可能与免疫及蛋白酶抑制相关,两个KRT6A基因与角蛋白细胞囊泡激活相关。尽管之前研究表明皱胃与非反刍哺乳动物的真胃相似,但皱胃很可能演化出了适应微生物富集环境的消化能力,反刍动物中溶菌酶c基因(与协助消化细菌及微生物细胞相关)(图4D)的扩张很好地印证了这一点。再者,溶菌酶c基因伴随着反刍动物的分化而扩张,拷贝数在牛科动物中达到顶峰,这可能说明皱胃的消化能力在反刍动物中是不断增强的。瓣胃是有角类反刍动物新演化出的消化器官,转录组分析揭示瓣胃中有75个基因特异性高表达。在这些基因中,有一个新演化的基因(LOC101107119)以及一个具有有角类动物特有氨基酸突变的基因(SCNN1D)。LOC101107119可能与钾-酮代谢相关,SCNN1D则在水分和钠离子吸收方面具有重要作用。此外,研究还发现,SIM2(转录因子), PAX9(转录因子), KCNK5, 和DENND2C基因上下游10kbp 范围内具有有角类动物特有的CNEs,且一个PAX9基因相关的CNEs还具有有角类动物特有的两个碱基突变。SIM2和PAX9均与鳞状上皮细胞分化相关,可能与瓣胃上皮与瘤胃不同有关。
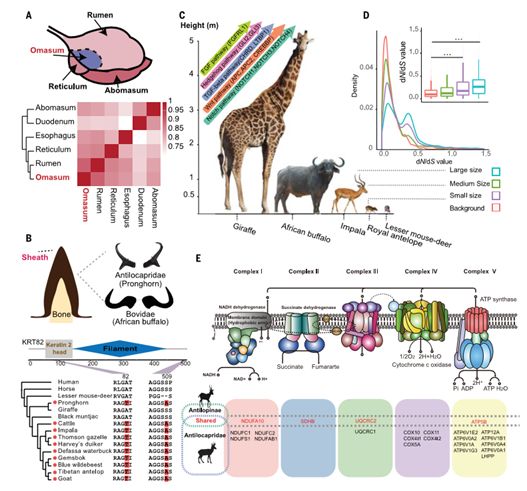
图5. 与反刍动物特殊性状相关联的遗传特征。(A)不同胃腔基因表达斯皮尔曼相关性;(B)牛科和插脚羚科角质鞘趋同特征;(C)反刍动物体型大小及与之相关基因和通路;(D)反刍动物中642体型相关基因dN/dS分布;(E)叉角羚和羚羊亚科线粒体电子转运链相关基因的演化
反刍动物“角”形态差异较大,为了阐述“角”的起源,说明鹿角快速再生能力的遗传背景,研究人员运用大规模比较转录组学方法和功能实验进行了研究(另以长文形式同期发表)。麝科和獐亚科(Hydropotinae)动物没有角,可能是RXFP2基因(角发育相关基因)趋同假基因化导致。在鹿科和牛科动物中高表达的201个基因中有36个具有有角类特有的CNEs,其中有9个基因(ALX1,VCAN,COL1A1,SATB2,RUNX2,POSTN,SP7,TNC,和 COL4A2)与骨骼发育相关。这些基因的表达不同可能导致角形态的差异的形成,这为下一步功能验证提供了研究材料。研究人员还分析了牛科和叉角鹿科角质鞘的趋同演化(图5B)。基于牛科动物山羊和绵羊的转录组数据,7个高表达的基因(KRT1,KRT2,KRT3,KRT5,KRT10,KRT14,KRT84)被找到。除了KRT10 和KRT14基因,其余基因均编码II型α角蛋白,这表明II型α角蛋白可能与牛科动物角质鞘的形成有关。分子趋同分析结果显示叉角羚(Antilocapraamericana)和牛科动物有106个蛋白质包含至少2个趋同氨基酸替换。其中,KRT82(牛角特异表达II型α角蛋白)具有两个趋同氨基酸替换,且一个替换位于角蛋白头部结构域(图5B)。这表明KRT82可能对牛科和叉角鹿科角质鞘趋同进化有重要作用。
为了调查反刍动物体型变化背后的遗传驱动力,本项目研究人员评估了642个体型大小发育相关基因在不同体型反刍动物中的非同义和同义替换率比值(dN/dS)(图5C)。这些基因在体型增加和体型减小的反刍动物中均具有显著高的dN/dS。其中6个基因(CXCL13,RNF115,NPNT,KL,SLC9A3R1和MSTN)在体型增加的反刍动物中具有显著高的dN/dS,并且MSTN(与肌肉发育相关)和SLC9A3R1(与骨骼发育相关)的dN/dS值大于1。这表明这两个基因可能是自然选择通过调节肌肉骨骼发育增加反刍动物体型的靶标基因。另一方面,在体型减小的反刍动物中,5个基因(SBDS,BMP3,LRRN3,NFATC3,和SMARCAL1)具有显著高的dN/dS值,其中,SBDS(骨骼长度正常发育有关基因的dN/dS显著>1。以上的这些基因可能为家畜的育种(体型)提供重要的依据。另外,研究人员在长颈鹿这种已知最大的反刍类动物的366个骨骼发育相关基因中,发现115个基因具有长颈鹿特有的突变,这些基因包括了变形生长因子β以及Hedgehog, Notch, Wnt和FGF信号通路中的基因。其中11个具有超过4个非同义突变的基因可能与长颈鹿拉长的身体表型相关(TGF-b(CHRD和LTBP1),Wnt(APC,APC2,和CREBBP), Notch (NOTCH1, NOTCH3,和 NOTCH4), Hedgehog (GLI2 和 GLI3) 和FGF (FGFRL1))。FGFRL1,NOTCH4和 CREBBP在之前的研究中已有报道。
善于奔跑是反刍类动物的普遍特征,为了快速奔跑,这类动物演化出了形态学独特的四肢以及潜在的生理特点。叉角羚科和羚羊亚科动物是陆地上跑的最快的哺乳动物之一。研究发现尽管在不同物种中线粒体电子转运链相关基因呈现不同演化模式,这类基因在不同物种中均有受到强烈自然选择成员。在羚羊亚科中,两个基因(SUOX和NLN)受到正选择。在叉角羚中,COX5A和PPOX受到正选择。另外有4个蛋白质(NDUFA10,SDHB,UQCRC2,ATP5B)具有趋同的氨基酸替换(Figure 5E)。这些结果暗示相同和不同的分子机制可能在反刍类动物表型趋同演化过程中扮演重要角色。
反刍动物具有特化的齿系:普遍缺少上门牙但具有高牙冠牙齿,这与其磨碎食物的需求相关,有利于拓展反刍动物食物种类增强其适应性。本研究人员发现11个与齿系相关的基因上下游10kbp内具有反刍类特有CNEs, 其中ENAM基因与特定食性的齿系形成及牙釉质厚度密切相关。另外,ENAM基因也具有反刍动物特异性两个氨基酸插入和突变。全基因组关联分析揭示FGF14基因一个内含子区域的snp与反刍动物高齿冠高度关联且该区域也是EBF1(与牙齿发育相关转录因子)的绑定位点。这些基因为进一步哺乳动物牙齿发育的实验研究提供了候选。
基于大规模基因组测序数据,该研究解决了反刍动物部分支系系统发育地位冲突的问题,并让模糊的系统关系变得清晰。尽管部分结论有待功能试验验证,但是,综合基因组和转录组数据,该研究揭示了反刍动物不同性状的演化过程及背后的遗传机制,意义重大,大量有价值的反刍动物基因组的公布也可为将来反刍动物乃至哺乳动物的演化研究提供可靠的材料。
作者简介:李家堂中国科学院成都生物研究所研究员,中国科学院青年创新促进会优秀会员,生命分会会长。主要从事两栖爬行动物系统学,进化遗传学与基因组学方面的研究工作。
参考文献:
1. IUCN, The IUCN Red List of Threatened Species, Version 2017-3. (2017); www.iucn.org.
2. D. E. Wilson, D. M. Reeder, Mammal Species of the World: A Taxonomic and Geographic Reference (JHU Press, 2005).
3. R. Heller, P. Frandsen, E. D. Lorenzen, H. R. Siegismund, Are there really twice as many bovid species as we thought? Syst. Biol. 62, 490–493 (2013). doi: 10.1093/sysbio/syt004;pmid: 23362112
4. T. F. Randolph et al., Invited review: Role of livestock in human nutrition and health for poverty reduction in developing countries. J. Anim. Sci. 85, 2788–2800 (2007). doi: 10.2527/jas.2007-0467; pmid: 17911229
5. P. H. Hemsworth, G. J. Coleman, Human-Livestock Interactions: The Stockperson and the Productivity of Intensively Farmed Animals. (CABI, 2010).
来源:Science-AAAS ScienceAAAS
原文链接:http://mp.weixin.qq.com/s?__biz=MzI3NDY3NzQ2Mg==&mid=2247487333&idx=4&sn=0ae11bf7669f1f11de0333639659005a&chksm=eb1124dcdc66adca8ff9e03462b4bf78a3e9be602b85adea92382b644901ffdf2ad80ea7d93d&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
基因组 生物进化 哺乳纲 动物进化 基因组注释 外显子测序 全基因组测序 全基因组关联分析
“垃圾DNA”对于染色体组连接至关重要

小麦D基因组完整图谱首绘成功

中国人类基因组研究有望“弯道超车”

中国研究团队破解中国种茶树全基因组密码
Prokka:快速原核基因组、宏基因组基因注释

首次对喀斯特植物实施全基因组测序

动物全物种基因组测序“顾此失彼”

日本完成一种毒蛇全基因组测序

全基因组测序重现小麦“家世”

全基因组测序:新发地病毒来自欧洲