科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-06-28
来源:BioArt
原标题:Cell | 武汉大学肖锐组与付向东组合作系统揭示新型转录因子-RNA结合蛋白参与调控转录
2019年6月27日,武汉大学肖锐研究员与美国加州大学圣地亚哥分校(UCSD)付向东教授课题组在Cell杂志上发表长文Pervasive Chromatin-RNA Binding Protein Interactions Enable RNA-based Regulation of Transcription,该研究刷新了我们对RNA结合蛋白的传统认知,揭示RNA结合蛋白是一类新的转录因子,可通过RNA依赖的染色质相互作用直接参与转录调控。肖锐研究员为共同第一和通讯作者,加州大学圣地亚哥分校陈加余博士和梁征宇博士为共同第一作者,付向东教授为共同通讯作者。

RNA结合蛋白在基因转录后的剪接加工、修饰、转运、细胞定位、稳定性、翻译和降解等RNA代谢过程中发挥着重要的调控功能,目前的研究也主要是围绕着RNA结合蛋白这些经典的功能而开展。然而,最近系统的RNA交联实验发现了大量非经典的RNA结合蛋白【1,2】,其中包括转录因子CTCF、DNA修复因子Ku80/XRCC5和转录调控复合物PRC2,暗示RNA结合蛋白可能参与更多的生物学过程。目前估计,人类基因组编码高达1500多种RNA结合蛋白【3】,另考虑到RNA结合蛋白功能的不可预知性,我们需要突破对其功能研究的局限,勇敢地去探索和发现其新的功能。
哺乳动物基因组中的转录远比我们想象的活跃。GENCODE数据显示,>80%人类基因组存在转录的证据,其中3%的区域编码蛋白质,绝大部分转录区域可产生丰富的非编码RNA,包括参与转录调控的长非编码RNA(lncRNAs)和增强子RNA(eRNAs)【4】。原则上,这些调控RNA(Regulatory RNAs)需要招募特异性的RNA结合蛋白来行使功能。的确,初步的研究发现,有些RNA结合蛋白直接参与了转录调控,包括SC35/SRSF2【5】,RBFox2【6】,NONO【7】,HNRNPL【8】和HNRNPK【9】。
基于特异性的RNA结合蛋白参与转录调控这一新兴的分子调控机制,作者提出一个基础的科学问题:RNA结合蛋白是否广泛作用于染色质水平?如果是,它们有什么功能,作用机制又是什么?为了回答这个科学问题,作者参与了ENCODE项目,大胆地运用ChIP-seq(染色质免疫沉淀测序)技术大规模地检测了63个有细胞核定位的RNA结合蛋白与染色质的相互作用,意外地发现其中约60%的RNA结合蛋白与染色质有广泛的相互作用,并且富集在基因启动子和增强子区域。更令人惊讶的是,作者虽然只检测了人类基因组中一小部分的RNA结合蛋白(<5%),却发现约40%的活性染色质区域和约70%的活性启动子区域都存在RNA结合蛋白的特意性结合,这提示RNA结合蛋白在染色质水平存在着广泛的功能和作用。随后,作者通过GRO-seq(Global Nuclear Run-On测序)实验揭示,部分RNA结合蛋白直接参与了转录调控过程。为了进一步阐明作用于染色质水平的RNA结合蛋白调控转录的具体机制,作者以RNA结合蛋白RBM25为例开展了深入研究。RNA结合蛋白和转录因子的染色质结合区域的共定位分析提示,RBM25与转录因子YY1存在紧密关联。前期研究表明,转录因子YY1的染色质结合活性,部分依赖于RNA【10】;同时,YY1也是一种重要的三维基因组结构蛋白,介导启动子和增强子之间的长程相互作用【11】。那么,RNA结合蛋白RBM25是否作为RNA和YY1之间的纽带,调控YY1的功能呢?作者通过RBM25敲除等一系列实验发现,RBM25介导了转录因子YY1的所有功能。RBM25通过调控YY1的染色质结合活性,进一步影响YY1介导的启动子和增强子的长程相互作用以激活或者抑制目标基因的转录。该研究提出了RNA结合蛋白作为转录因子或辅助因子广泛参与调控转录的新概念,也开辟了RNA结合蛋白功能研究的新领域。
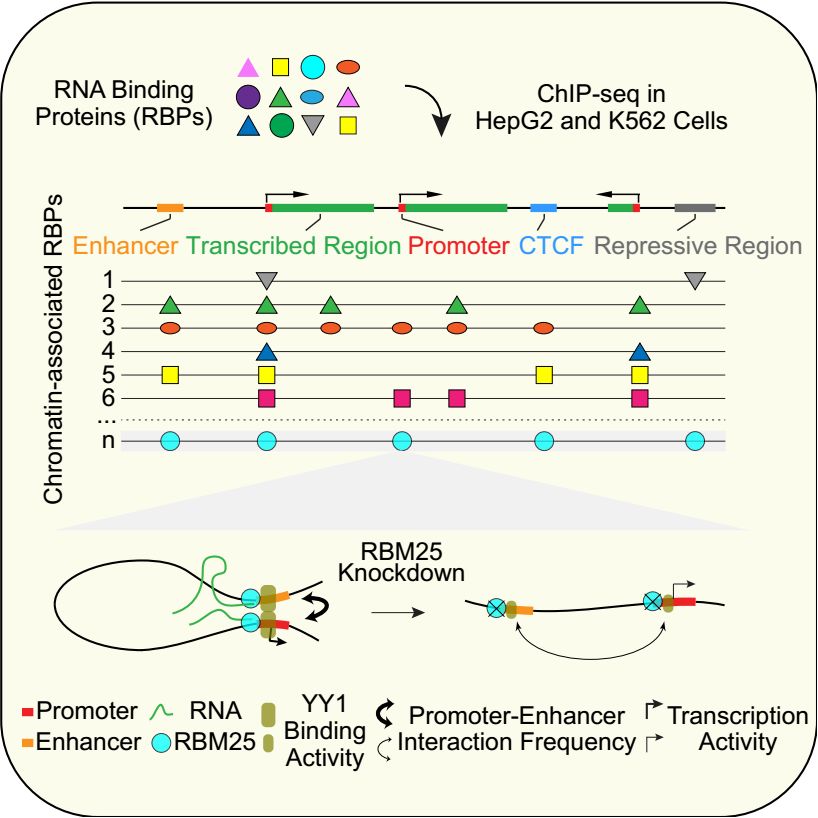
该论文的共同作者还包括武汉大学周宇教授和罗大极副教授、清华大学鲁志教授、张奇伟教授和陈阳助理研究员、厦门大学刘文教授、美国MIT Christopher Burge教授、UCSD Gene Yeo教授和康涅狄格大学Brenton Graveley教授和加拿大蒙特利尔大学Eric Lecuyer教授。
原文链接:
https://doi.org/10.1016/j.cell.2019.06.001
制版人:珂
参考文献
1. Baltz, A.G., et al., The mRNA-bound proteome and its global occupancy profile on protein-coding transcripts. Mol Cell, 2012. 46(5): p. 674-90.
2. Bao, X., et al., Capturing the interactome of newly transcribed RNA. Nat Methods, 2018. 15(3): p. 213-220.
3. Gerstberger, S., M. Hafner, and T. Tuschl, A census of human RNA-binding proteins. Nat Rev Genet, 2014. 15(12): p. 829-45.
4. Consortium, E.P., An integrated encyclopedia of DNA elements in the human genome. Nature, 2012. 489(7414): p. 57-74.
5. Ji, X., et al., SR proteins collaborate with 7SK and promoter-associated nascent RNA to release paused polymerase. Cell, 2013. 153(4): p. 855-68.
6. Wei, C., et al., RBFox2 Binds Nascent RNA to Globally Regulate Polycomb Complex 2 Targeting in Mammalian Genomes. Mol Cell, 2016. 62(6): p. 875-889.
7. Shav-Tal, Y. and D. Zipori, PSF and p54(nrb)/NonO--multi-functional nuclear proteins. FEBS Lett, 2002. 531(2): p. 109-14.
8. Kuninger, D.T., et al., Human AP-endonuclease 1 and hnRNP-L interact with a nCaRE-like repressor element in the AP-endonuclease 1 promoter. Nucleic Acids Res, 2002. 30(3): p. 823-9.
9. Michelotti, E.F., et al., Heterogeneous nuclear ribonucleoprotein K is a transcription factor. Mol Cell Biol, 1996. 16(5): p. 2350-60.
10. Sigova, A.A., et al., Transcription factor trapping by RNA in gene regulatory elements. Science, 2015. 350(6263): p. 978-81.
11. Weintraub, A.S., et al., YY1 Is a Structural Regulator of Enhancer-Promoter Loops. Cell, 2017. 171(7): p. 1573-1588 e28.
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652471839&idx=2&sn=48c54f1d7ebe9337df056a1a38392d88&chksm=84e211abb39598bdb49d55d9620570940fffd072140e6de575eedf3f5355e99c1d3a32d72cc1&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
复旦大学董爱武/沈文辉合作揭示组蛋白分子伴侣OsChz1调控染色质结构的分子机制

周政课题组在组蛋白变体的染色质组装研究中取得新进展
Cell丨抑制性染色质如何调控基因的转录?
【科技前沿】Protein & Cell :孙育杰/李程合作揭示核纤层蛋白对人源细胞中染色质高级结构及染色质运动状态的调控
Molecular Plant 出版“植物激素”专辑
关于举办“环境友好的农林害虫生态调控与生物防治技术高级研修班”通知
董爱武和沈文辉团队合作揭示组蛋白分子伴侣OsChz1调控染色质结构的分子机制

Cell : 破解异染色质细胞核周定位之谜

北京大学周岳课题组揭示组蛋白H2A的单泛素化调控转录热点染色质开关的分子机制
浙大34岁“歌神教授”:科研也是在创造艺术品


