科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-09-30
来源:BioArt
在酿酒酵母中,超过80%约6200个基因为非关键基因,这些非关键基因的存在表明基因组能够缓冲基因敲除后所带来的影响【1】。以往关于酵母方面的里程碑式工作是基因敲除品系库(Yeast Saccharomyces cerevisiae gene-knockout collection, YKOC)的完成【1,2】,该库促使了高通量的反向遗传学研究、表型筛选以及综合性分析蛋白相互作用等方面的研究。
关于单个基因功能的研究,在不同的模式生物中都取得了很大的进展。但是即使是在非常简单的模式生物中,对全基因组功能的深入理解还远远不够。关于YKOC中的一些错误或者与单个基因功能研究不一致的地方已有研究中提及【3】,但是仍然缺乏对YKOC的完整审视。
2019年9月12日,剑桥大学的Stephen P. Jackson研究组在Nature杂志上发表文章Genome architecture and stability in the Saccharomyces cerevisiae knockout collection,对YKOC几乎全部的纯合二倍体酵母品系进行全基因组测序,解析了基因敲除对全基因组的结构以及稳定性方面的影响,并进行更高维度的观测。

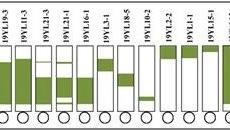
为了了解基因缺失是如何影响基因组结构与稳定性的,Jackson研究组对YKOC中几乎全部4732个纯合二倍体品系进行了全基因组测序。在确认缺失存在后,再对不同品系的基因组不稳定性进行总结。为了对基因组不稳定性进行评判,作者们将注意力放在了重复DNA序列上(图1a)。
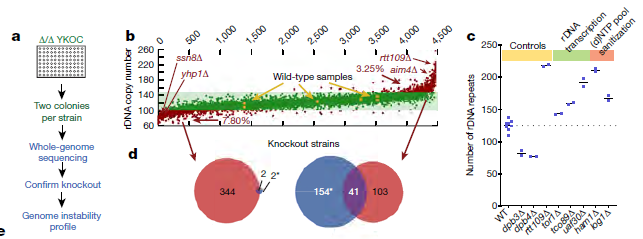
图1 YKOC品系全基因组测序及rDNA拷贝数变化
对于重复核糖体DNA(rDNA)序列的全基因组分析后发现,大约有10%的基因敲除品系中的rDNA拷贝数发生变化,并且其中很多敲除的基因也是与rDNA转录等方面相关的基因,说明测序结果是比较符合预期的(图1b, c)。通过测序作者们发现,基因组中其他元件的例如端粒、2μ质粒以及Ty转座子与rDNA相比,变异性相对来说更低一些。
测序的结果还提供了有关染色体之间以及染色体内拷贝数变异的相关信息。值得注意的一点大约52%的菌落中出现了2n倍性的偏差,并且少数的菌落中出现了稳定的染色体增加或者是损失。有一些“非完整”染色体出现的非整倍体可能是由于细胞培养过程的异质性引起的。但是高度非整倍体的菌株主要是由于包括编码参与染色体分离的基因敲除以及那些与核型维持相关的基因敲除的菌株。
为了进一步确认高度的非整倍体是否与染色体不稳定性呈相关性,作者们使用了在基因敲除的菌株中迷你染色体报告子【4】的方式进行检测。结果表明大多数这些菌株的染色体不稳定性只是略有增加,仅有一小部分显示出强烈的染色体不稳定性表型,这表明大多数非整倍体不是由基因敲除引起的严重染色体分离缺陷引起的。
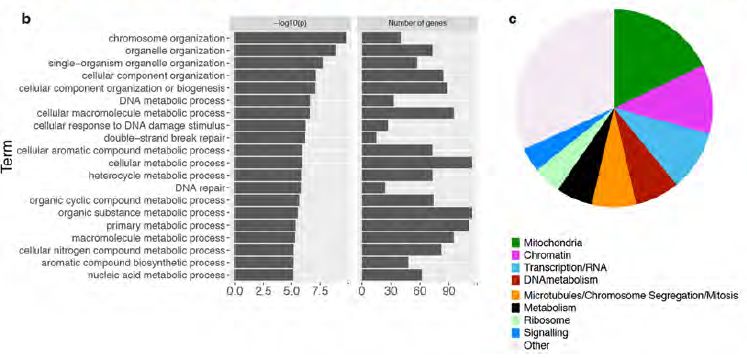
图2 测序发现对于酵母基因组稳定性极为重要的151个基因及其对应分类
Jackson研究组的工作对基因组和核型特征的系统性研究突出了基因敲除对基因组结构的影响,并且发现酵母的基因组具有很高的灵活性可以缓冲基因敲除带来的影响。
总的来说,约36%的YKOC菌株携带有重复DNA或者是染色体异常。其中,151个基因的任何一个缺失或者是多个均改变都会引起基因组结构的变化或者是基因组不稳定性产生,表明这些基因产物可以保护基因组免受不稳定性因素的影响(图2)。该工作对于与DNA拷贝数、非整倍性的变异与发育障碍、癌症以及其他疾病的研究提供了全基因组尺度的信息。
原文链接:
https://doi.org/10.1038/s41586-019-1549-9
参考文献
1. Tong, A. H. et al. Systematic genetic analysis with ordered arrays of yeast deletion mutants. Science 294, 2364-2368, doi:10.1126/science.1065810 (2001).
2. Giaever, G. & Nislow, C. The yeast deletion collection: a decade of functional genomics. Genetics 197, 451-465, doi:10.1534/genetics.114.161620 (2014).
3. van Leeuwen, J. et al. Exploring genetic suppression interactions on a global scale. Science 354, doi:10.1126/science.aag0839 (2016).
4. Zhu, J. et al. Single-Cell Based Quantitative Assay of Chromosome Transmission Fidelity. G3 5, 1043-1056, doi:10.1534/g3.115.017913 (2015).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652475874&idx=5&sn=e78862ef5770fd17dd05fe0e3f560afe&chksm=84e20256b3958b40f46ad545dc1c427d6ecba08be0496463c67f9350f5f72c0b14938818529b&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
基因组 Nature 酿酒酵母 基因敲除 基因结构 染色体结构变异

技术的新突破 | 纳米孔测序-解析染色体复杂结构变异
利用基因组图揭示癌症复杂结构变异特征
DNA的超级卷曲:其结构本身参与了基因组调节

小麦D基因组完整图谱首绘成功
小麦-簇毛麦易位染色体6VS·6AL的基因组结构和易位过程中发生的微小结构变异

乳腺癌有望实现精准治疗,科学家揭开基因组秘密

Science :首次应用原位基因组测序在单细胞水平上了解基因组空间结构
宏基因组公共数据挖掘基因组集再发Nature

新突破! 二维实验系统成功应用于四维材料研究

华中农业大学教授赵书红:在基因组上“跳舞”