科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-11-12
来源:Nextomics
近日,来自法国国家科研中心的Rainer Waldmann团队在预印本bioRxiv在线公布了题为“High throughput, error corrected Nanopore single cell transcriptome sequencing”的研究论文。该研究报道了一种将Nanopore测序技术与UMI以及10xGenomics单细胞分离系统相结合的方法,以获得经过纠错的全长序列信息。这一方法使在单细胞水平上检测差异RNA剪接和RNA编辑成为可能。

单细胞RNA测序(scRNA-Seq)是分析细胞间异质性的一项关键技术。目前基于微流控技术的高通量scRNA-Seq方法能够快速分析成千上万个细胞,但是二代测序的短读长缺少深入了解细胞间异质性的关键信息,例如可变剪接、嵌合转录本和序列变异(SNP,RNA编辑,印迹)。长度长测序技术能够较好的克服上述限制,但是PacBio的低通量导致转录组数据覆盖不足,限制了对高表达转录本的分析。相比以上两种测序方法,Nanopore测序能够产生充足的长度长数据用于转录本分析,其较高的单碱基错误率使单细胞测序中Cell barcode(cellBC)和Unique molecular identifiers(UMI)的鉴定具有挑战性。
本研究报道了一种了可以高效可靠的将cellBC和UMI分配到Nanopore reads的策略。首先利用Illumina对10xGenomics文库进行短读长测序,为每个基因和基因组区域定义相关的cellBC,然后为每个细胞、基因或基因组区域定义相关的UMIs组合。随后利用上述信息指导cellBC和UMI分配到已经比对至基因组的Nanopore reads上。
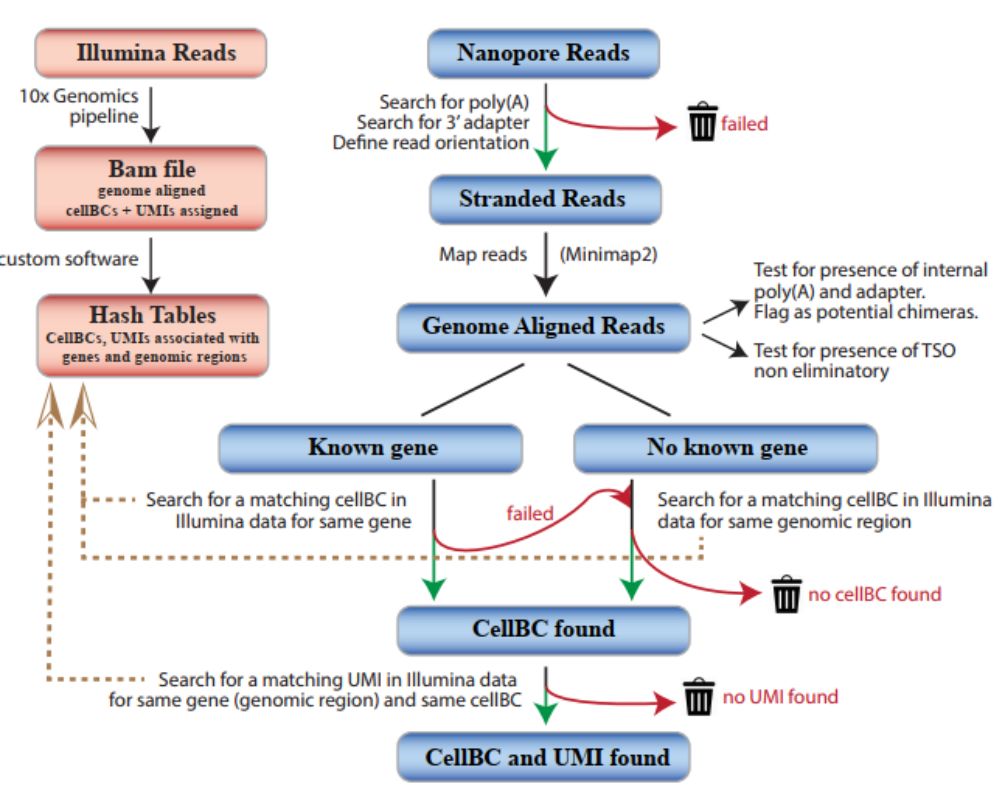
图1 CellBC 与 UMI 分配策略
研究者使用10×Genomics制备了190和951个E18小鼠大脑细胞文库,随后分别用Illumina和Nanopore测序。UMls分子条码能够使嵌合cDNA被错误地注释为新转录本的风险降至最低。但是,先前的研究未能在单细胞纳米孔测序中使用UMls。本研究的UMl分配策略中,将97.4%的UMI分配给76%的已鉴定cellBC的Nanopore reads(图2b,c)。通过二代测序测序在单细胞中鉴定到的UMIs和基因分别有75%和82%同样在Nanopore数据集中找到,并且Nanopore与二代测序之间的基因表达相关性也很好(图2e-g)。因此,该策略cellBC和UM1指定的Nanopore数据集很好地代表了10xGenomics方法中捕获的转录组。
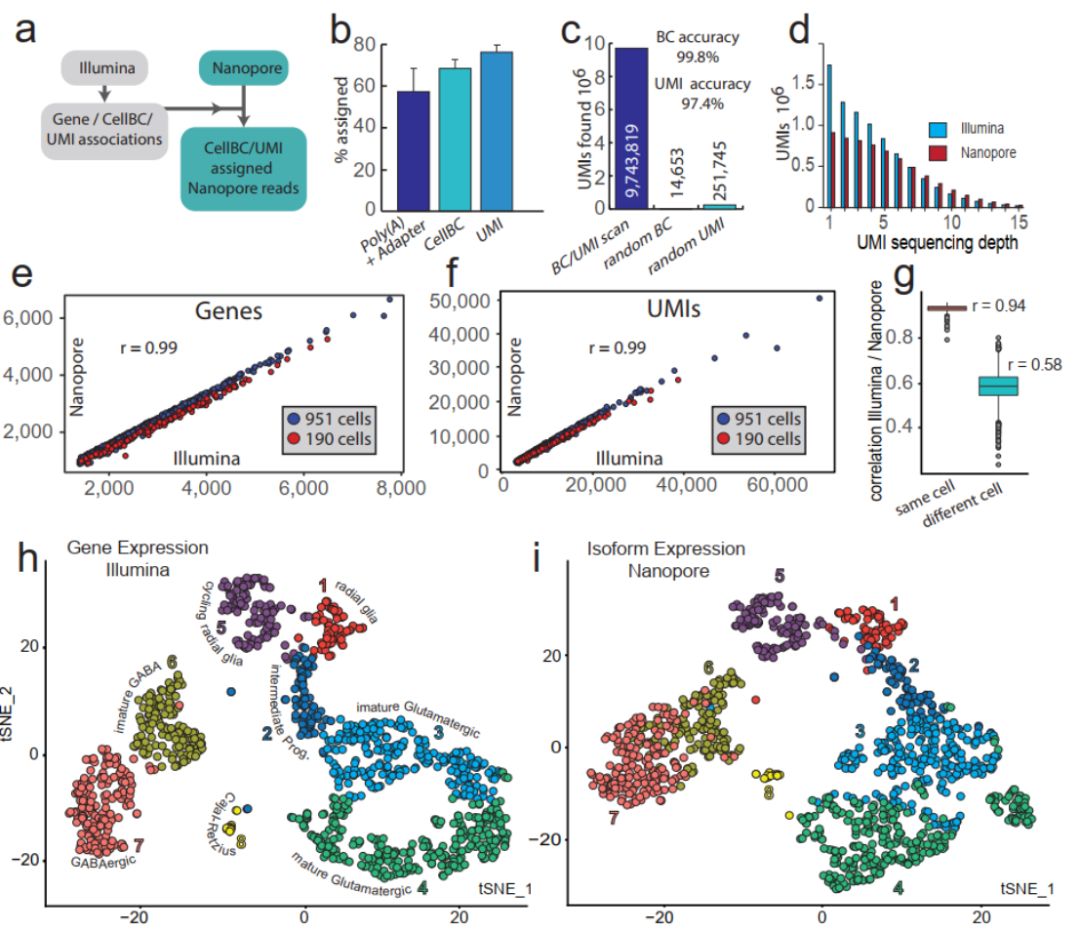
图2 Nanopore单细胞转录组测序。(a)Cell barcode与UMI 分配策略;(b,c)Cell barcode与UMI 分配效率(b)与准确度(c);(d)Illumina和Nanopore数据集的UMI测序深度; (e,f)通过Illumina和Nanopore测序,检测1141个细胞的基因数量(e)和UMIs数量(f);(g)Illumina和Nanopore测序数据之间的基因表达相关性;(h,i)Illumina基因表达(h)和Nanopore转录异构体表达(i)的t-SNE图。
Nanopore与二代测序确定的E18小鼠大脑典型的细胞类型t-SNE图显示出相似的聚类(图2 h,i)。研究者进一步寻找到139个在簇间具有差异转录异构体表达的基因,例如网格蛋白轻链A (Clta)在神经元成熟过程中经历明显的异构体转换(图3 a-d),异构体转换可能对该蛋白在不同发育阶段的作用进行微调。
研究者通过对相同UMI的reads进行分组,来纠正Nanopore测序错误,为每个RNA分子生成一致序列,并在单个细胞内识别这种序列异质性(图3 e,f)。通过对Gria2校正后的一致性序列分析,证实了之前关于Gria2编辑的发现,并且揭示了一个位点的编辑增加了另一个位点被编辑的可能性(图3 g)。因此,单细胞长读测序既证实又扩展了之前关于中枢神经系统中Gria2编辑的知识。
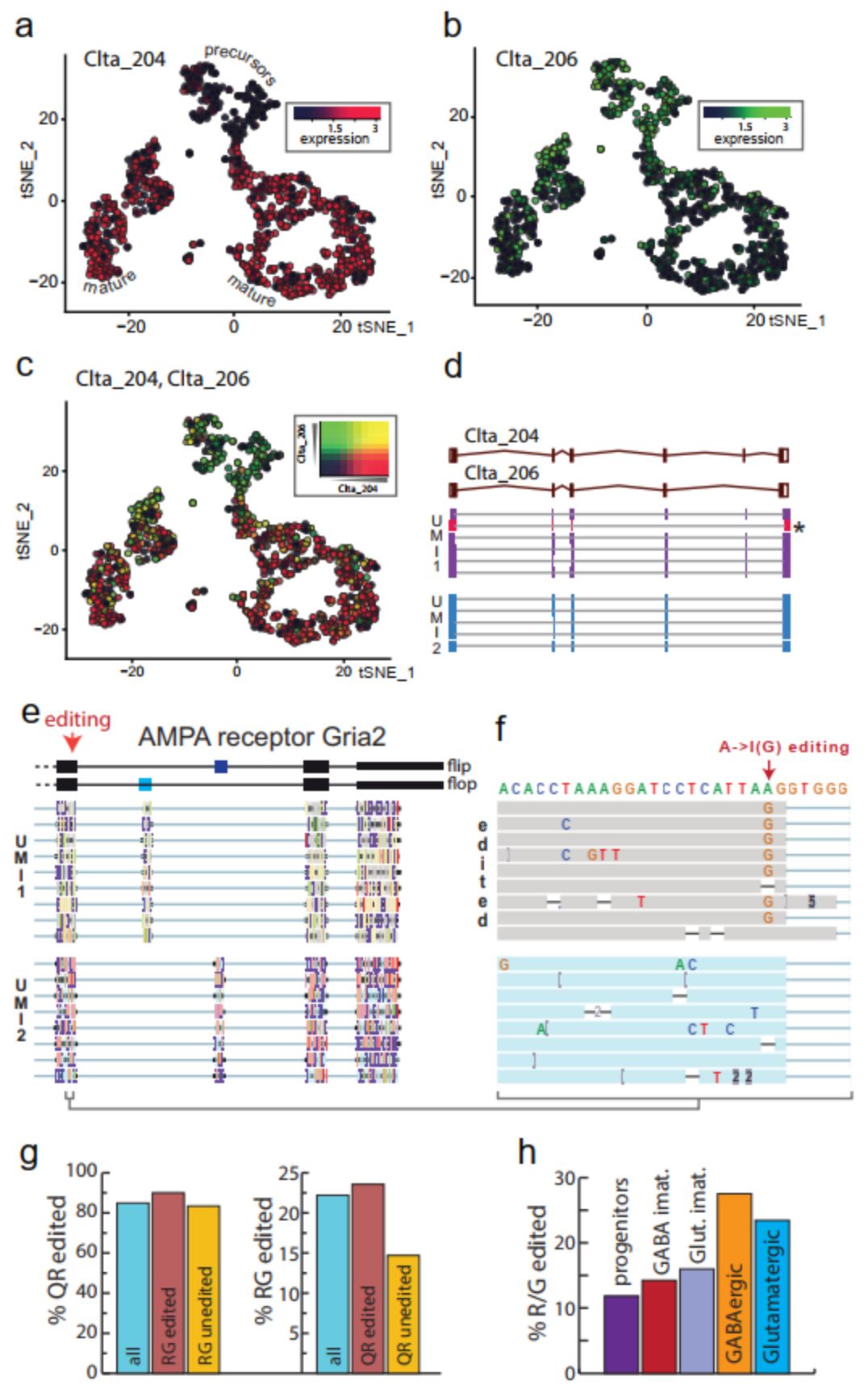
图3 Nanopore单细胞转录组测序显示转录本多样性。(a-c)t-SNE图显示了神经元成熟过程中的Clta亚型表达开关;(d)两个UMIs的主要Clta剪接变异体和支持的基因组比对read;(e)Gria2的主要剪接变体;(f)R/G A->I 编辑位点放大;(g)Q/R和R/G编辑;(h)在神经元成熟期间,R / G位点的A-> I编辑扩展会增加。
综上所述,将Nanopore测序的高通量与UMI引导的校正相结合,既可以对转录异构体进行高置信度的定义,又可以在单个细胞中识别序列的异质性。利用UMIs 进行单细胞纳米孔测序(ScNaUmi-seq)将促进RNA剪接、编辑和印迹的高通量单细胞研究,也将成为深入研究肿瘤异质性的有价值的工具。
希望组自成立以来致力于三代测序技术应用于服务,自2017年搭建Oxford Nanopore测序平台便开展基于Nanopore测序技术的研发工作,先后突破ONT Ultra-long测序、ONT微量建库、ONT全长转录组测序等前沿技术。希望组将继续深耕Nanopore测序技术,为全球客户提供快速、高效的纳米孔长读长测序服务!
参考文献:
Lebrigand K, Magnone V, Barbry P, et al. High throughput, error corrected Nanopore single cell transcriptome sequencing[J]. bioRxiv, 2019: 831495.
来源:gh_ef83defd2d1d Nextomics
原文链接:http://mp.weixin.qq.com/s?__biz=MzA5MzI3NDczNQ==&mid=2650986112&idx=1&sn=45e0c588ee6e79826795cde354eacdaa&chksm=8b962180bce1a896b9dbb6e82c23f89f80160437574313f7160499f611cb5c1689b40fb851b1&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
动物学: 侦察蚁能够引导搬运食物的同窝蚂蚁回家
神经科学:协同工作的大脑

转录组测序“指证”老年性黄斑变性更多遗传线索

血液转录组测序可提高罕见病的诊断率
青岛能源所开发出二代拉曼激活细胞弹射耦合测序技术

2018国内重磅科技新闻盘点

科学家开发出活细胞转录组测序技术

血液转录组测序可提高罕见病的诊断率
遗传学:巨型基因组研究提供了关于人类疾病和遗传多样性的新知识

单细胞转录组测序揭示拟南芥气孔和叶片的系谱轨迹