科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2020-12-28
来源:植物科学最前沿
2020年12月28日,北京市农林科学院玉米基因组编辑团队在Molecular Plant上发表了题为“Expanding Base Editing Scope to Near-PAMless with Engineered CRISPR-Cas9 Variants in Plant”的研究文章,成功开发了多个新型碱基编辑器,极大的拓展了植物基因组碱基编辑范围,尤其是C/T碱基编辑接近无任何PAM限制,PAM识别达到60种;A/G碱基编辑也达到了40种。

碱基编辑技术(Base editing)是基于CRISPR/Cas系统发展起来的新型靶基因修饰技术,在2017年被Science杂志评为年度十大科学技术突破之一。碱基编辑范围受限于核酸酶识别的PAM序列,现有植物碱基编辑技术在总共64种PAM中仅能识别约20种。因此,亟需继续拓展碱基编辑的PAM范围。
本研究利用SpCas9的三个变体SpRY,SpNRRH和SpNRTH,开发出三款新的胞嘧啶碱基编辑器(CBE),在植物中实现了除NTG PAM外几乎无任何其他PAM限制的C/T碱基替换;与此同时,本研究还利用SpRY开发出新的腺嘌呤碱基编辑器(ABE),将A/G碱基替换范围拓展到NAB(B=C,T,G), NCR(R=A,G)和NTK(K=T,G)PAM靶点。
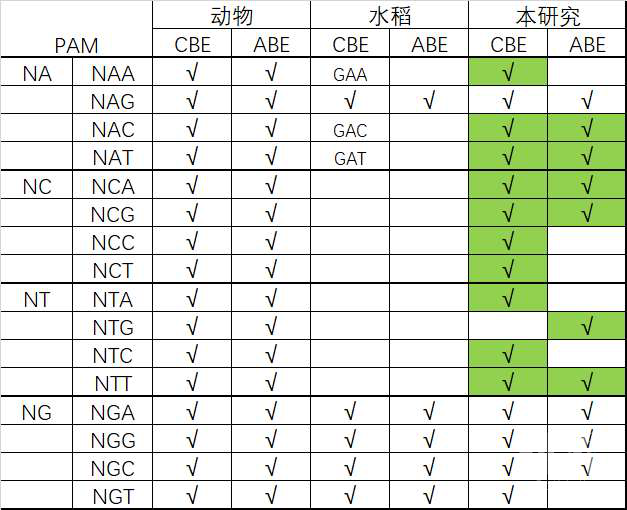
此外,对于NGC PAM靶点,SpRY碱基编辑器可以互补已有SpCas9-NG编辑器的不足。无论是SpRYn-CBE还是SpRYn-ABE对NGC PAM靶点的编辑效果都显著优于相应的SpCas9-NG碱基编辑器。最后,本研究还对编辑器SpRYn-CBE和SpRYn-ABE的技术参数进行了详细分析,发现SpRYn-CBE的编辑窗口为靶点5端1-13bp,且第2,3,4,5位的C较其他位的C更易被编辑;SpRYn-ABE的编辑窗口为靶点5端2-14bp,且第4,5,6,7,8位的A较其他位的A更易被编辑。由于无论SpRYn-CBE还是SpRYn-ABE都能够识别esgRNA骨架上与靶点相连的GTT PAM序列,检测发现大约一半发生了碱基编辑的靶点产生了一定程度的T-DNA自切割;同时也由于二者都能够识别更多的PAM,脱靶效应都被检测到,但是脱靶位点比例总体上仍然较低。
除上述新型碱基编辑器外,本团队还在近两年开发出基于SpCas9及其变体VQR和xCas9、以及SaCas9变体SaKKH的多款新型碱基编辑器,实现了对PAM 为NAG、GA和松弛型的NG、NNMRRT及NNKRGT靶点的C至T碱基替换。综合应用这些具有不同特点的碱基编辑器,可以更大范围地拓展作物基因组上基因组编辑的可靶向区域,具有很大的应用潜力,同时可以为植物功能基因研究、快速精准育种提供技术支撑。
张成伟、王瑶、王飞鹏为本文共同第一作者,杨进孝和赵久然研究员为本文共同通讯作者。本研究得到北京学者(BSP041)项目和北京市自然科学基金(6204041)资助。玉米基因组编辑团队于2017年组建成立,目前团队主要聚焦于碱基编辑、精确替换、组学编辑及其在玉米和水稻等作物中的高效应用。这是该团队自成立以来在国际学术期刊上发表的第9篇高水平研究论文。
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247506345&idx=4&sn=c03ea3b513cd67c778c32a7ecd8bdc71
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

PLOS Genetics:植物表观基因组编辑系统来了

华中农业大学教授赵书红:在基因组上“跳舞”

乳腺癌有望实现精准治疗,科学家揭开基因组秘密

日本科研人员发现无呼吸微生物

中国人类基因组研究有望“弯道超车”
“垃圾DNA”对于染色体组连接至关重要
遗传学:解密古埃及木乃伊基因组
碱基编辑工具全基因组水平特异性检测的实验流程与分析方法研究取得进展

中国研究团队破解中国种茶树全基因组密码

基因组编辑技术尚不能安全用于人类