科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-01-10
来源:植物科学最前沿
大豆是地球上主要的蛋白质和油料作物,其栽培品种主要从距今7000-9000年前的野生大豆驯化而来。历史上,大豆的种植仅限于东亚地区,随后扩展到北美,南美和印度,并成为全球种植面积最大的作物之一。野生大豆和驯化大豆都以自交繁殖为主。多年自交使得在世代间积累大量重组事件,可能导致有利突变和有害突变的快速固定。有害突变被认为是具有异交配种系的其他主要农作物(包括玉米和木薯)近交衰退和杂种优势的遗传基础,因此,在全基因组内了解大豆从野生到驯化过程中的有害突变,可以帮助更好地优化大豆育种。
大豆基因组测序完成后,通过SNPs基因分型和全基因组重测序(WGS) 对大豆遗传变异进行研究,已经对大豆基因组常见和罕见SNPs进行了大量的鉴定。然而,这些数据并没有像在其他植物中得以充分的利用。此外,与驯化后的大豆相比,野生大豆的遗传变异在很大程度上仍未被充分的鉴定和挖掘。
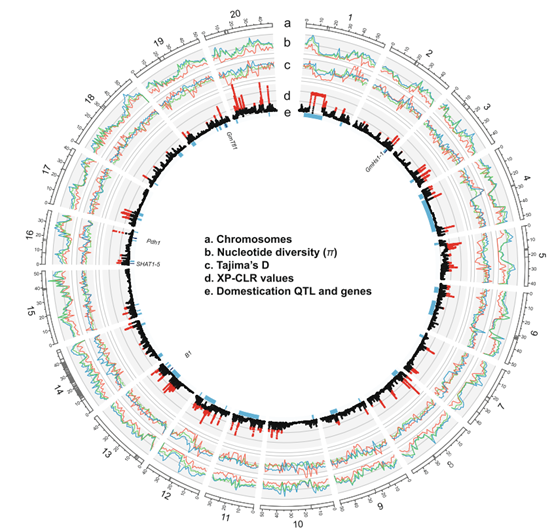
近日,Nature Communications杂志在线发表了韩国生命工学研究院Soon-Chun Jeong团队题为“The patterns of deleterious mutations during the domestication of soybean”的研究论文。作者通过对781份大豆(包括418个驯化大豆(Glycine max)、345个野生大豆(Glycine soja)和18个天然杂交大豆(G. max/G. indels))进行WGS(> 13X)测序,将鉴定到的10.6 million SNPs和1.4 million indels与表型进行关联分析。

在该项研究中,作者共鉴定到183个驯化“选择性清除”(selective sweeps)区域和在驯化和育种过程中推测的有害变异积累模式。结果表明,与野生大豆相比,驯化大豆的有害突变总数减少了7.1%,而从地方品种到改良品种的有害突变总数进一步减少了1.4%。检测到的驯化选择性清除区域也显示出在有害等位基因水平的降低。
另外,考虑到大豆基因组的更新,作者将已发表的12,116大豆种质资源(包含大量重复品种)的种子蛋白含量和油脂相关重测序数据进行再次分析,最终将鉴定到的4,467,134 个SNPs位点与8844份去重复后的大豆种质资源的种子蛋白质含量和油脂性状进行全基因组关联分析,SNPs数据与两个性状间的线性混合模型(LMM)分析结果表明,得到的GWAS结果与基于现有SoySNP50K基因型和表型数据的再分析结果非常相似。正如预期的那样,与种子油脂和蛋白质相关的主峰被鉴定到。有趣的是,在油脂和蛋白质相关性状的多变量GWAS分析中出现了10多个新的次要显著峰,如2号、4号和10号染色体上的峰。但在之前的GWAS分析中,这些区域没有一个SNP在SoySNP50K基因型数据集中表现出显著性。然而,当在同一数据集上进行多位点混合模型(MLMM)分析时,之前鉴定到的次要显著峰不再具有显著性。总之,作者提供了一份高质量的大豆种子蛋白含量和油脂相关SNPs信息,将有助于促进大豆的遗传改良。
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247507110&idx=5&sn=0b9b3f75edf1927c2fc80e5ebecdfcc4
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
山东农大科研团队为补全大豆基因组提供“拼图”
遗传学:解密古埃及木乃伊基因组

2017年度“何梁何利基金科学与技术成就奖”获得者彭士禄:用数据说话的“彭拍板”

大豆胞囊线虫染色体水平基因组序列组装成功

北京基因组所数据资源建设取得进展

华中农业大学教授赵书红:在基因组上“跳舞”

15000年前“农民”全基因组数据出炉
北京基因组所发布原核生物泛基因组数据库

小麦D基因组完整图谱首绘成功

乳腺癌有望实现精准治疗,科学家揭开基因组秘密