科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-01-11
来源:BioArt植物
古语云:龙生九子,九子各不同。自然界中有大量植物为异花授粉,包括木薯、土豆、甘薯等根茎类植物,茶树、果树等多年生林木,以及大多数药用植物。在这些植物中,每个单株的基因组都是高度杂合的,解析难度大,一对亲本杂交生成的子一代个体相互间基因型各不相同。与自交植物中两个纯合亲本杂交生成的实验群体不同,这些异交植物的实验群体在遗传上更为复杂,分子遗传学研究也相对薄弱。而且,不少异交植物还属于多倍体,子代群体中蕴含着丰富且复杂的重组交换,给基因分型和遗传定位带来很大难度。
1月8日,上海师范大学黄学辉团队与上海辰山植物园、中国林科院、中国科学院分子植物科学卓越创新中心等多家单位合作在 Molecular Plant 发表了题为Genome-wide identification of agronomically important genes in outcrossing crops using OutcrossSeq的文章。该研究针对异交植物的三类实验群体,开发了一套构建高密度基因型图谱的新方法——OutcrossSeq。该方法有望提高异交植物中基因分型、定位和遗传育种的效率。 群体构建与软件开发
群体构建与软件开发
异交实验群体有三类常见的情形:二倍体异交植物的子一代群体、同源多倍体植物的子一代群体、双亲高度杂合但有单倍型信息的实验群体。针对这三类情形,研究人员分别构建了核桃子一代群体(情形1)、甘薯子一代群体(情形2)、杂交水稻和杂交玉米双交种群体(情形3)。根据这三类情形的遗传规律,研究人员分别开发了OutcrossSeq软件包的三大模块,可基于群体的低丰度测序数据获得高质量的基因型图谱,直接用于遗传定位和育种选择。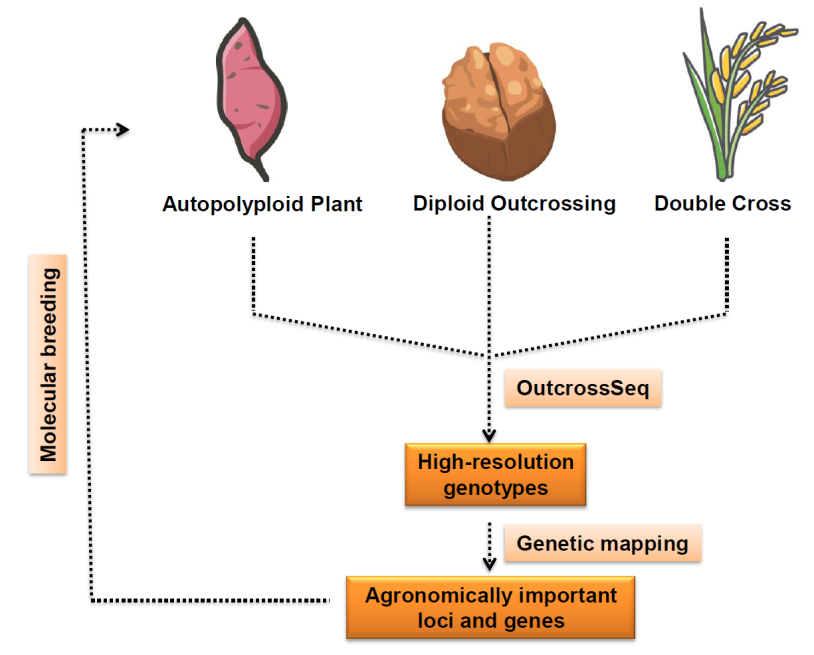
从低丰度测序数据到高质量图谱
通过对这三类群体进行低成本的全基因组低丰度测序,可以获得超高密度的分子标记。但是,低丰度测序会漏测很多区域,还有部分区域可能只偏向于测到了其中一种单倍型,同时还存在大量测序错误或比对错误引起的噪音干扰,会造成大量缺失数据和不准确基因型。研究人员利用对异交植物群体中等位变异和重组交换规律的充分了解,设计了三套算法,有效地解决了低丰度测序带来的“不确定性”问题,准确地复原了群体中每个个体全基因组范围精细的基因型(或单倍型)图谱信息。
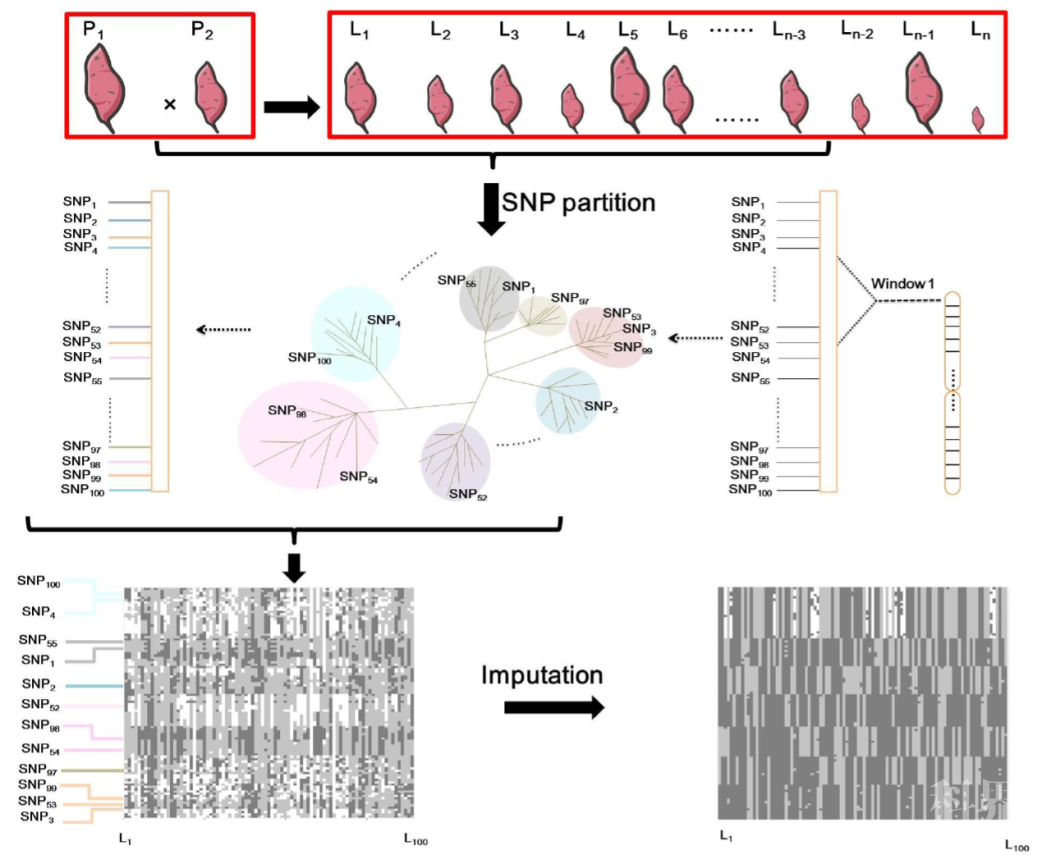
从分子图谱到基因定位
OutcrossSeq生成的高质量基因型图谱,结合实验群体的表型考察,可以直接用于高精度的遗传定位。研究人员对核桃群体的叶部性状、甘薯群体的各类农艺性状及基因表达量数据、杂交水稻和杂交玉米双交种群体的株高、花期、产量性状进行了考察和遗传定位,鉴定到了控制这些性状的QTL位点和候选基因。OutcrossSeq在不同物种中的成功应用,表明其能够为众多异交植物中复杂性状的遗传解析和分子育种以及基因组选择育种提供了一套高效、精准的解决方案。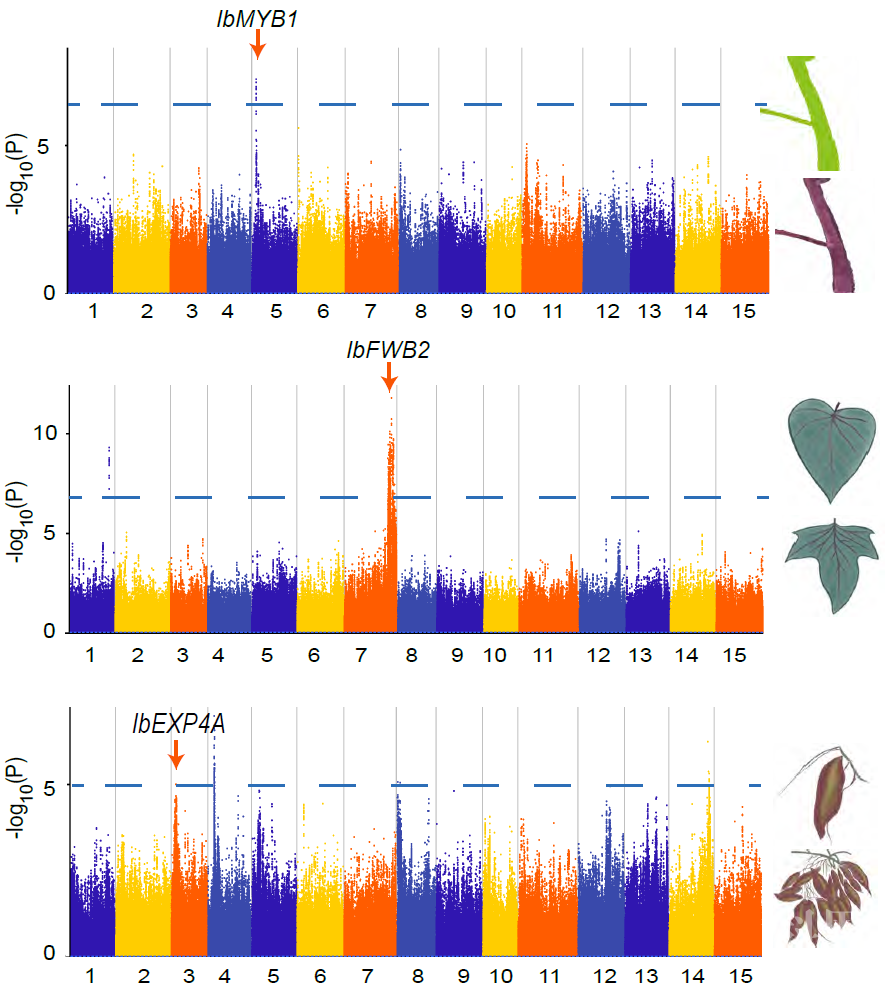
上海师范大学博士研究生陈蒙娇、上海辰山植物园范维娟博士、中国林科院博士研究生纪飞杨为论文共同第一作者,上海师范大学黄学辉教授、上海辰山植物园杨俊研究员、中国林科院裴东研究员、中国科学院分子植物科学卓越创新中心张鹏研究员为论文共同通迅作者。研究得到了国家重点研发计划、国家杰青等项目的资助。
来源:bioartplants BioArt植物
原文链接:http://mp.weixin.qq.com/s?__biz=MzU3ODY3MDM0NA==&mid=2247501889&idx=5&sn=ab64a6e10c01fc3737abe34f0129176e
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
自花授粉
人工辅助授粉技术

“授粉者”计划丨熊蜂授粉可缓解疫情期间人工短缺难题

中国作物学会甘薯专委会组织甘薯科普科技济宁行
中国作物学会甘薯专委会组织甘薯产业发展情况调研
稀有植物授粉优势揭晓

蜜蜂授粉,减本增益,应对寒潮 | 蜂巢基金“授粉者”计划之梨树蜜蜂授粉成功案例
人工授粉
果树授粉技术
自花授粉植物