科技工作者之家
科界APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-11-19
来源:BioArt
撰文 | 木兰之枻
分子条形码系统可用于细胞谱系的示踪,从而重建单细胞发育的完整历程,该系统正成为发育及疾病研究的革命性方法【1】。其基本原理是借助于CRISPR-Cas9或位点特异性重组酶,对基因组的特定位点进行持续性的编辑以制造多样化的“DNA条形码(DNA barcoding)”,从而实现对细胞的标记。近年来,单碱基编辑技术(base editing)的兴起为分子条形码系统带来新的“编码”策略,为该系统的进一步发展提供了更多可能性【2】。
目前DNA条形码的识别多借助于高通量测序,其检测精度高,可以识别条形码上单个碱基的差异【3】。不过,高通量测序的实现需要组织消化和细胞分离,这会导致细胞空间信息的丢失,而且难以涵盖所有细胞,最终导致大量有效信息的丢失。如若能实现DNA条形码的高精度原位标记,我们或能更好的从组织结构上了解细胞的动态发育历史,这对探究多种生物系统的功能意义重大。近年来有多项技术可实现DNA/RNA的原位检测,它们可以检测活细胞中表达的条形码,但条形码的表达水平受多种因素影响,这会干扰后续检测的准确度;而且这类技术的检测精度不高,往往难以区分单个碱基的差异【4】。因此,开发一种不依赖于条形码表达的高精度原位检测技术显得尤为必要。
2019年11月18日,来自加州理工学院、霍华德休斯研究所(HHMI)的Michael B. Elowitz实验室在Nature Biotechnology杂志上发表了题为In situ readout of DNA barcodes and single base edits facilitated by in vitro transcription的论文。文章成功的开发出了一种不依赖条形码表达的高精度原位检测技术Zombie,可在单碱基精度上对固定细胞中的DNA条形码实现原位成像检测。该技术在培养的细胞、鸡胚及小鼠脑组织中均可适用,对未来分子条形码系统的进步有重要的推动作用。

Zombie技术的创新点之一是不依赖于DNA条形码的表达。要实现这一目标,首先需要将T7/SP6/T3噬菌体启动子驱动的DNA条形码序列整合入基因组中(图1),该序列在活细胞中并不表达。细胞固定后,借助于噬菌体的RNA聚合酶驱动DNA条形码序列的原位转录扩增,而后便可借助于单分子FISH(smFISH)或杂交链反应(HCR)对条形码信号进行原位检测。通过构建DNA条形码单拷贝整合的小鼠干细胞系,研究者对不同聚合酶的检测效率进行了分析,发现T3和T7的效率明显高于SP6(88%,85%,75%)。研究还发现,增加条形码的拷贝数有助于降低检测的假阴性。
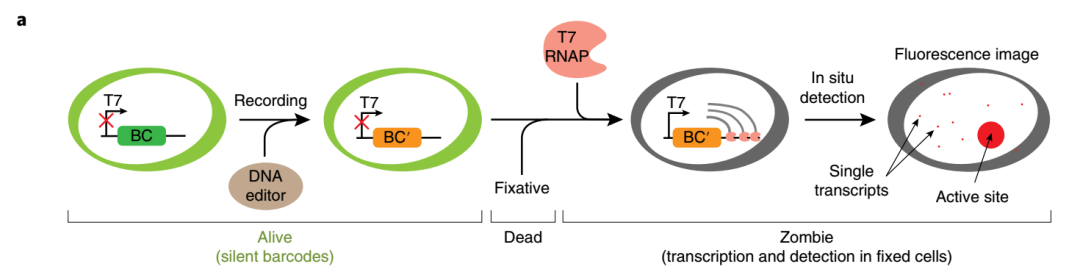
图1 Zombie技术的基本原理
Zombie技术的创新点之二是条形码的高精度检测,可有效区分20bp长度DNA条形码上单个碱基的差异。为实现高精度检测,研究者采取的策略是同时孵育浓度相同的全匹配和单碱基错配的探针,完全匹配的探针会竞争性结合条形码,而有单碱基错配的探针会被洗脱(图2)。研究证实,该策略能有效区分2-7位差异碱基为A/C/G的条形码,若差异碱基为U,错配碱基为G的探针同样能有效结合,这与早先报道的U-G之间存在摆动配对的结果相吻合。
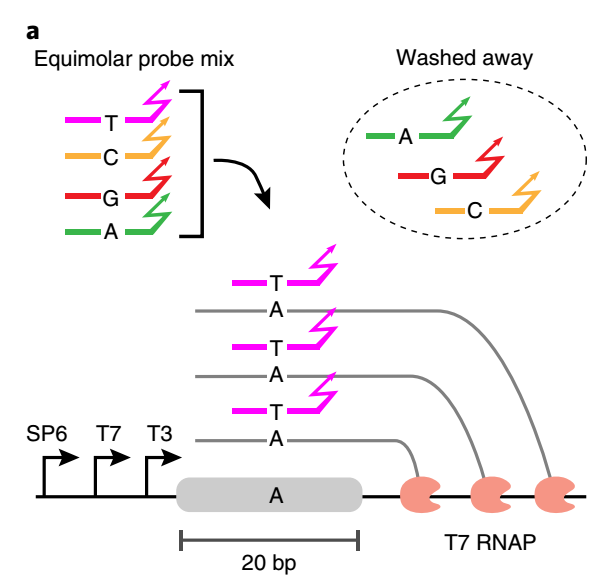
图2 条形码的高精度检测策略
之后研究者进一步证实了Zombie技术的应用潜力:借助于单碱基编辑技术,研究者可对条形码进行编辑,由此产生的多样化的条形码在追溯细胞的发育进程等研究中有广泛应用。而Zombie技术可成功的区分未编辑和编辑后的DNA条形码并进行定量分析;此外,除体外培养的细胞外,Zombie技术同样适用于鸡胚和小鼠脑组织研究;研究还证实,Zombie技术并能有效检测不同条形码的组合,且检测效果与高通量测序吻合,而且Zombie技术还能与RNA原位标记技术联用,从而实现对特定细胞内基因表达的原位检测。
总体而言,本研究开发出的DNA条形码新技术Zombie,无需DNA条形码的表达,能够实现细胞的高精度原位标记,是分子条形码领域的重大突破。传统条形码技术或依赖于高通量测序分析,但无法保留细胞的空间信息;或依赖于成像分析,但精度不足。Zombie技术兼具两者的优点,或将成为分子条形码研究带来新的思路。
原文链接
https://doi.org/10.1038/s41587-019-0299-4
参考文献
1. Farzadfard, F. & Lu, T.K. Emerging applications for DNA writers and molecular recorders. Science 361, 870-875 (2018).
2. Rees, H.A. & Liu, D.R. Base editing: precision chemistry on the genome and transcriptome of living cells. Nat Rev Genet (2018).
3. Shah, S., Lubeck, E., Zhou, W. & Cai, L. seqFISH Accurately Detects Transcripts in Single Cells and Reveals Robust Spatial Organization in the Hippocampus. Neuron 94, 752-758 e751 (2017).
4. Zheng, G.X. et al. Massively parallel digital transcriptional profiling of single cells. Nat Commun 8, 14049 (2017).
来源:BioGossip BioArt
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652477929&idx=1&sn=f4e0e7011d227a3cc84ea613c86aad2d&chksm=84e20a5db395834bf89450b286f64800a9091937430df4c42a766e92850011cdb29f1973fb4e&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
合成生物学: 一个用来控制转基因生物的内置毁灭开关

DNA“条形码”瞄准生物大发现

植物DNA条形码2.0与系统发育基因组学精品培训班在昆成功举办

DNA条形码:让中药材鉴定摆脱经验依赖

夫妻相真的存在,研究发现夫妻DNA相似度高过陌生人

一周好文 | 科技助力残障人士、DNA条形码、燕窝的背后,一周热点速递来了

DNA“条形码”有望发现200万新物种
中国蛇类DNA条形码参考数据集发布
DNA条形码揭示癌细胞逃避免疫能力

DNA到底能不能预测外貌?