科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-01-29
来源:植物科学最前沿
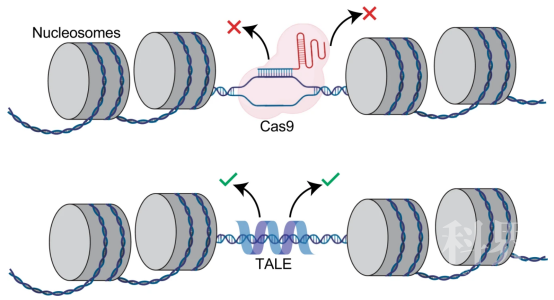
近日,美国伊利诺伊大学香槟分校赵惠民教授团队在Nature Communications 杂志上发表了一篇给题为“TALEN outperforms Cas9 in editing heterochromatin target sites”的文章。文中通过活细胞单分子荧光显微镜直接观察了dCAS9和TALE蛋白在体内常染色质和异染色质区域搜寻靶标的表现,观察发现TALEN在异染色质区域的编辑效率高于Cas9。
CRISPR-Cas9和TALEN技术作为两种强有力的基因组编辑工具,二者都可以识别自定义的DNA序列,但在靶点的结合机制上存在明显区别。Cas9蛋白在sgRNA的引导下识别具有PAM结构的DNA序列,通过DNA-RNA配对介导靶点结合。而TALEN的DNA结合域由33-34个串联的氨基酸模块构成,每个模块靶定单个核苷酸。体外单分子研究表明TALEs通过“molecular zip-line”机制沿DNA骨架进行靶标位点搜索。而CRISPR/Cas9搜索靶标位点的机制仍不清晰,可能通过3-D diffusion 或沿DNA进行1-D diffusion。
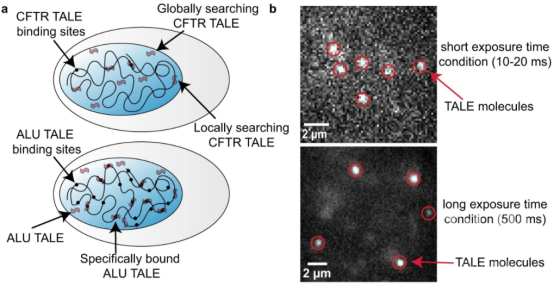
在本研究中,活细胞单分子荧光显微镜被用来观察TALE和Cas9蛋白在搜索靶标过程中的动态变化。作者设计了两个特殊的TALE蛋白:CFTR-TALE在基因组中的结合位点≤4个(该蛋白一直处于搜索靶标状态),而Alu-TALE则大概有100万个结合位点;同样地,作者还设计了CFTR和Alu的sgRNA。采用两种不同的成像条件对蛋白搜索靶点的动力学进行了分析:短曝光时间(10-20ms)研究快速扩散动力学;长曝光时间(500ms)表征结合分子的停留时间。结果表明TALE蛋白通过local search和3-D diffusion的方式搜索靶标,而dCAS9蛋白则依赖local search的方式。
在单分子层面上,对不同染色质状态下基因组编辑蛋白搜索靶标的机制进行研究,对着丝粒结构的异染色质环境下的TALE和dCas9搜索动力学进行成像,结果表明TALE和dCAS都能观察到“跳跃”(短期的“解离-再结合”事件)行为,但是TALE跳跃的频率仅取决于染色质的紧密程度,而dCas9还会额外地对PAM位点有要求。由此作者推断出一个可能的异染色质中的Cas9和TALE靶标搜索模型,即Cas9被困在结构紧密的异染色质结构中,而TALE通过“跳跃”可以有效地在异染色质中滑动。最后,通过TIDE分析对靶标位点的编辑效率进行计算,结果显示Cas9在4个常染色质位点表现出相似或更高的编辑效率,而TALEN则在异染色质中编辑效率则更高。这一结果进一步证实了TALEN更适用于异染色质编辑。
来源:frontiersin 植物科学最前沿
原文链接:http://mp.weixin.qq.com/s?__biz=MzIyOTY2NDYyNQ==&mid=2247508020&idx=4&sn=2db635c793ca23c560075ee6749e9f44
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
【大师讲堂】“单分子下拉”——以单分子分辨率解析蛋白“身份”

超高分辨率荧光显微镜成像技术再次登上《Nature》!
活细胞3D打印“有生命的纹身”,制造可穿戴传感器

新技术使荧光显微镜图像后处理效率提高数十甚至上千倍
全内反射荧光显微镜
【纳米】人工改造的巨噬细胞用于逆转免疫抑制微环境及抗肿瘤免疫治疗

组织可由活细胞创建定制

中美科学家开发出更快的荧光显微镜图像处理技术,可将后处理时间缩短高达几千倍

活细胞中氧化聚合

中国科大合作开发最轻的头戴式荧光显微镜(0.43g)