科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-02-08
来源:BioArt
真核生物的转录是一个高度复杂且被准确调控的动态过程,RNA聚合酶 II (RNA polymerase II, Pol II) 作为转录过程(特别是转录延伸阶段)的核心成员,对遗传信息的传递起着关键作用。研究表明,在转录延伸过程中,普遍存在Pol II暂停(Pol II pausing)现象,其参与下游基因的表达调控、影响基因可变剪切的发生,与细胞分化、发育息息相关【1, 2】。目前,Pol II暂停相关的转录调控机制研究主要依赖于NET-seq(Native elongating transcript sequencing)等高通量测序技术【3】,成本高、周期长。因此,如何实现在全基因组上水平上快速、高效的分析Pol II 暂停的偏好位点及其序列特征,以及其与相关转录因子间的关系,是转录调控领域亟待解决的技术难点。
近日,清华大学交叉信息研究院的曾坚阳/赵诞团队在PNAS上发表了题为A machine learning based framework for modeling transcription elongation 的研究论文,首次使用深度神经网络模型在全基因组上预测Pol II的暂停位点,并发现Pol II暂停与部分转录因子、表观遗传标记显著相关,同时为潜在的可变剪切方式提供了可解释性分析。

NET-seq是一种能够在全基因组上检测Pol II暂停位点的技术,这种技术可以将DNA单链上的Pol II暂停位点以单核苷酸精度检测出来。研究者采用基于注意力机制(attention mechanism)的卷积神经网络(Polymerase II Elongation Pausing Modeling through Attention-based deep neural Network, PEPMAN) 在NET-seq数据集上对Pol II暂停位点进行预测。在HeLa S3和HEK293T两种细胞系中进行验证,PEPMAN均取得了AUROC 0.87和0.85的高准确率,效果显著优于现有的机器学习模型。同时,研究者发现在跨细胞系的预测中,PEPMAN仍然取得了高于0.84的准确率,表明Pol II暂停的特征在HeLa S3和HEK293T两种细胞系中具有高度重合性。
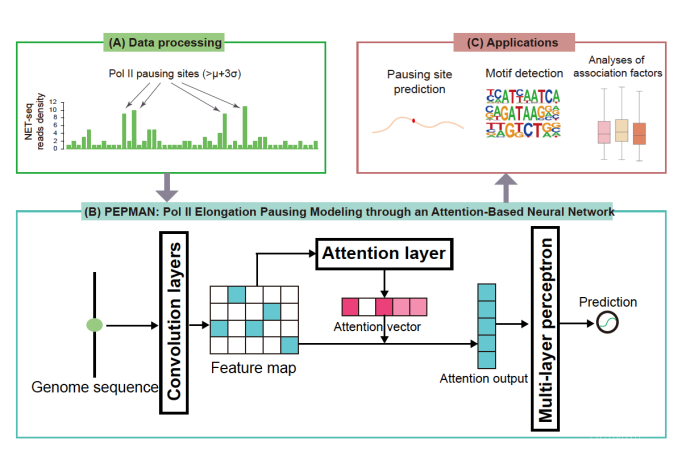
图 1 PEPMAN模型架构
随后,研究者通过引入深度学习模型的注意力机制,发现决定Pol II暂停发生的位点多集中在暂停点附近。在HeLa S3细胞系中,决定序列分别位于上游-14-0 bp处和下游7-12 bp处。而在HEK293T细胞系中,只位于-14-0 bp处。通过分析这些区域位点的序列特征,研究者发现他们主要是SMAD3,SF1,EWS:ERG等转录因子的结合区域,而这些转录因子大部分都曾被报道参与转录调控、可变剪切等生物过程。
在模型应用方面,研究者尝试用模型预测基因组上的一些特定位点。比如,研究者发现Pol II在可变剪切位点上具有更高的暂停偏好,且3’剪切点相比于5’剪切点偏好性更高。研究者进一步在五种不同的可变剪切类型(skipping exons, mutual exlusive exons, retained introns, alternative 3’ splice sites, alternative 5’ splice sites) 的剪切点上预测Pol II的暂停倾向,发现不同的可变剪切类型Pol II暂停的位置有所不同,其中一些位置特征在其他文献中已得到验证。这一方面肯定了模型的准确性,另一方面也揭示了Pol II在决定可变剪切类型上发挥的重要作用。
研究表明,多种调控因子参与Pol II暂停/释放(Pol II pause release)的动态过程【4, 5】,研究者进而探究了Pol II在转录因子结合位点、组蛋白修饰位点、以及DNA甲基化位点上的暂停倾向。结果显示在所有9种转录因子结合位点上,Pol II暂停倾向都显著升高;在10种组蛋白修饰位点上,有9种暂停倾向升高;非甲基化位点比甲基化位点具有更高的暂停倾向。这些结论与之前的一些研究结果相吻合,比如DNA的甲基化会抑制转录因子CTCF的结合,从而减弱Pol II的暂停。值得一提的是,虽然这些相似的结论可以从NET-seq数据中部分获得,但在NET-seq测序深度不足的区域却无法得到相同的结论,因此 PEPMAN很好的克服了测序深度不足造成的影响,并能够发现一些潜在的Pol II暂停参与的转录延伸新机理。
Pol II暂停与基因表达调控关系密切,因此,研究者进一步探讨了Pol II暂停倾向与基因表达量之间的关系。作者将每个基因的平均暂停倾向与基因的RNA-seq表达量作了对比,发现Pol II暂停与基因表达量呈现正相关 (Spearman correlation=0.41) 。随后对这些高度相关基因进行Gene ontology (GO) 分析后,发现这些基因多参与DNA结合和转录调控过程。
综上所述,本研究首次使用深度学习模型对Pol II的暂停现象进行了建模,全面解析了决定Pol II暂停的基因位置和序列特征,阐明了Pol II暂停与可变剪切、转录因子、组蛋白修辞、DNA甲基化以及基因表达量之间的关系,为转录调控机制的研究提供了高效、有力的预测工具。
清华大学交叉信息研究院的博士生研究生冯沛源和硕士研究生肖安为论文的共同第一作者。清华大学交叉信息研究院的曾坚阳副教授和赵诞助理研究员为论文的共同通讯作者。
曾坚阳研究组长期致力于开发先进的机器学习和人工智能技术,应用于药物发现和基因组学研究。近几年的主要研究工作包括药物-靶点互作预测(Li et al, Cell Systems 2020; Wan et al, GPB 2019),药物重定位(Luo et al, Nature Communications 2017),药物-靶点关系自动文本挖掘(Hong et al, Nature Machine Intelligence 2020),基因组学分析建模(Zhang et al, Cell Systems 2017; Li et al, Nucleic Acids Research 2017; Zhang et al, Nucleic Acids Research 2015),三维基因组结构建模(Abbas et al, Nature communications 2019; Zhu et al, Nucleic Acids Research 2018; Wang et al, Nucleic Acids Research 2015),基因转录过程建模(Feng et al, PNAS 2020)等。
专家点评
马坚(美国卡内基梅隆大学计算机科学学院教授)
RNA聚合酶II (Pol II) 在基因转录中有至关重要的作用。在基因转录时,有非常多的基因存在Pol lI暂停现象,直接影响到转录延伸速率、调控基因的表达量、以及可变剪切的结果,从而影响下游调控通路和相关非常基本的细胞功能。随着高通量测序技术的发展,现在已经能够测定基因组上Pol II暂停的位点,但是用实验去确定Pol II的暂停和延伸依然需要大量工作。所以,用计算模型去更好的预测Pol II的延伸速率并对Pol II的延伸提供量化的深层次理解有非常重要的应用价值。
近日,清华大学交叉信息研究院曾坚阳/赵诞研究组利用Pol II测序数据,通过深度学习模型首次完全基于DNA序列,对转录过程中Pol II的停顿现象进行系统性的预测。相较于传统的计算方法,该模型取得了最优的预测效果。该研究的重要性包括:1)首次发现通过DNA序列特征就能精确预测基因上任何一个位点的Pol II停顿倾向,从而建立起DNA序列特征与Pol II停顿的量化关系;2)通过对模型注意力机制(attention mechanism)的分析,发现决定Pol II停顿位点上游的序列特征在决定Pol II停顿中起主导作用并通过序列分析发现决定Pol II停顿的序列特征与部分转录因子序列高度相似,从而建立转录因子与Pol II停顿的潜在联系;3)通过模型发现针对不同的可变剪切类型,Pol II具有不同的停顿方式;4)全面分析了Pol II 停顿转录因子结合区域,组蛋白修饰区域,以及DNA甲基化的相关性; 5)分析发现,在测序深度低的区域,模型依旧能预测得到一致的结论,很好体现了深度学习模型可以弥补测序技术的不足,对Pol II的延伸速率提供更全面的刻画。
深度学习方法在基因组研究中应用近几年一直非常火热,但是通过模型去进一步理解生物机理和功能并不容易。曾坚阳/赵诞团队的这个工作基于深度学习模型有效通过DNA序列预测Pol II停顿现象,从而为在无Pol ll测序数据的细胞上研究Pol II停顿提供了坚实的辅助。或许更有意义的是,基于模型可解释性的分析为进一步研究Pol II停顿现象的提供了一个全新的分析框架,以及后续实验验证的思路去进一步发现潜在的基本机制。
原文链接:
https://www.pnas.org/content/118/6/e2007450118.short?rss=1
参考文献
1. Dujardin, G. et al. Transcriptional elongation and alternative splicing. Biochim. Biophys. Acta 1829, 134–140 (2013).
2. Li, J. & Gilmour, D. S. Promoter proximal pausing and the control of gene expression. Curr. Opin. Genet. Dev. 21, 231–235 (2011).
3. Mayer, A. et al. Native elongating transcript sequencing reveals human transcriptional activity at nucleotide resolution. Cell 161, 541–554 (2015).
4. Carrillo Oesterreich, F., Bieberstein, N. & Neugebauer, K. M. Pause locally, splice globally. Trends Cell Biol. 21, 328–335 (2011).
5. Zhou, J., Ha, K. S., La Porta, A., Landick, R. & Block, S. M. Applied force provides insight into transcriptional pausing and its modulation by transcription factor NusA. Mol. Cell 44, 635–646 (2011).
原文链接:http://mp.weixin.qq.com/s?__biz=MzA3MzQyNjY1MQ==&mid=2652524951&idx=5&sn=765c5bf8475d1bee1767e006d5a7db48
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn

这么多机器学习的应用场景,金融领域到底有何不同?

Nature: | 相分离调控转录起始至转录延伸的转换过程

新研究揭示动物断腿再生的转录调控机制

万建民院士团队系统总结水稻抽穗期的转录调控和转录后调控机制
生长素响应的转录抑制调控网络

大赛预告丨第二届Byte Cup来袭,赢得2万美元奖金,登上字节跳动面试直通车
【前沿】机器翻译新突破!“普适注意力”模型:概念简单参数少,性能大增

研究揭示噬菌体蛋白调控宿主转录的分子机制
PNAS :转录因子ENO调控番茄果实大小

Plant Journal:赤霉素对根瘤共生的转录调控机制