科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-02-24
来源:宏基因组
土壤水稳性团聚体微生物组样品制备方法
Methods for the Preparation of Microbiome in Water-stable Soil Aggregates
郑伟1, *
1资源环境学院,西北农林科技大学,杨凌示范区,陕西省; $现工作单位:资源环境学院,西北农林科技大学,杨凌示范区,陕西省
摘要:土壤团聚体是土壤结构的基本单位,其数量多少、粒径比例及稳定性与土壤物质循环、养分供应、持水性等息息相关。然而,针对土壤团聚体微生物组的研究还比较少。真菌、细菌可通过物理、化学等作用能促使土壤团聚体的形成及稳定,而土壤团聚体能为真菌、细菌等微生物提供良好的栖息环境。本方法针对土壤水稳性团聚体微生物组学研究,通过自身研究经历结合其它文献报道,详细论述土壤水稳性团聚体微生物组研究样本的制备方法,主要为湿筛法。通过改进传统的湿筛法(在水的作用下使土壤颗粒或团聚体分散并进行大小分级),用不同粒级的套筛在无菌水中筛分不同粒径的水稳性团聚体,最后提取不同粒径团聚体的DNA,用高通量测序手段测定不同粒径团聚体的微生物群落结构。无论是湿筛法还是干筛法都可能会对原有微生物组的种群分布造成一定影响,本文将详细讲述如何在水稳性团聚体样本制备过程中最大程度的避免团聚体筛分过程对微生物的影响。
关键词: 团聚体,微生物组,土壤,湿筛法
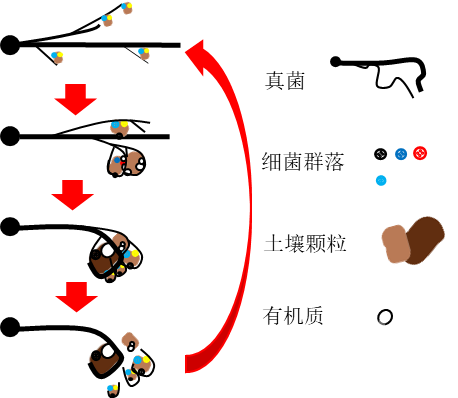
图1. 真菌、细菌与团聚体周转模式图
材料与试剂
1.0.2 M CaCl2溶液
2.50 ml 离心管
3.250 ml 离心瓶
4.无菌水 (见溶液配方)
仪器设备
1.土壤团聚体湿筛仪 (浙江托普云农科技有限公司)

图2. 土壤团聚体湿筛仪及套筛(套筛最好高温灭菌,并用酒精擦拭套筛及桶内壁)
实验步骤
该实验步骤在参考Davinic等 (2012) 的基础上进行了改进。
1.用直径15 cm环刀采集土壤原状土,将原状土轻轻掰成小块,并过8 mm筛,筛好的土样放置4 ℃冰箱过夜。
2.称取80 g过筛土样 (土壤为鲜土样,不经过风干),置于2 mm筛上,然后放入盛有无菌水的桶中,在放入土壤样品时需保证套筛处于最低点时水面高度不超过套筛最上方,浸泡5分钟,在浸泡过程中用镊子或夹子将土块轻轻掰开,这里有4个桶,先选取1个桶浸泡土样并过2 mm筛,另外3个桶用作后续筛分。
3.启动筛分仪 (频率调整为2分钟内上下50次,振幅大约为3 cm)。在筛分的过程中可用手使套筛微倾,促进细微土粒过筛。筛分结束后取出2 mm筛,并将其残留在外部的土壤用无菌水冲洗到桶中。保留在2 mm筛上的土样即为 > 2 mm水稳性团聚体。将该土样放入-80℃冰箱保存用于提取DNA。
4.将桶中的水通过0.25 mm筛(倒入第二个桶),继续上述筛分步骤,筛分结束后留在0.25 mm筛上的样品即为 0.25-2 mm水稳性团聚体。将该土样放入-80℃冰箱保存用于提取DNA。
5.继续将桶中的水通过0.053 mm筛 (倒入第三个桶),继续上述筛分步骤,筛分结束后留在0.053 mm筛上的样品即为 0.053-0.25 mm水稳性团聚体。将该土样放入-80℃冰箱保存用于提取DNA。
6.残留于桶中的组分即为尺寸 << span=""> 0.053 mm粉黏粒的悬浊液,将提前配置好的CaCl2溶液3.5 mL加入桶中悬浊液加速沉淀,并倒入到250 mL的离心瓶中离心(4 ℃,3900 x g,10分钟)。最后将上层水倒掉,得到<< span="">0.053 mm粉黏粒组分。将该土样放入-80℃冰箱保存用于提取DNA。
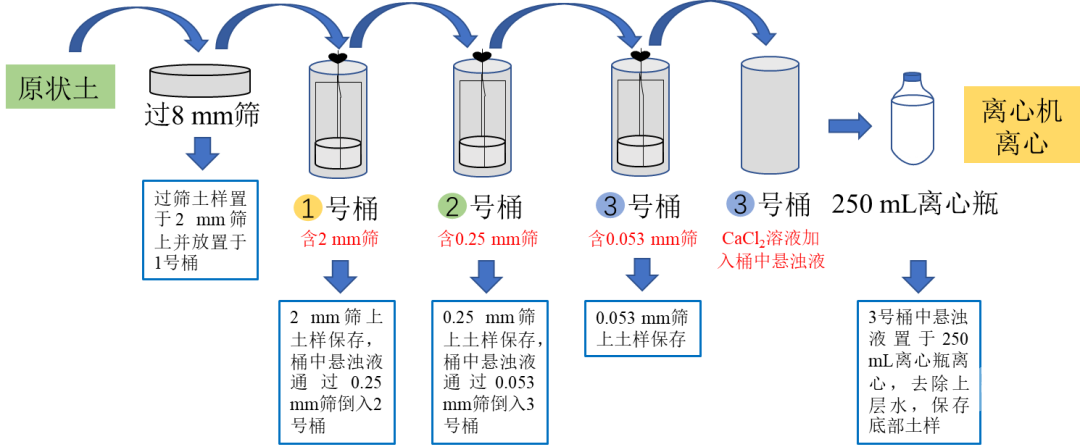
图3. 土壤水稳性团聚体微生物组土壤样品制备流程
溶液配方
1.无菌水
本实验所采用的水均为无菌水(高压灭菌锅,120℃半小时)。无菌水制备好后放入冰箱4 ℃冷藏,筛分的时候取出直接使用。
致谢
本实验得到公益性行业 (农业) 科研专项 (201303104) 及陕西省农业科技创新转化项目(NYKJ-2015-17) 的资助。该实验方法在已发表文章Zheng等(2018)中得到了应用。本实验方法在Davinic等 (2012)的基础上进行了一定的改进,在此对Davinic等人前期的优秀工作表示感谢。另外本方法仅对团聚体湿筛法的步骤做了详细描述,干筛法和湿筛法略有区别(湿筛法需要用水浸润,主要筛分水稳性团聚体),在Jiang等(2017)用干筛法筛分团聚体研究了土壤动物对不同粒径团聚体内细菌群落的影响。
参考文献
1.Davinic, M., Fultz, L. M., Acosta-Martinez, V., Calderón, F. J., Cox, S. B., Dowd, S. E., Allen, V. G., Zak, J. C., Moore-Kucera, J., (2012). Pyrosequencing and mid-infrared spectroscopy reveal distinct aggregate stratification of soil bacterial communities and organic matter composition. Soil Biolo Biochem 46: 63-72.
2.Jiang, Y., Liu, M., Zhang, J., Chen, Y., Chen, X., Chen, L., Li, H., Zhang, X., Sun, B., (2017) Nematode grazing promotes bacterial community dynamics in soil at the aggregate level. The ISME J 11: 2705–2717.
3.Zheng, W., Zhao, Z. Y., Gong, Q. L., Zhai, B. N., and Li, Z. Y., (2018). Responses of fungal–bacterial community and network to organic inputs vary among different spatial habitats in soil. Soil Biolo Biochem 125: 54-63. https://doi.org/10.1016/j.soilbio.2011.11.012
原文链接:http://mp.weixin.qq.com/s?__biz=MzUzMjA4Njc1MA==&mid=2247498238&idx=2&sn=918b1235dbb6d7f9a08705ae13ac4b21
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn