科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2021-04-07
近日,中国科学院海洋所张国范研究组完成的论文Construction of a chromosome-level genome and variation map for the Pacific oyster Crassostrea gigas(《长牡蛎染色体水平基因组和变异图谱构建》),在线发表在Molecular Ecology Resources(《分子生态资源》)上。该研究首次构建出长牡蛎染色体水平基因组和贝类中第一张包含SNP、INDEL和CNV的综合序列变异图谱,发现长牡蛎基因组中存在高比例的长片段重复序列,CNV结构变异可导致个体间平均21%的区域差异,更新了长牡蛎基因组资源和对基因组复杂性的认知。
长牡蛎(Crassostrea gigas),俗称太平洋牡蛎,是重要的养殖贝类,具有经济和生态价值。张国范研究组长期从事贝类养殖和育种相关工作,致力于牡蛎基因资源挖掘和分子育种研究。2012年,研究组在Nature发表的牡蛎第一张基因组图谱得到广泛应用,引领并开启了水产物种基因组学相关研究。之后,研究组对牡蛎基因资源进行维护和更新。随着新一代测序技术的普及和对长牡蛎染色体水平基因组需求的增加,研究组于2019年正式启动长牡蛎染色体水平基因组构建工作。
研究利用高覆盖第三代测序(PacBio,~200×)结合HIC技术,并以遗传连锁图谱辅助,对一个中国青岛的长牡蛎进行从头组装,获得基因组总长度为587M,包含10条染色体水平scaffold序列,ContigN50达到3.1M,ScaffoldN50达到60M,组装完整性和连续性均有较大提高。研究发现,基因组中高比例的长片段重复在基因进化中发挥重要作用,也是导致之前基于二代测序技术组装的长牡蛎基因组碎片化的主要原因。基于495个野生长牡蛎的重测序数据分析,构建了长牡蛎综合变异图谱,包含480万个高质量SNP,60万个INDEL和4.9万个CNV。这些新的数据资源对牡蛎比较基因组学、分子育种和适应进化等研究具有重要意义。美国南加州大学教授Dennis Hedgecock团队的成员、博士Xiaoshen Yin对该基因组开展分析,发现和连锁图谱一致性达到90%以上,明显的组装错误不到0.1%,肯定了其较高的组装质量(Yin et al, 2020)。
海洋所副研究员亓海刚为论文第一作者,研究员李莉和张国范为论文通讯作者。研究工作得到国家自然科学基金、山东省科技创新重大专项、中科院海洋大科学研究中心重点部署项目、国家贝类产业技术体系专项基金等的资助。海洋所高性能计算中心对部分生物信息分析提供了技术支持。
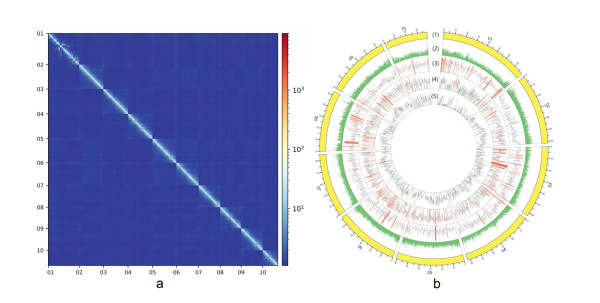
长牡蛎基因组序列图谱
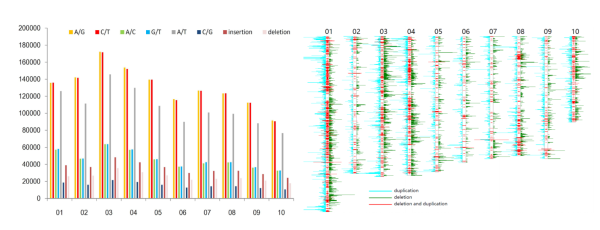
长牡蛎基因组变异图谱
来源:中国科学院
原文链接:http://www.cas.cn/syky/202104/t20210406_4783844.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
国家技术转移体系建设有了“施工图”
遗传学:解密古埃及木乃伊基因组
研究构建完成长牡蛎染色体水平基因组和变异图谱

O2O平台进入“下半场” 技术为教育赋能

小麦D基因组完整图谱首绘成功

Hortic Res 直播 | 茶树基因组解析及后基因组研究展望

NBT:超高速细菌基因组检索技术
芥菜基因组
油菜基因组
先进储能技术有哪些?