科技工作者之家
科技工作者之家APP是专注科技人才,知识分享与人才交流的服务平台。
科技工作者之家 2019-12-17
来源:中国农业科学院
12月9日,中国农业科学院深圳农业基因组研究所阮珏团队在《自然·方法学(Nature Methods)》上发表第三代测序数据组装算法wtdbg,该算法极大提高三代测序数据的分析效率,与2019年4月1日发表在《自然·生物技术(Nature Biotechnology)》上的Flye算法相比,分析速度提升了5倍,并首次将测序数据分析时间降低到少于测序数据产出时间。
基因组学技术飞速进步既源于测序技术的发展,同时也依赖于数据分析技术的提高。如今完成一个人的全基因组测序已经是件普通实验室甚至家庭都可以负担起费用的“平常”事情,以三代测序为例,完成个人全基因组测序仅需1天时间、费用低于5万元。但是完成这样数据规模的全基因组组装分析,在2014年需要消耗50万个CPU小时,只能在超大计算机集群上进行。这种情况下,同时对大量个体进行组装分析是难以想象的,然而以全基因组组装方式对群体进行测序分析已经成为生物和医学研究的趋势。
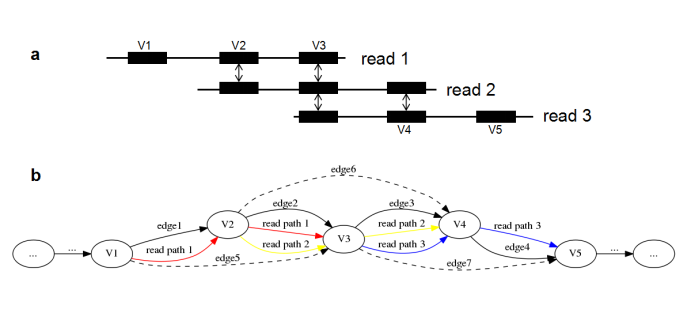
近年来,生物信息学领域的科学家们致力于改变这种数据产出速度远高于数据分析速度的尴尬状况,不断开发出更高效的组装分析算法。基因组所在成立之初就布局组学技术研究,致力于将前沿测序技术引入农业科学研究中,阮珏团队多年来始终专注于测序数据分析方法,如组装算法的开发,力求推动测序数据的分析速度更快、分析质量更高。
Wtdbg算法的开发得益于一个新的组装图理论(模糊布鲁因图)的提出。模糊布鲁因图借鉴了德布鲁因图的思想,将测序数据切分为固定长度的短串,再从短串构建出的图上恢复出全基因组序列。德布鲁因图以速度优势常用于第二代测序数据的组装分析,但因测序噪音极高的难题,从未成功应用在第三代测序数据。模糊布鲁因图重新对短串进行定义,使之能够容忍高噪音数据,并随后对生成组装图和恢复基因组序列做了大量相应的重构,使其兼具高效率和高容错的优点。
该项研究始于2013年,由基因组所阮珏研究员与美国哈佛大学医学院的李恒博士合作完成。研究成果自2016年起对所有人免费开放使用,不仅被几十篇学术论文引用,还被国内多家基因测序分析公司作为主要组装分析工具,并且在2019年世界大学生超算竞赛中作为性能测试赛题。
来源:CAAS_WX 中国农业科学院
原文链接:http://mp.weixin.qq.com/s?__biz=MzIzMDgwMDc2Ng==&mid=2247489014&idx=2&sn=85ca69222c5e4fce290f46a7c9502206&chksm=e8acb9e7dfdb30f1808c291a5172de1be1cc2f45fc1948d370a121d1ce9290c13f3b9b7c2fd4&scene=27#wechat_redirect
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
数据分析报告的三个层级

云计算浪潮下,数据分析技术将何去何从
资讯 | 高德纳咨询公司(Gartner)预测:2019年七大人工智能科技趋势
微生物所发表中国微生物组数据平台----中国科学院

收藏 | 如何运用大数据分析进行科研选题?

2019年七大AI科技趋势,百万行业将颠覆!

迎来智能数据分析的新时代

Nature评论:800名科学家联名反对统计学意义,放弃P值“决定论”
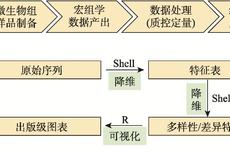
遗传:微生物组数据分析方法与应用
数据科学难在实践,有哪些弯路可以不走?