孙熙宸
加好友
孙熙宸 2019-12-20
来源:中国科学院
近日,中国科学院城市环境研究所杨军研究组的研究成果以The impacts of different high-throughput profiling approaches on the understanding of bacterial antibiotic resistance genes in a freshwater reservoir 为题发表于环境生态领域国际期刊Science of the Total Environment(2019, 693: 133585)。城市环境所研究实习员刘轩、助理研究员肖鹏为共同第一作者,研究员杨军为通讯作者。该研究得到中科院A类战略性先导科技专项、国家自然科学基金和厦门市科技计划的资助。
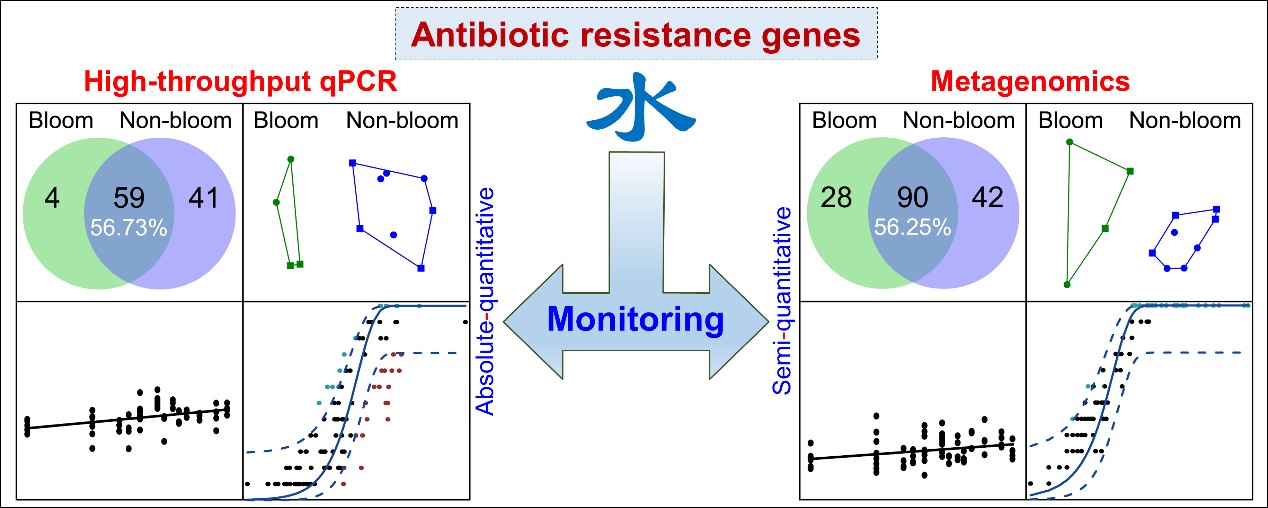
抗生素抗性基因(antibiotic resistance genes, ARGs)是一种新兴环境污染物,可通过水平基因转移等方式进入病原菌并赋予宿主对抗生素的耐药性,从而严重威胁人类的健康。环境中ARGs多样复杂,导致对其全面、系统的定量和定性检测分析难度极大。目前,高通量定量PCR和宏基因组学方法是两种最常用也是应用潜力最大的环境ARGs定性、定量检测手段。然而,在研究水环境ARGs时采用不同ARGs检测方法分析同一份环境样本很可能会得出不同的结果,目前针对ARGs检测方法的比较研究尚十分缺乏,这在一定程度上阻碍了人们对水环境ARGs的客观认知,并极大地限制了检测方法在水环境抗生素耐药性风险监测评估方面的推广应用。
城市环境所水生态健康研究组(杨军团队)以水库细菌为对象,同时采用高通量qPCR技术和宏基因组学技术进行比较研究,分析ARGs时空变化特征、ARGs与细菌类群和环境因子的关系,并对基于不同ARGs检测方法得到的结果进行比较。结果发现,相较于高通量qPCR方法,宏基因组学方法可检出环境中更多的ARGs亚类和更高的杆菌肽类ARGs丰度。基于两种不同的检测方法,发现了相同的ARGs时空动态格局、相似的ARGs与细菌和环境因子的关系,表明使用不同的ARGs检测方法(高通量qPCR和宏基因组学方法)对理解淡水环境细菌抗生素抗性基因动态规律方面上的影响较小甚至可忽略不计。高通量qPCR方法具有快速省时、可绝对定量ARGs丰度、对操作人员的生物信息学技能要求较低等优点,但其与宏基因组方法相比,存在PCR扩增偏好性和引物偏好性高、引物依赖性高等缺点。因此,该研究建议在进行日常水环境监测时可选择高通量qPCR方法,以快速获取特定高危害ARGs的定量分析数据,而在对水环境ARGs进行综合性调查时可选择宏基因组学方法以获取更全面的ARGs组成信息。
该研究结果可为研究者在环境ARGs检测分析方法的选择上提供有价值的参考,使其能更科学、更高效地监测和评估水环境抗生素耐药性风险。
文章摘要图
来源:中国科学院
原文链接:http://www.cas.cn/syky/201912/t20191219_4728243.shtml
版权声明:除非特别注明,本站所载内容来源于互联网、微信公众号等公开渠道,不代表本站观点,仅供参考、交流、公益传播之目的。转载的稿件版权归原作者或机构所有,如有侵权,请联系删除。
电话:(010)86409582
邮箱:kejie@scimall.org.cn
化学物质环境风险评估与基准/标准国际学术研讨会在北京顺利召开
城市环境所在宁波流域水环境监测研究中取得进展
水环境退化
赵京慰:环保部门有行政权,还有必要借助司法框架来主张权利吗?

生态环境部首次公布地级及以上城市水环境质量排名
中国人口日 | 生态环境如何在日益增长的人口压力中获得有效保护

【会议预告】中检协水环境专委会换届大会暨2021水环境发展论坛
水环境腐蚀
水环境保护标准
绿会副秘书长对“环境损害担责原则制度化研究”提五方面问题建议